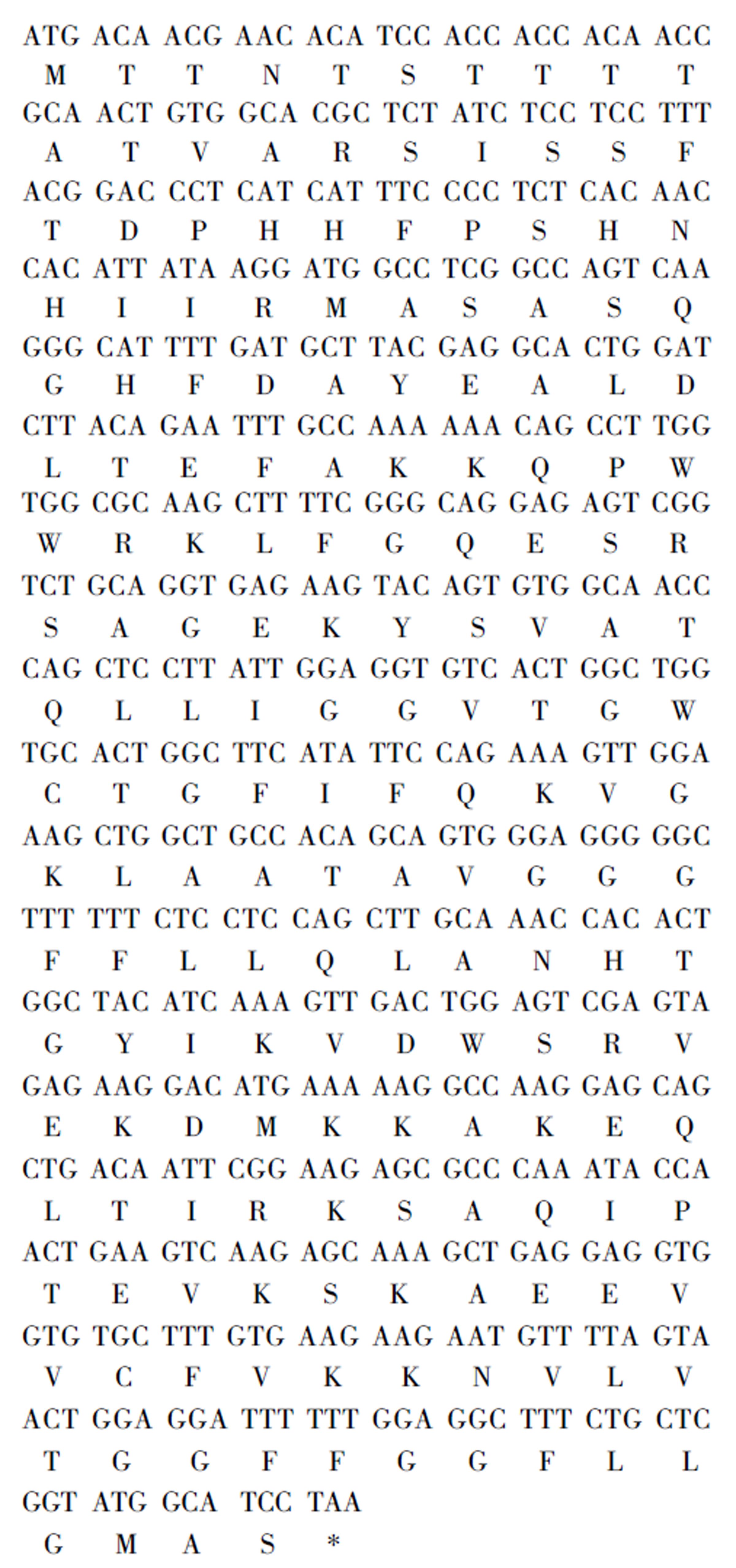修回日期: 2002-11-15
接受日期: 2002-11-28
在线出版日期: 2003-04-15
克隆丙型肝炎病毒(HCV)核心蛋白结合的肝细胞的蛋白基因, 对新发现的基因及编码产物的结构与功能, 及其基因表达的调节机制的结构基础进行生物信息学分析.
应用酵母双杂交技术, 以HCV的核心蛋白作为"诱饵(bait)", 筛选鉴定与其结合的肝细胞中蛋白的编码基因. 应用生物信息学(bioinformatics)技术, 对其中筛选得到的人HCV核心蛋白结合蛋白6(HCBP6)全长基因及其编码产物的一级结构序列, 进行生物信息学分析. 获得该基因的基因组序列, 并利用同源基因序列的比对, 确定该基因在染色体上的定位, 并确定人HCBP6基因的内含子序列. 对其编码上游的基因组DNA序列进行分析, 获得该基因启动子序列的一些信息. 同时, 根据同源基因序列的比较, 确定了小鼠HCBP6的基因序列和氨基酸残基序列. 通过对氨基酸残基序列的疏水性特点的分析, 确定了HCBP6蛋白的潜在的抗原结构位点. 通过蛋白质一级结构的在线计算机软件的分析, 对人HCBP6蛋白质一级结构潜在的功能性结构位点进行了计算机辅助分析预测.
通过酵母双杂交技术的筛选和鉴定, 结合生物信息学分析, 证实人HCBP6基因由456 nt组成, 编码产物由152 aa组成. 人HCBP6的基因组DNA是没有内含子序列的DNA结构. 通过生物信息学技术分析, 确定人HCBP6基因组DNA定位于人22号染色体上. 在对人HCBP6基因启动子序列的生物信息学分析中发现了几段可能的启动子序列结构, 以及可能的转录因子蛋白潜在的结合位点. 利用同样的技术, 确定了小鼠的HCBP6的基因序列和蛋白质一级结构序列. 对人HCBP6的蛋白质一级结构序列进行分析, 发现在其序列中第80-110氨基酸残基序列之间存在疏水性结构位点, 提示抗原位点所在, 对其进行免疫学分析提供了可能. 利用在线软件, 对人的HCBP6蛋白质结构中潜在的功能位点进行了初步的预测, 为进一步研究HCBP6蛋白的生物学功能的实验研究, 提供了丰富的信息. 这些生物信息学分析的结果, 虽然目前还只是初步的, 有些还不能被实验所证实, 但是, 对新基因的结构与功能的研究来说, 毕竟这种生物信息学分析, 能够提供一些线索, 对下一步结构与功能分析实验的设计, 具有很大的帮助.
酵母双杂交技术结合生物信息学技术, 是克隆、分析蛋白结合蛋白的基因, 对于其功能结构域的预测的有效工具, 在病毒性肝炎的致病机制研究中具有重要应用前景.
引文著录: 成军, 李克, 陆荫英, 王琳, 刘妍. 丙型肝炎病毒核心蛋白结合蛋白6基因和蛋白的生物信息学分析. 世界华人消化杂志 2003; 11(4): 378-384
Revised: November 15, 2002
Accepted: November 28, 2002
Published online: April 15, 2003
To investigate the structure and function of gene encoding human hepatitis C virus core protein-binding protein 6 (HCBP6) with yeast two-hybrid technique and bioinformatics methods.
Yeast two-hybrid technique and bioinformatics method were used to screen and identify the human HCBP6 gene and its amino acid sequences.
Human HCBP6 cDNA and genomic DNA sequences were identified. Human HCBP6 coded for 456 nt and corresponding protein was composed of 152 amino acids. The promoter sequence of HCBP6 gene was identified and promoter motif was predicted by 3 different bioinformatics methods. No intron in HCBP6 gene was identified by homologous search of cDNA and genomic DNA sequence database. The hydrophilic domain was predicted by bioinformatics methods, and the functional domains were determined with online search tools. Mouse HCBP6 gene was identified on the basis of the mouse homologous DNA sequence database. However, all data obtained from bioinformatics analysis were preliminary and further study is required for confirmation.
The bioinformatics is a powerful method for the analysis and prediction of a new gene and corresponding protein. Although the predicted result is not accurate, it provides valuable information for further study.
- Citation: Cheng J, Li K, Lu YY, Wang L, Liu Y. Bioinformatics analysis of human hepatitis C virus core protein-binding protein 6 gene and protein. Shijie Huaren Xiaohua Zazhi 2003; 11(4): 378-384
- URL: https://www.wjgnet.com/1009-3079/full/v11/i4/378.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v11.i4.378
丙型肝炎病毒(HCV)作为输血传播的非甲非乙型病毒性肝炎(NANBH)的主要病原体, 是一种单股正链RNA病毒, 属于黄病毒属, 因此HCV感染的发病机制与乙型肝炎病毒(HBV)这种DNA病毒可能有很大的区别[1-4]. HCV与肝细胞之间的相互作用, 可能是HCV感染发病机制的重要部分[5-8]. HCV基因组编码的核心(core)蛋白除了与HCV RNA结合, 保护HCV RNA免受RNA酶的消化作用, 维持HCV RNA的稳定性之外, 还具有一系列不同的生物学调节作用[9-12]. HCV核心蛋白作为一种反式激活蛋白, 对感染的肝细胞中基因表达谱产生影响, 同时, HCV核心蛋白自身结合形成同二聚体结构, 也可以与肝细胞中其他类型的蛋白之间进行结合, 形成异二聚体或多聚体结构, 对肝细胞中的信号转导产生严重干扰. 通过这些生物学作用, 对肝细胞的细胞凋亡、细胞周期进行调节, 从而参与HCV感染的发病机制[13-15]. HCV感染除了引起急性和慢性病毒性肝炎、肝纤维化、肝细胞癌(HCC), 还包括肝脏脂肪变、B细胞淋巴瘤、冷球蛋白血症等, 这些病理改变的分子生物学机制, 目前我们还知之甚少, 需要进行细致深入的探索, 以阐明HCV感染与这些病理改变之间的相互关系[16-18]. 本研究我们采用酵母双杂交技术, 以HCV核心蛋白作为"诱饵", 对肝细胞cDNA文库进行酵母双杂交筛选, 获得了一些与HCV核心蛋白结合的肝细胞中的蛋白的编码基因, 其中包括功能未知基因6号, 我们命名为HCV核心蛋白结合蛋白6(HCBP6)[12,19], 为了阐明HCBP6基因的生物学功能, 我们对HCBP6基因进行了生物信息学分析.
利用核苷酸序列数据库和基因序列同源性在线分析途径(http://www.ncbi.nlm.nih.gov/blast), 获得与酵母双杂交技术筛选结果同源性的核苷酸序列. 因为构建肝细胞表达型cDNA文库时, 插入位点的确定性, 因此, 筛选得到的序列的终止密码子可以根据与插入位点规定的ORF结构特点进行确定, 即序列中发现的第一个出现的框架内终止密码子就是该基因的终止密码子[20]. 上游翻译起始密码子的确定, 就是根据该编码基因序列的三联体密码子结构上溯, 直至没有发现进一步的框架内翻译起始密码子(ATG)为止, 来确定该基因的翻译起始密码子. 另外, 还需要查对新基因的起始密码子是否符合Kozack结构原则[21].
利用美国国立图书馆国立生物工程信息中心(NCBI)建立的核苷酸数据库(GenBank)以及同源基因序列的在线搜索分析(http://www.ncbi.nlm.nih.gov/blast)软件进行分析.
以人HCBP6的cDNA序列为参照, 应用在线分析工具(http://www.ncbi.nlm.nih.gov/blast)分析, 确定人HCBP6的基因组DNA的核苷酸序列.
首先以人HCBP6的cDNA序列为参照, 对GenBank数据库中与其同源的人基因组DNA序列进行搜寻, 确定人HCBP6基因组DNA序列中编码基因上游约3 000 nt的核苷酸序列, 并且认为人HCBP6基因启动子序列即位于此段序列之中. 然后采用NCBI的GenBank核苷酸数据库以及相关的3种基因启动子序列在线分析软件(http://www.cbs.dtu.dk/services/promoter/)、(http://www.fruitfly.org/cgi-bin/seq_tools/promoter.pl)和(http://bimas.dcrt.nih.gov/molbio/proscan/)版本1.7, 对潜在的启动子序列和可能的结合的转录因子蛋白的结构位点进行分析.
利用核苷酸序列数据库以及在线软件(http://bioinformatics.weizmann.ac.il/hyd-bin/plot_hydroph.pl)对人HCBP6蛋白质一级结构序列进行分析, 发现疏水位点, 对HCBP6蛋白质一级结构中潜在的抗原位点进行分析预测.
经过以HCV核心蛋白为"诱饵"的肝细胞cDNA文库的酵母双杂交筛选, 根据其能够突破4重缺陷型培养基的限制而生长, 且能够在X-α-半乳糖苷酶(X-α-gal)培养基上变成蓝色酵母集落, 确定了能够与HCV核心蛋白结合的一系列阳性克隆. 根据克隆的随机编号, 将这一基因编码产物命名为HCV核心蛋白结合蛋白6(HCBP6), 这一cDNA克隆的部分序列如图1所示. 根据建立肝细胞cDNA文库时外源基因插入载体的位点及其上游翻译起始点ATG的序列, 以及此开放读码框架(ORF)的翻译终止密码子(TAA), 确定HCBP6基因编码产物羧基末端的这一段多肽片段(GRVDGYIKVDWQRVEKDMKKAKEQLKIRKSNQIPTEVRS KAEEVVSFVKKNVLVTGGFFGGFLLGMAS)是与HCV核心蛋白结合的结构域.
利用NCBI建立的核苷酸数据库同源基因序列的在线分析(http://www.ncbi.nlm.nih.gov/blast), 在核苷酸序列数据库中检索, 与我们发现的HCBP6同源的核苷酸序列共有6个(表1). 但这些基因序列的功能目前不清楚. 说明我们克隆的人肝细胞基因序列是新型基因序列, 而且在国际上首次证实HCBP6与HCV核心蛋白有结合功能. 通过核苷酸序列同源性分析发现两个显著的特点: (1)人和小鼠组织中, 都存在HCBP6不同的基因类型, 这种基因类型的生物学意义非常重要, 目前还不清楚; (2)人HCBP6和利用同样技术筛选鉴定的人HCV核心蛋白结合蛋白1(HCBP1)两种基因序列的同源性在44 %以上, 提示这两种基因可能组成一个基因超家族, 也很可能还有其他超家族的成员有待于进一步的研究发现. 从目前的研究结果来看, HCBP6的基因表达类型以152 aa为主, 但其他类型蛋白的生物学意义有待于进一步的研究.
| 基因的种属与组织来源 | 核苷酸长度(nt) | 氨基酸残基长度(aa) | |
| 人 | 眼睛/视网膜母细胞瘤 | 456 | 152 |
| 树突状细胞基因序列 | 417 | 139 | |
| 22号染色体基因组DNA序列 | |||
| 小鼠 | 新生小鼠头 | 456 | 152 |
| 脑海马回 | 456 | 152 | |
| 睾丸 | 555 | 185 | |
从核苷酸序列的同源性比较分析结果确定人HCBP6的cDNA序列长度为456 bp, 编码产物HCBP6由152个氨基酸残基组成. 以此序列上溯, 上游序列中的ATG三联体密码子不再是框架内的ATG; 下游首先出现的终止密码子是TAA, 所以确定无疑. 本序列的ATG翻译起始密码子也符合Kozack原则(图2).
以人HCBP6的cDNA序列为参照, 应用GenBank数据库分析和在线分析工具(http://www.ncbi.nlm.nih.gov/blast)分析, 确定人HCBP6的cDNA与人22号染色体的基因组核苷酸序列100%同源, 所以将人的HCBP6基因定位于第22号染色体. 人HCBP6基因组序列与其cDNA序列完全同源, 未发现内含子(intron)序列.
利用在线软件(http://bioinformatics.weizmann.ac.il/hyd-bin/plot_hydroph.pl)对人HCBP6蛋白质一级结构序列进行分析, 发现疏水位点在蛋白质一级结构的第80-110氨基酸残基序列之间. 可以根据此序列人工合成抗原多肽, 进行动物免疫获得抗体或经过噬菌体表面展示技术筛选人源化单链可变区抗体(scFv), 进行免疫组织化学染色, 探讨正常肝组织中或在急性、慢性病毒性肝炎、肝硬化、肝细胞癌的肝组织中HCBP6蛋白表达的特点及变化规律.
对人HCBP6基因组DNA序列翻译起始密码子ATG上游的序列进行分析, 试图确定调节人HCBP6基因启动子序列的结构与调节基础. 对人HCBP6基因组序列翻译起始密码子上游的3000 nt的序列(图3)进行3种不同的在线软件的分析, 提示潜在的调节结构基础. 但是, 这些调节的结构基础只是生物信息学的初步分析, 还必须以实际的实验研究结果来进一步证实.
第一种分析方法: 采用在线分析软件(http://www.cbs.dtu.dk/services/promoter/), 以预测值为2.0作为其判断的cut-off值, 分析结果如下:
Promoter 2.0 Prediction Results
Sequence, 3003 nucleotides
Position Score Likelihood
600 0.596 Marginal prediction
1 000 1.055 Highly likely prediction
1 500 0.585 Marginal prediction
第二种分析方法: 采用在线分析软件(http://www.fruitfly.org/cgi-bin/seq_tools/promoter.pl)进行分析, 以预测值为0.80作为其判断的cut-off值, 分析结果如下:
Promoter predictions for 1 eukaryotic sequence with score cutoff 0.80 (transcription start shown in larger font):
Promoter predictions for seq 0:
Start End Score Promoter Sequence
625 675 0.88 ACAGTTGTATCTAAAAGTCACTATTAACTGTAGGGAAAACAAAGTTTTGA
830 880 0.96 GCTGTGCTTATATAGTCACGCTAGATCCCACCCTGATGGCAGCACCTATC
1 326 1 376 0.92 GCTTATTGTGTAAATAAGGGTTTGTGTTCGTGGCTATGGATTAATATGGC
2 896 2 946 0.97 ACATATTACATAAATAAGAAAGGGTGCTCACACCGCCCGCTGAGCTCTGT
第三种分析方法: 采用在线分析软件(http://bimas.dcrt.nih.gov/molbio/proscan/)版本1.7进行分析, 以预测值为53.00作为其判断的cut-off值, 分析结果如下:
Proscan: Version 1.7
Processed Sequence: 3003 Base Pairs
Promoter region predicted on reverse strand in 2664 to 2414
Promoter Score: 54.84 (Promoter Cutoff = 53.000 000)
TATA found at 2437, Est.TSS = 2405
Significant Signals:
Name Strand Location Weight
Y - 2 553 9.680 000
CTF - 2 551 1.704 000
MBF-I + 2 438 1.036 000
TFIID - 2 435 1.971 000
Promoter region predicted on reverse strand in 259 to 9
Promoter Score: 53.04 (Promoter Cutoff = 53.000 000)
TATA found at 27, Est.TSS = -5
Significant Signals:
Name Strand Location Weight
TFIID - 259 1.971 000
TFIID - 259 2.920 000
TFIID - 259 2.618 000
retroviral_TATA - 150 1.067 000
TFIID - 23 1.971 000
TFIID - 23 2.618 000
根据核苷酸序列同源序列的搜索分析, 确定了小鼠的HCBP6基因以及蛋白质一级结构的序列, 如图4所示. 小鼠的HCBP6核苷酸序列由555 nt组成, 其氨基酸残基序列由184 aa组成.
利用在线软件对人HCBP6蛋白质一级结构中潜在的功能性结构位点进行预测, 如图5所示. 关于这些结构位点预测的意义以及可靠性, 还需要实验研究结果的进一步证实.
研究新基因的结构与功能是目前分子生物学研究领域中最为具有挑战性的工作之一[22,23]. 特别是人类基因组计划(HGP)完成以后, 进入到后基因组计划(Post-HGP), 新基因的功能研究任务更为紧迫. 随着各种生物种类基因序列的测定, 已经积累了大量的核苷酸序列, 建立了基因序列强大的数据库, 同时, 关于基因序列, 特别是编码的蛋白质一级结构序列和功能结构位点保守序列的数据库的建立, 结合计算机和计算机网络技术的不断进步, 使我们可以通过对基因序列和蛋白质序列的比较分析, 进行结构与功能相互关系的预测, 为进一步的分子生物学研究提供方向和理论根据[24-26]. 但是, 应该清醒地认识到, 目前对基因序列和蛋白质一级结构序列的功能位点的认识还是初步的, 已经积累的核苷酸和蛋白质一级结构的数据库还是非常初步的, 关于这些数据的计算机分析工具也还是初步的, 因此, 关于这些数据的生物信息学分析结果, 还只能是具有一定的参考意义. 这些基于核苷酸和蛋白质结构特点、数据库的建立以及计算机分析为基础的所谓的生物信息学分析技术才刚刚开始. 这些生物信息学预测的结果有些具有一定的指导意义, 有些还不能完全照搬, 必须有实验结果的证实[27].
研究未知功能基因或新基因, 可以从不同的角度入手. 我们在肝炎病毒致病的分子生物学机制研究中, 经常会遇到研究蛋白和蛋白间相互结合、作用, 病毒基因表达产物对肝细胞表达基因谱的影响[28-30], 以及肝炎病毒蛋白的抗体[31-44]、抗原(配体)、结合蛋白、模拟表位[45-47]、DNA/RNA-结合蛋白的筛选等方面的问题, 其中现代的分子生物学技术如酵母双杂交技术(yeast two-hybrid)[48]、抑制性消减杂交技术(SSH)[49]、噬菌体表面展示(phage display)技术[50]等的应用, 都是研究蛋白质的功能与基因的克隆化相耦联的技术途径. 因为这些研究技术, 不是以研究对象的背景资料作为先决条件, 所以, 筛选的结果, 既能得到已知功能基因, 又能得到未知功能的基因. 对所得到的未知功能的基因序列的研究, 往往觉得无从下手. 生物信息学技术的分析可能会提供很好、很多有价值的信息. 我们利用酵母双杂交技术, 首先筛选到了HCV核心蛋白结合的蛋白, 在众多可以与HCV核心蛋白结合的蛋白质类型中, 包括已知蛋白基因, 例如, 载脂蛋白A1(APO-A1)、载脂蛋白A2(APO-A2)、染色体转位蛋白(translin)等[5], 从这些研究结果, 我们开始重视了HCV感染与肝脏脂肪变的问题, 并进行了系统的研究, 为HCV感染引起肝脏脂肪变提供了许多证据, 发现了一些慢性丙型肝炎肝脏脂肪变的有规律的现象[51,52]. 同时, 在酵母双杂交筛选过程中也得到了一些其他的信息, 例如获得了一些编码基因序列, 但这些蛋白的生物学功能目前还没有研究报道, 还不特别清楚. 因此, 对于这些未知功能基因的研究迫切需要新的思路. 当初, 获得部分基因序列以后, 如果要对全长基因进行研究, 需要应用互补末端快速扩增(RACE)技术, 扩增其余部分的基因序列. 而现在只需要以此序列为参照, 利用GenBank的在线同源基因搜索, 就可以获得这部分序列, 快速有效, 十分简便. 如果我们得到的是人或小鼠等种属的基因类型, 利用同样的技术, 就可以得到其他种属的同源性的基因. 如在本研究中, 我们应用人HCBP6的cDNA序列作为参照, 进行GenBank数据库搜寻时, 就可以获得小鼠、大鼠、牛等种属生物的HCBP6的基因序列. 但是, 由于目前还没有得到全部的人类染色体的编码基因序列, 对于大部分功能位点结构更是所知甚少, 因此由此建立的蛋白质功能位点的预测软件还有许多不完善的地方. 因此, 目前应用计算机分析技术对新型蛋白质分子中功能结构进行预测, 都还是初步的, 其结果也仅供参考. 但是, 对蛋白质结构的疏水性位点的分析结果, 还是相对可靠的. 我们已经利用这些分析结果获得了人HCBP6蛋白的抗原位点, 而且通过噬菌体表面展示技术对半合成的抗体基因文库的筛选获得了相应的人源化单链可变区抗体[33-43]. 生物信息学技术不仅可以用于结构基因的功能预测, 而且还可以用于调节基因的预测. 例如, 我们应用不同的生物信息学技术对人HCBP6基因编码区上游的核苷酸序列进行分析, 初步获得了HCBP6基因启动子的序列结构特点. 关于HCBP6基因启动子的功能分析, 目前我们的报告基因表达载体的构建以及细胞转染实验结果也证实了这一点.
总之, 目前以克隆新基因为目的的分子生物学技术, 其实验结果的分析, 必须利用、结合生物信息学技术的研究成果, 根据生物信息学技术预测的初步结果, 进行实验设计, 以获得事半功倍的效果; 当然, 目前的生物信息学技术, 由于核酸和蛋白一级结构与功能之间的相互关系的研究资料还不是特别完整, 因此, 这种预测的结果也还是初步的. 某些方面的研究结果比较可靠, 例如对同源核苷酸序列的比对以及全基因的克隆化, 蛋白质一级结构的分析中关于疏水位点的确定等, 都还是具有一定的可信度的, 但是关于蛋白质功能结构位点的预测, 关于启动子序列中转录因子蛋白结合的种类以及结合位点的预测, 则与实际情况还有相当大的差距, 还需要进行不断的更新与改进. 现代分子生物学技术与生物信息学技术的结合, 相互补充和改进, 将是当前基因的分子生物学研究、重大疾病发病的分子生物学机制研究的主流方向, 相信随着分子生物学技术本身的发展以及数据库和计算机分析技术的发展, 这些技术的综合运用, 将迎来分子医学发展的崭新阶段.
| 4. | Cheng J. Molecular pathogenesis of viral hepatitis. J Gastroenterol Hepatol. 2002;16:A185. |
| 8. | 成 军, 朱 传琳. 肝炎病毒对双链RNA激酶PKR的调节作用. 国外医学微生物学分册. 2000;23:1-3. |
| 9. | 刘 妍, 成 军, 王 刚, 李 克, 段 惠娟, 王 琳, 董 菁, 洪 源, 张 跃新, 李 莉. 应用抑制性消减杂交技术克隆丙型肝炎病毒核心蛋白反式激活基因. 解放军医学杂志. 2001;26:880-883. |
| 11. | 成 军. 丙型肝炎病毒基因组的翻译及其产物的加工. 国外医学微生物学分册. 1995;18:14-16. |
| 12. | 李 克, 王 琳, 成 军, 张 玲霞, 段 惠娟, 陆 荫英, 杨 继珍, 刘 妍, 洪 源, 夏 小兵. 酵母双杂交技术筛选克隆HCV核心蛋白结合蛋白基因1. 世界华人消化杂志. 2001;9:1379-1383. |
| 13. | 刘 妍, 成 军. 丙肝病毒致肝细胞癌的分子生物学机制. 国外医学流行病学传染病学分册. 2000;27:10-13. |
| 15. | 成 军, 陈 菊梅. 丙型肝炎病毒5'-非翻译区及其结合蛋白的研究进展. 国外医学微生物学分册. 2001;24:7-9. |
| 17. | 李 克, 王 琳, 成 军, 陆 荫英, 张 玲霞, 牟 劲松, 洪 源, 刘 妍, 段 惠娟, 王 刚. 丙型肝炎病毒核心蛋白与染色体转位蛋白的相互作用. 中华医学杂志. 2002;82:673-677. |
| 18. | Li K, Wang L, Cheng J, Lu YY, Zhang LX, Mu JS, Hong Y, Liu Y, Duan HJ, Wang G. Interaction between hepatitis C virus core protein and translin protein- a possible molecular mechanism for hepatocellular carcinoma and lymphoma caused by hepatitis C virus. World J Gastroenterol. 2003;9:300-303. [DOI] |
| 20. | 李 克, 王 琳, 成 军, 张 玲霞, 陆 荫英, 李 莉, 刘 妍, 段 惠娟. 丙型肝炎病毒NS2基因酵母双杂交"饵"载体构建及表达. 世界华人消化杂志. 2002;10:129-132. |
| 22. | Lahm A, Yagnik A, Tramontano A, Koch U. Hepatitis C virus proteins as targets for drug development: the role of bioinformatics and modelling. Curr Drug Targets. 2002;3:281-96. [DOI] |
| 23. | Walewski JL, Gutierrez JA, Branch-Elliman W, Stump DD, Keller TR, Rodriguez A, Benson G, Branch AD. Mutation Master: profiles of substitutions in hepatitis C virus RNA of the core, alternate reading frame, and NS2 coding regions. RNA. 2002;8:557-571. [DOI] |
| 24. | Husmeier D, Wright F. A Bayesian approach to discriminate between alternative DNA sequence segmentations. Bioinformatics. 2002;18:226-234. [PubMed] [DOI] |
| 25. | Kernebeck T, Lohse AW, Grotzinger J. A bioinformatical approach suggests the function of the autoimmune hepatitis target antigen soluble liver antigen/liver pancreas. Hepatology. 2001;34:230-233. [PubMed] [DOI] |
| 26. | Husmeier D, Wright F. Probabilistic divergence measures for detecting interspecies recombination. Bioinformatics. 2001;17:S123-S131. [PubMed] [DOI] |
| 27. | Klinck R, Westhof E, Walker S, Afshar M, Collier A, Aboul-Ela F. A potential RNA drug target in the hepatitis C virus internal ribosomal entry site. RNA. 2000;6:1423-1431. [PubMed] [DOI] |
| 28. | Cheng J. Progress in gene therapy for liver diseases. J Gastroenterol Hepatol. 1999;14:A261-A262. |
| 29. | 成 军, 陈 菊梅. 丙型肝炎病毒NS5A蛋白的生物学调节作用. 国外医学微生物学分册. 2001;24:12-14. |
| 32. | Cheng J, Zhong YW, Liu Y, Dong J, Yang JZ, Zhang LX. Screening and identification of a humanized single chain variable region antibody for hepatitis C virus non-structural 3 protein. Chin J Immunol. 2000;16:246-249. |
| 34. | 成 军, 施 双双, 钟 彦伟, 夏 小兵, 王 刚, 王 琳, 刘 妍, 陈 菊梅. 丙型肝炎病毒核心蛋白可溶性单链可变区抗体在大肠杆菌中的表达. 解放军医学杂志. 2000;25:394-397. |
| 35. | 成 军, 钟 彦伟, 施 双双, 王 刚, 董 菁, 夏 小兵, 杨 继珍, 陈 菊梅. HCV非结构蛋白NS5A人源单链抗体可变区基因的筛选与鉴定. 中华微生物和免疫学杂志. 2000;20:567. |
| 36. | 钟 彦伟, 成 军, 施 双双, 杨 继珍, 董 菁, 夏 小兵, 李 克, 刘 妍, 陈 菊梅. 丙型肝炎病毒非结构蛋白NS5A人源单链抗体的筛选与表达. 中华中西医杂志. 2001;2:97-99. |
| 37. | 钟 彦伟, 成 军, 施 双双, 王 刚, 夏 小兵, 田 小军, 李 莉, 张 玲霞. 抗丙肝病毒包膜蛋白E2人源单链抗体的制备及免疫组织化学研究. 中华肝脏病杂志. 2002;10:109-112. |
| 38. | 钟 彦伟, 王 松山, 赵 景民, 成 军, 张 玲霞. 抗丙肝病毒非结构蛋白NS3单链抗体的制备及免疫组织化学研究. 中华实验与临床病毒学杂志. 2001;15:186-188. |
| 39. | 成 军, 钟 彦伟, 施 双双, 倪 勤, 刘 妍, 王 刚, 董 菁, 夏 小兵, 刘 友昭, 王 琳. HCV非结构蛋白NS5A人源单链可变区抗体基因的筛选与鉴定. 中华实验与临床病毒学杂志. 2001;15:216-218. |
| 44. | 成 军, 钟 彦伟, 刘 妍, 董 菁, 杨 继珍, 杨 守纯. 丙型肝炎病毒非结构区NS3基因在大肠杆菌中的可诱导性高表达. 中华实验与临床病毒学杂志. 2002;16:85-87. |
| 45. | 钟 彦伟, 成 军, 陈 新华, 王 刚, 洪 源, 王 琳, 李 莉, 张 玲霞, 陈 菊梅. 丙型肝炎病毒非结构蛋白NS4A抗原模拟表位的筛选和鉴定. 免疫学杂志. 2002;18:42-45. |
| 46. | 钟 彦伟, 成 军, 陈 新华, 王 刚, 洪 源, 王 琳, 李 莉, 张 玲霞, 陈 菊梅. 应用噬菌体表面展示技术筛选丙型肝炎病毒NS5A抗原模拟表位. 世界华人消化杂志. 2002;10:133-136. |
| 49. | 刘 妍, 成 军, 王 刚, 李 克, 段 惠娟, 王 琳, 董 菁, 洪 源, 张 跃新, 李 莉. 应用抑制性消减杂交技术克隆丙型肝炎病毒核心蛋白反式激活基因. 解放军医学杂志. 2001;26:880-883. |