修回日期: 2024-04-07
接受日期: 2024-04-15
在线出版日期: 2024-04-28
长链非编码RNA长基因间非蛋白编码RNA 996(long intergenic non-protein coding RNA 996, LINC00996)在多种肿瘤中发挥促癌或抑癌作用, 而其在胃癌中的表达和作用尚不清楚.
探索LINC00996在胃癌组织和细胞系中的表达, 并探讨其对胃癌细胞生物学行为的作用及机制.
用生物信息学法分析胃癌中LINC00996的表达, 及其对胃癌患者总生存期的影响. 用RT-qPCR检测胃癌及癌旁组织、胃癌及正常胃上皮细胞系中LINC00996表达. 胃癌细胞(SGC7901、NCI-N87)转染针对LINC00996的小干扰RNA(si-LINC00996)和细胞周期蛋白依赖性激酶抑制蛋白2A(cyclin-dependent kinase inhibitor 2A, CDKN2A)的小干扰RNA(si-CDKN2A)后, 用细胞计数试剂盒-8法、EDU染色法、流式细胞术法、划痕法和Transwell法分别检测细胞增殖、细胞周期、凋亡、迁移与侵袭; Western blot法检测细胞中CDKN2A、周期蛋白D1、B淋巴细胞瘤-2(B-cell lymphoma-2, Bcl-2)、Bcl-2相关X蛋白(Bcl-2 associated X protein, Bax)和劈开的半胱胺酸蛋白酶-3(Cleaved cysteinyl aspartate-specific proteinase-3, Cleaved caspase-3)表达.
LINC00996在胃癌组织和细胞中表达较癌旁组织和胃正常细胞系显著升高(P<0.05), Kaplan Meier Plotter数据库显示LINC00996高表达组的胃癌患者的总生存期较低表达组显著缩短(P<0.05). 敲降LINC00996能抑制胃癌细胞增殖、细胞周期运行、迁移、侵袭(P<0.05), 促进细胞凋亡(P<0.05); 降低cyclin D1和Bcl-2表达(P<0.05); 升高CDKN2A、Bax和Cleaved caspase-3表达(P<0.05). 敲降CDKN2A能部分逆转敲降LINC00996对胃癌细胞的作用(P<0.05).
敲降LINC00996能通过上调CDKN2A诱导胃癌细胞凋亡, 抑制其增殖、迁移与侵袭.
核心提要: 长基因间非蛋白编码RNA 996在胃癌中发挥促癌作用, 敲降其表达能抑制胃癌细胞增殖、迁移与侵袭并促进凋亡; 其发挥功能可能与调节细胞周期蛋白依赖性激酶抑制蛋白2A表达有关.
引文著录: 程国雄, 刘明, 陈正伟, 叶巧萍. 长链非编码RNA LINC00996通过抑制CDKN2A促进胃癌进展. 世界华人消化杂志 2024; 32(4): 302-312
Revised: April 7, 2024
Accepted: April 15, 2024
Published online: April 28, 2024
The expression and function of the long chain noncoding RNA (lncRNA) long intergenic non-protein coding RNA 996 (LINC00996) in gastric cancer remain elusive, despite its established pro- or anti-tumor roles across various tumor types.
To investigate the expression of LINC00996 in gastric cancer tissues and cell lines, and to explore its role and underlying mechanism in the biological behavior of gastric cancer cells.
Bioinformatics was used to analyze the expression of LINC00996 in gastric cancer and its effect on the overall survival of patients with gastric cancer. The expression of LINC00996 in gastric cancer and adjacent tissues as well as gastric cancer and normal gastric epithelial cell lines was detected by RT-qPCR. Gastric cancer cells (SGC7901 and NCI-N87) were transfected with small interfering RNA targeting LINC00996 (si-LINC00996) and cyclin-dependent kinase inhibitor 2A (CDKN2A)(si-CDKN2A), and cell proliferation, cell cycle progression, apoptosis, migration, and invasion were detected by cell counting kit-8, EDU staining, flow cytometry, scratch assay, and Transwell assay, respectively. The expression of CDKN2A, cyclin D1, B-cell lymphoma-2 (Bcl-2), Bcl-2 associated X protein (Bax), and cleaved cysteinyl aspartate-specific proteinase-3 (caspase-3) was detected by Western blot.
The expression of LINC00996 in gastric cancer tissues and cells was significantly higher than that in adjacent tissues and normal gastric cell lines (P < 0.05). Survival analysis based on the Kaplan-Meier Plotter database showed that the overall survival of gastric cancer patients with high expression of LINC00996 was significantly shorter than that of patients with low expression (P < 0.05). Knockdown of LINC00996 inhibited the proliferation, cell cycle progression, migration, and invasion of gastric cancer cells (P < 0.05), promoted apoptosis (P < 0.05), decreased the expression of cyclin D1 and Bcl-2 (P < 0.05), and increased the expression of CDKN2A, Bax, and cleaved caspase-3 (P < 0.05). Knockdown of CDKN2A partially reversed the effect of LINC00996 knockdown on gastric cancer cells (P < 0.05).
Knockdown of LINC00996 can induce apoptosis of gastric cancer cells and inhibit their proliferation, migration, and invasion by up-regulating CDKN2A expression.
- Citation: Cheng GX, Liu M, Chen ZW, Ye QP. Long non-coding RNA LINC00996 promotes gastric cancer progression by inhibiting CDKN2A. Shijie Huaren Xiaohua Zazhi 2024; 32(4): 302-312
- URL: https://www.wjgnet.com/1009-3079/full/v32/i4/302.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v32.i4.302
胃癌是常见的消化道恶性肿瘤[1]. 尽管临床治疗策略不断改善, 但大多数患者在确诊时已发生转移, 导致5年总生存期未明显延长[2,3]. 因此, 除了及早诊断和治疗外, 还需要进一步明确胃癌发生和转移的分子机制, 并采用靶向药物等手段阻断其发生和转移的信号通路以延长患者生存期[4,5]. 然而, 目前对影响胃癌发展及侵袭转移的关键靶点仍不清楚.
长链非编码RNA(long non-coding RNA, lncRNA)在癌症发展和转移中扮演重要角色[6,7], 但它们类型、数量和作用机制多样, 目前大部分尚不清楚. 因此有必要从众多胃癌细胞与正常组织中筛选出差异表达的功能性lncRNA, 并研究其调控细胞生物学行为的分子机制. 长基因间非蛋白编码RNA 996(long intergenic non-protein coding RNA 996, LINC00996)为新近被鉴定的lncRNA, 定位于7号染色体(7q36.1). 已有研究[8-11]表明LINC00996在肺癌、结直肠癌和多发性骨髓瘤中下调表达, 具有抑制癌细胞的作用; 而在弥漫性大B细胞淋巴瘤、宫颈鳞状细胞癌、食管癌和急性髓系白血病中上调表达, 促进了癌细胞的生长. 然而, 在胃癌中LINC00996的表达及其生物学作用尚不清楚. 因此, 本研究分析了LINC00996在胃癌中的表达情况, 并探讨了其对于胃癌细胞增殖、凋亡、迁移和侵袭的影响以及潜在机制.
1.1.1 临床组织样本收集: 经我院伦理委员会审批[批件号: 2020临审第(03-01)号]并获得患者知情同意书签署, 本研究收集了50例在2021-05/2023-05期间接受胃癌切除术的患者样本[包含胃癌组织和癌旁(距离癌浸润边缘5 cm)组织]. 所有标本均保存在-80 ℃条件下.
1.1.2 细胞: 永生化胃上皮细胞GES和胃癌细胞系(SGC7901、NCI-N87, BGC-823)均购自武汉普诺赛生命科技有限公司.
1.1.3 主要试剂: RPMI-1640培养基和胎牛血清购自武汉尚恩生物技术有限公司; 针对LINC00996的小干扰RNA(si-LINC00996)、针对细胞周期蛋白依赖性激酶抑制蛋白2A(cyclin-dependent kinase inhibitor 2A, CDKN2A)的小干扰RNA(si-CDKN2A)、阴性无关的小干扰RNA(si-NC)由北京六合华大基因科技有限公司合成; Trizol试剂购自北京百草泰克技术有限公司; InvitroRNA转染试剂购自南京英维沃生物科技有限公司; 逆转录试剂盒、RT-qPCR荧光定量试剂盒和细胞计数试剂盒-8(cell counting kit-8, CCK-8)细胞计数试剂盒购自上海经科化学科技有限公司; EDU-647细胞增殖检测试剂盒、细胞周期试剂盒、Annexin V-FITC细胞凋亡检测试剂盒、GAPDH抗体和HRP标记二抗购自上海碧云天生物技术有限公司; 周期蛋白D1(cyclin D1)、B淋巴细胞瘤-2(B-cell lymphoma-2, Bcl-2)、Bcl-2相关X蛋白(Bcl-2 associated X protein, Bax)和劈开的半胱胺酸蛋白酶-3(cleaved cysteinyl aspartate-specific proteinase-3, Cleaved caspase-3)抗体购自Santa Cruz公司; CDKN2A抗体购自美国Usbiological公司.
1.2.1 生物信息学分析: 根据GEPIA数据库(http://gepia.cancer-pku.cn/)检索LINC00996在胃癌中的表达情况; 利用Kaplan Meier Plotter数据库(https://kmplot.com/analysis/)获取LINC00996表达对胃癌总生存期(overall survival, OS)的影响曲线图.
1.2.2 检测胃癌及癌旁组织、胃癌细胞及胃正常上皮细胞系中LINC00996表达: 根据说明书步骤, 用Trizol试剂提取胃癌及癌旁组织、胃癌细胞及胃正常上皮细胞系中总RNA, 用超微量核酸蛋白检测仪测量总RNA纯度和浓度后, 各取1 μg总RNA用逆转录试剂盒合成总cDNA, 以cDNA为模板, U6为内参, 用RT-qPCR荧光定量试剂盒通过实时荧光定量PCR仪进行反应, 采用2-△△Ct方法计算出胃癌及癌旁组织、胃癌细胞及胃正常上皮细胞系中LINC00996表达量.
1.2.3 细胞培养及转染: 将人胃癌细胞(SGC7901和NCI-N87)置于含10%胎牛血清、100 U/mL青霉素和100 μg/mL链霉素的RPMI-1640培养液中, 之后将其放入培养箱(温度37 ℃、5%CO2)中进行培养, 每3 d更换1次培养液, 每日观察细胞生长状态, 密度达80%-90%时, 将培养基除去, 进行传代培养. 按照转染说明书步骤, 取生长状态良好的SGC7901和NCI-N87细胞, 分别稀释至1×106个细胞/mL后(无血清和无双抗的RPMI-1640培养液稀释), 接种于6孔板上, 每孔200 μL(1×106个), 待细胞融合度达80%, 采用InvitroRNA转染试剂进行分别将si-NC、si-LINC00996、si-LINC00996+si-NC和si-LINC00996+si-CDKN2A转染至细胞内, 记为si-NC组、si-LINC00996组、si-LINC00996+si-NC组和si-LINC00996+si-CDKN2A组, 转染48 h后进行后续实验.
1.2.4 细胞增殖检测: 用CCK-8法和EDU渗入法评估细胞增殖. CCK-8检测步骤: 分别将各组细胞接种至96孔板(每孔5000个细胞), 培养48 h后, 加入CCK-8试剂(每孔10 μL)并37 ℃孵育2 h, 用酶标仪检测各孔吸光度OD值, 细胞活性 = (实验组OD值/对照组OD值)×100%. EDU渗入法步骤: 分别将各组细胞接种至96孔板(每孔5000个细胞), 加入EDU-647检测液(每孔10 μL)并37 ℃孵育2 h, DAPI染核, 荧光显微镜下观察并计数EDU阳性细胞数.
1.2.5 细胞周期检测: 分别将各组细胞接种至6孔板(每孔5×106个细胞), 培养48 h后, 用无EDTA的0.2%胰酶消化, 1500 g离心5 min收集细胞, 用冷PBS洗涤2次, 用冷无水乙醇-20 ℃固定过夜, 再用冷PBS洗涤2次, 加入0.2 mL PI溶液(50 μg/mL)室温避光孵育30 min, 流式细胞仪上机检测.
1.2.6 细胞凋亡检测: 分别将各组细胞接种至6孔板(每孔5×106个细胞), 培养48 h后, 用无EDTA的0.2%胰酶消化, 1500 g离心5 min收集细胞, 用冷PBS洗涤2次, 加入0.5 mL结合缓冲液制成细胞悬液, 加入5 μL Annexin V-FITC试剂和10 μL PI试剂(50 μg/mL)室温避光孵育10 min, 流式细胞仪上机检测.
1.2.7 划痕实验检测细胞迁移: 取各组细胞, 用无血清RPMI-1640培养液稀释成1×105个/mL的细胞悬液, 并接种于载玻片上, 待细胞成单层汇合时, 用大头针尾端划线, 继续培养48 h, 倒置显微镜下拍照划痕后0 h和48 h的细胞划痕宽度. 愈合距离 = 0 h细胞划痕宽度-48 h细胞划痕宽度, 细胞迁移 = (实验组愈合距离/对照组愈合距离)×100%.
1.2.8 Transwell检测细胞侵袭: 用基质胶(1:8稀释)涂覆Transwell嵌套的聚酯纤维膜(0.8 μm孔径), 待风干后, 将包被后的嵌套置于24孔板中. 取各组细胞, 用无血清RPMI-1640培养液稀释成1×105个/mL的细胞悬液, 取200 μL细胞悬液加入嵌套中, 在下室加600 μL含10%胎牛血清的RPMI-1640培养液, 37 ℃孵育24 h. 取出嵌套, 擦除聚酯纤维膜上层内的细胞, 用4%多聚甲醛固定细胞20 min, 0.1%结晶紫溶液染色15 min. 倒置显微镜下, 任选5个视野对细胞计数.
1.2.9 Western blot检测蛋白表达: 用细胞蛋白质裂解液萃取各组细胞总蛋白质, 对各组蛋白质样本定量后, 各取30 μg蛋白质进行Western blot转印至PVDF膜上. 用5%脱脂奶粉对PVDF膜进行封闭后, 加Cleaved-caspase-3(1:1000)、cyclin D1(1:1000)、Bax(1:2000)、Bcl-2(1:1000)、CDKN2A(1:1000)和GAPDH(1:6000)抗体于摇床上室温孵育, 共孵育2 h, 用TBST洗膜3次, 5 min/次, 然后加HRP-二抗(1:2000)于摇床上室温孵育, 共孵育1 h, 用TBST洗膜3次, 10 min/次. 用ECL化学发光试剂显影, 以GAPDH为内参, 相对条带灰度光密度值采用Image J软件分析.
统计学处理 采用SPSS 19.0统计软件进行分析处理. 计量资料采用均数±标准差(mean±SD)描述, 多组间比较采用单因素方差分析, 两组间比较采用独立样本t检验. P<0.05为差异具有统计学意义.
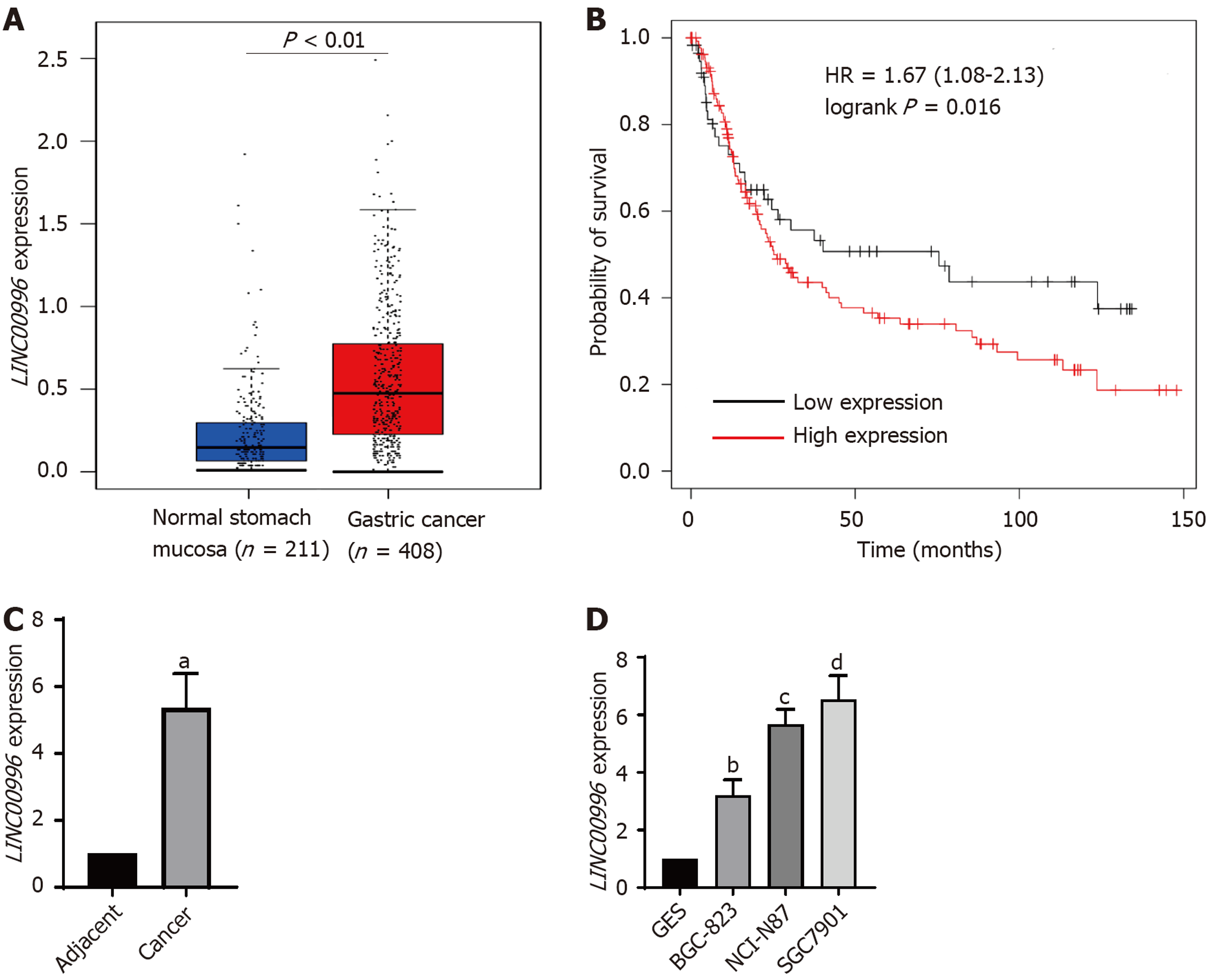
首先用GEPIA数据库获取LINC00996在胃癌组织(n = 408例)和正常胃黏膜组织(n = 211例)中的表达情况, 与正常胃黏膜组织比较, 胃癌组织中LINC00996表达显著上调(P<0.01), 见图1A. 用Kaplan Meier Plotter数据库分析LINC00996表达对胃癌患者OS的影响, 结果(图1B)显示, 以LINC00996表达量的中位值分为高表达组(n = 184例)和低表达组(n = 89例), 与低表达组比较, 高表达组的胃癌患者OS明显缩短(P = 0.016). 通过RT-qPCR分析胃癌及癌旁组织、胃癌细胞及胃正常上皮细胞系中LINC00996表达(图1C和D), 与癌旁组织和胃正常上皮细胞系比较, 胃癌组织及胃癌细胞系中LINC00996表达均显著增高(P<0.01). 进一步分析了LINC00996表达与胃癌患者临床病理特征的关系, 如表1所示, 在50例胃癌标本中, 有淋巴结转移、TNM(Ⅲ-Ⅳ期)、淋巴管血管侵袭的胃癌组织中LINC00996表达水平显著高于无淋巴结转移、TNM(Ⅰ-Ⅱ期)、无淋巴管血管侵袭的胃癌组织(P<0.01); 但LINC00996表达与性别、年龄、肿瘤部位和大小无关(P>0.05).
| 临床特征 | 例数(n) | LINC00996表达水平 | t或F值 | P |
| 年龄(岁) | 0.354 | >0.05 | ||
| <60 | 20 | 5.24±0.85 | ||
| ≥60 | 30 | 5.48±1.04 | ||
| 性别 | 0.689 | >0.05 | ||
| 男 | 37 | 5.29±1.21 | ||
| 女 | 13 | 5.46±1.38 | ||
| 肿瘤大小(cm) | 0.402 | >0.05 | ||
| <3 | 16 | 5.35±1.02 | ||
| ≥3 | 34 | 5.42±1.44 | ||
| 肿瘤部位 | 1.302 | >0.05 | ||
| 上三分之一 | 16 | 5.12±0.73 | ||
| 中三分之一 | 11 | 5.42±1.15 | ||
| 下三分之一 | 23 | 5.34±1.23 | ||
| 浸润深度 | 5.324 | <0.01 | ||
| 局限于浆膜下层 | 15 | 3.54±0.56 | ||
| 超过浆膜下层 | 35 | 6.21±1.32 | ||
| 淋巴结转移 | 6.793 | <0.01 | ||
| 阴性 | 13 | 3.02±0.63 | ||
| 阳性 | 37 | 6.42±1.53 | ||
| TNM分期 | 4.895 | <0.01 | ||
| Ⅰ-Ⅱ期 | 19 | 3.74±0.68 | ||
| Ⅲ-Ⅳ期 | 31 | 6.35±1.42 | ||
| 淋巴管血管侵袭 | 5.189 | <0.01 | ||
| 阴性 | 32 | 4.02±0.71 | ||
| 阳性 | 18 | 7.76±1.39 |
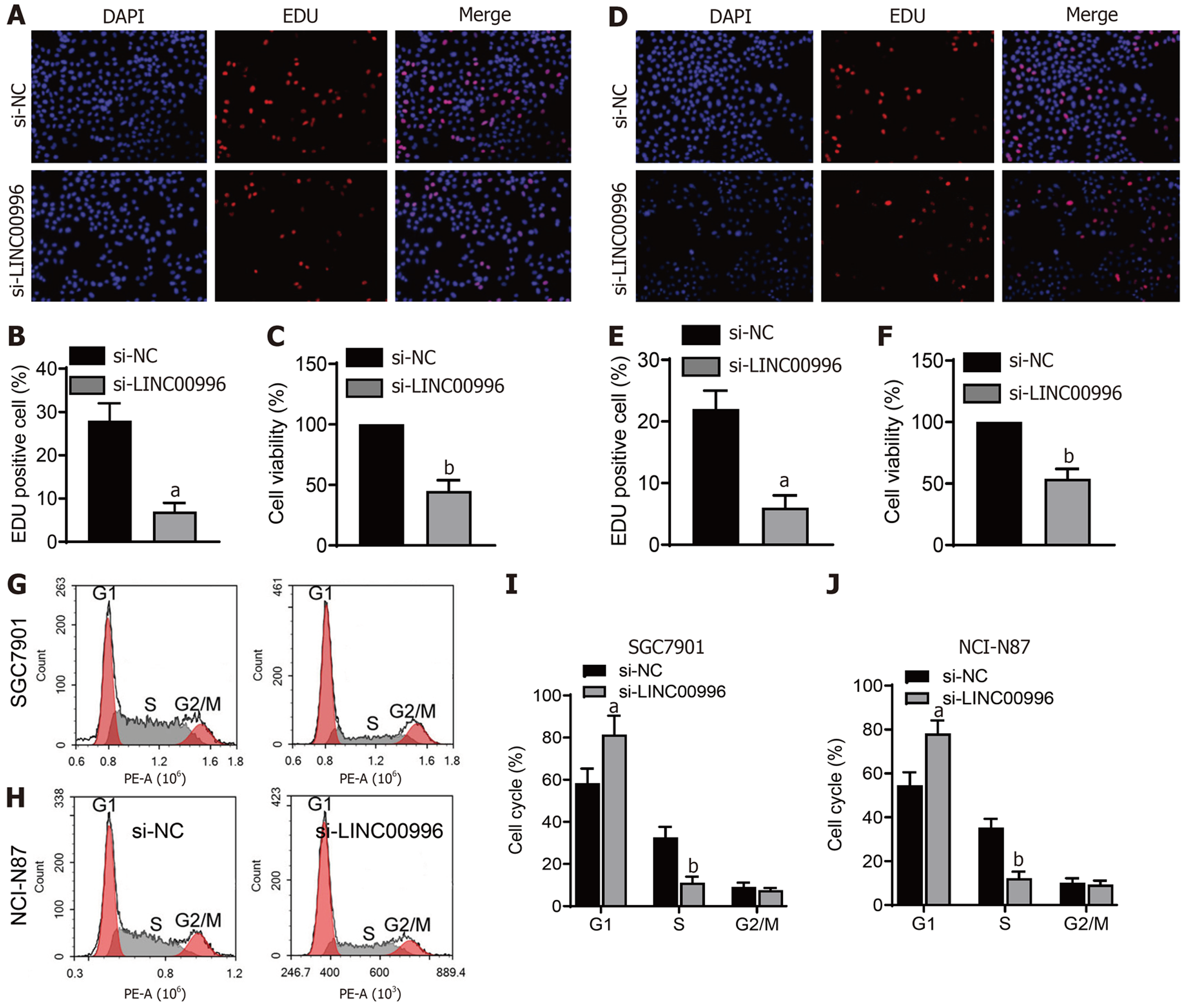
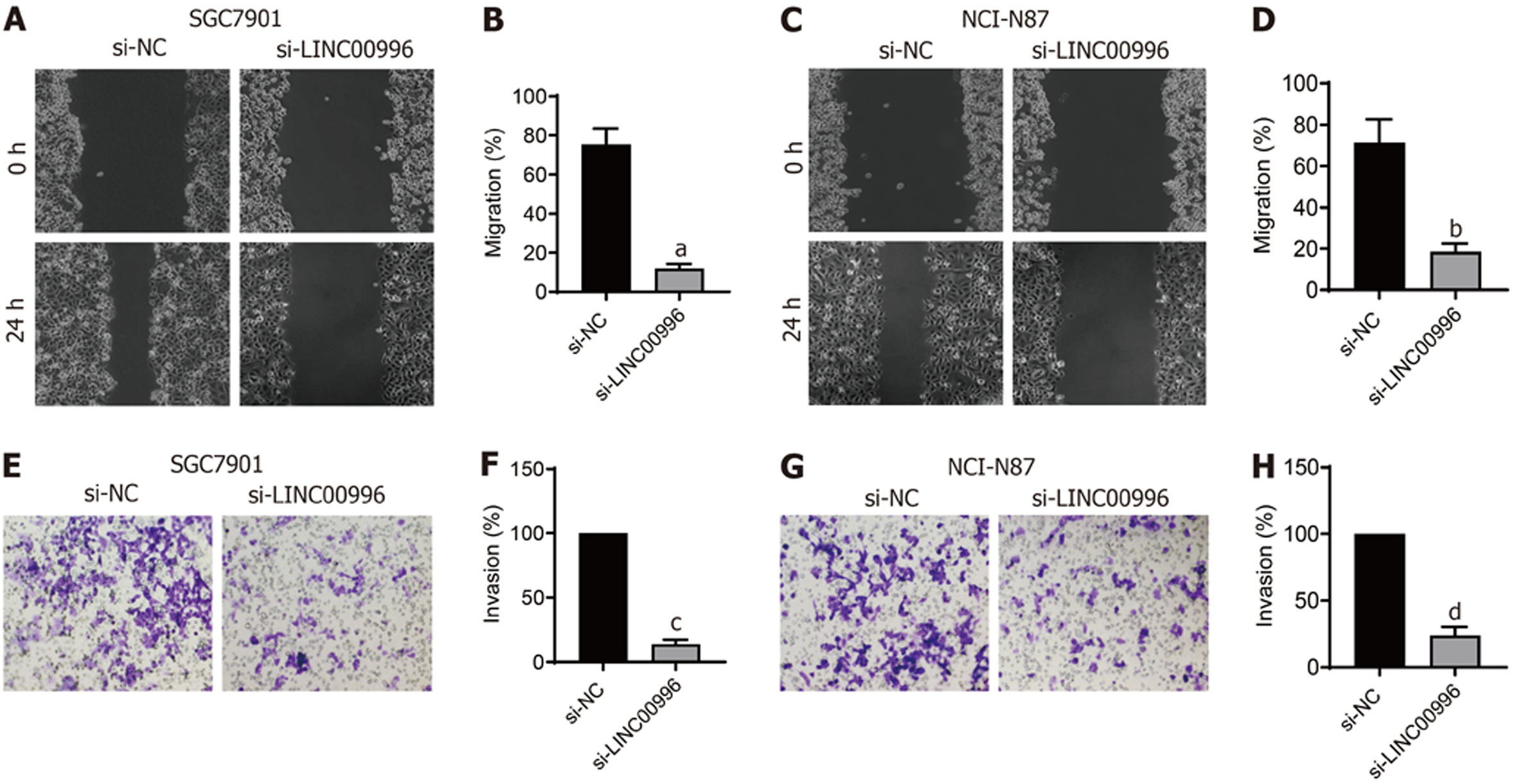
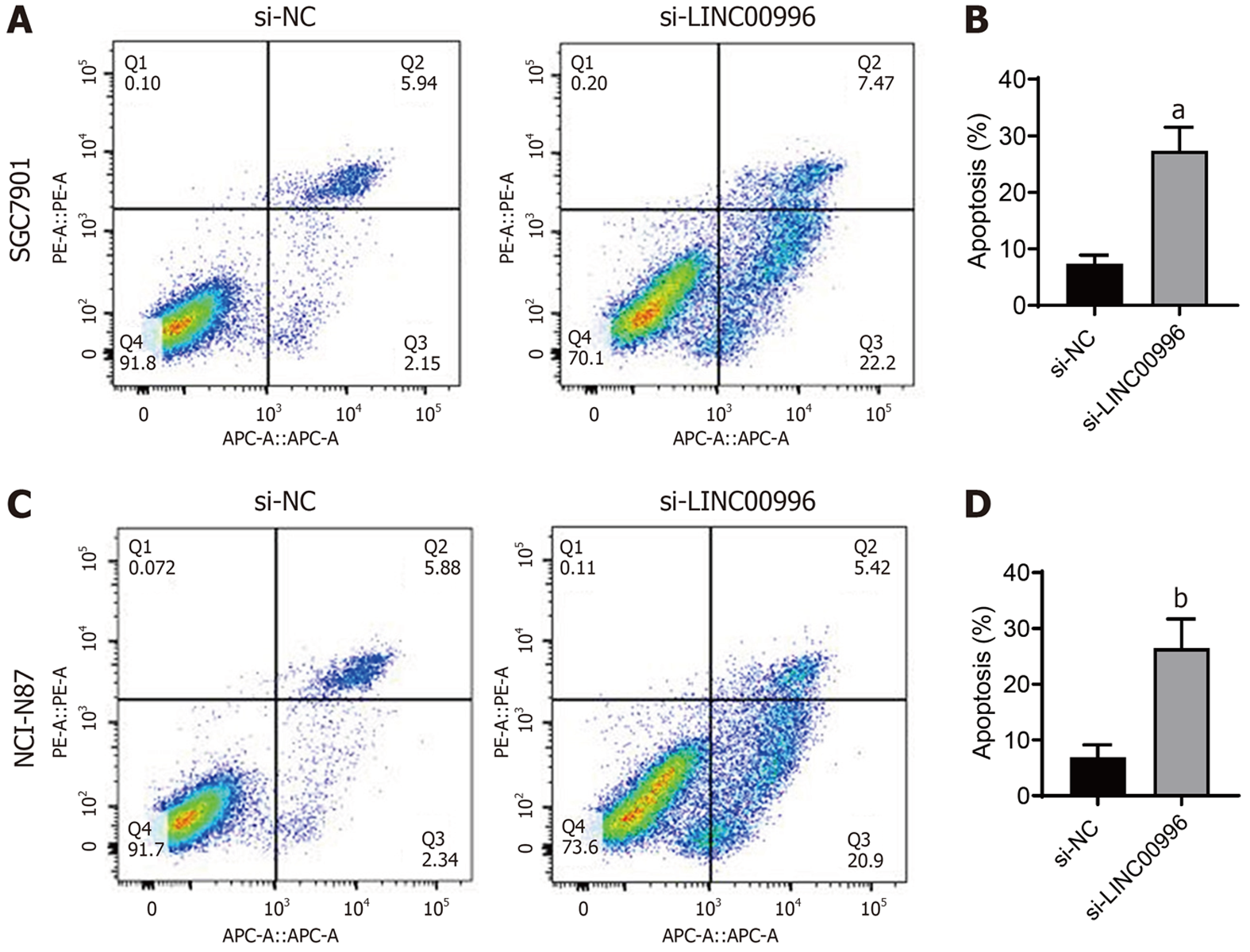
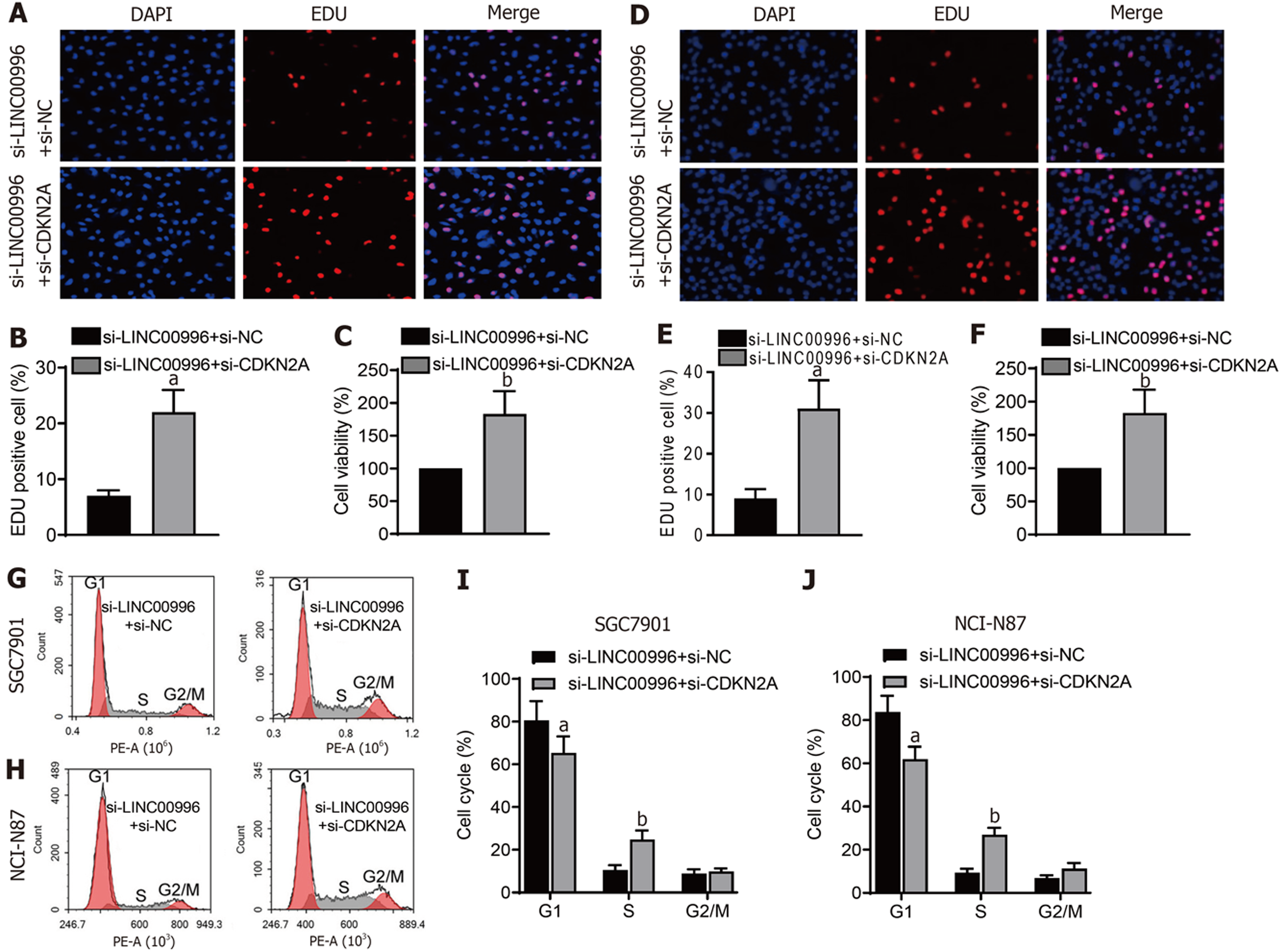
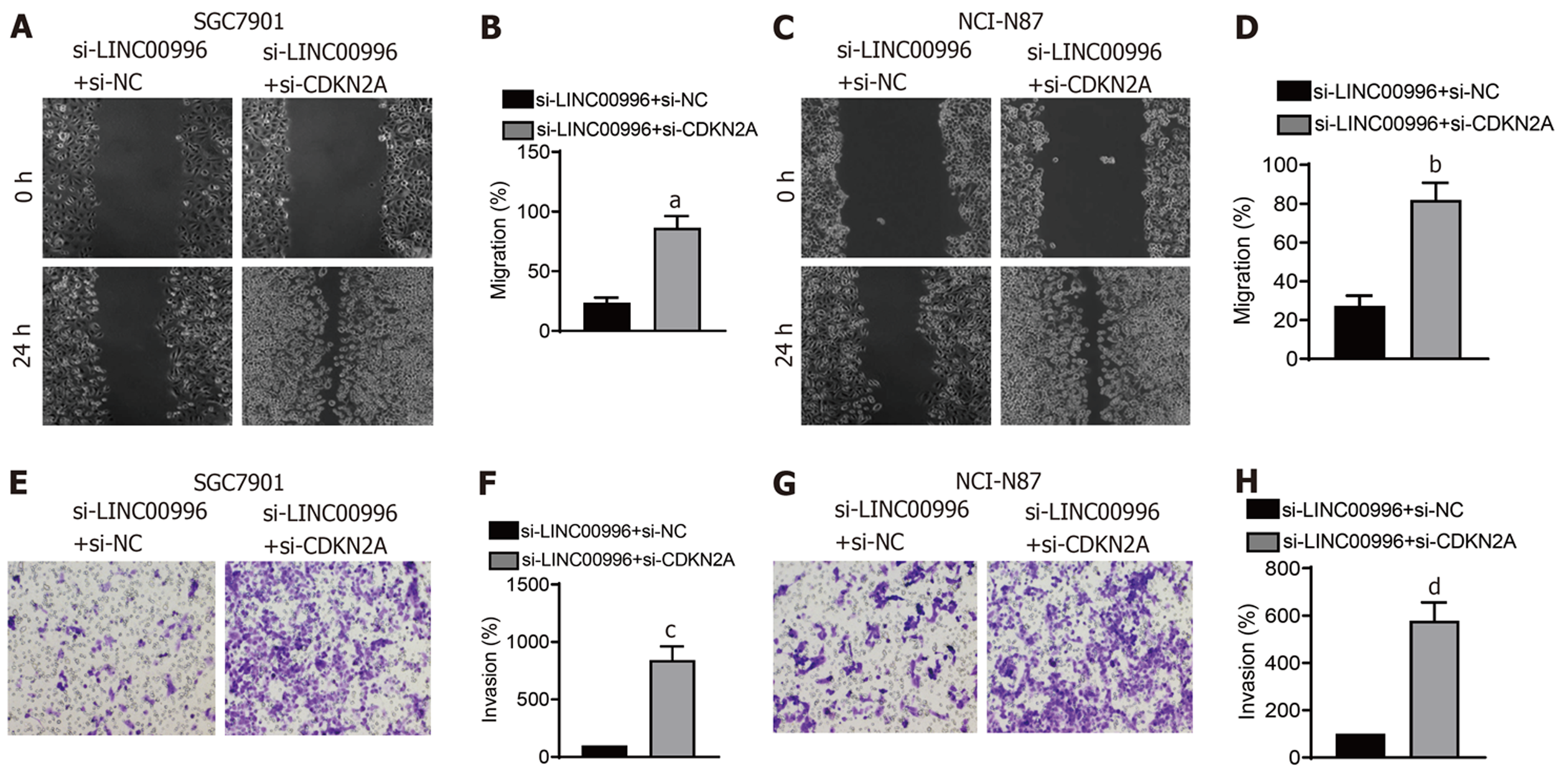
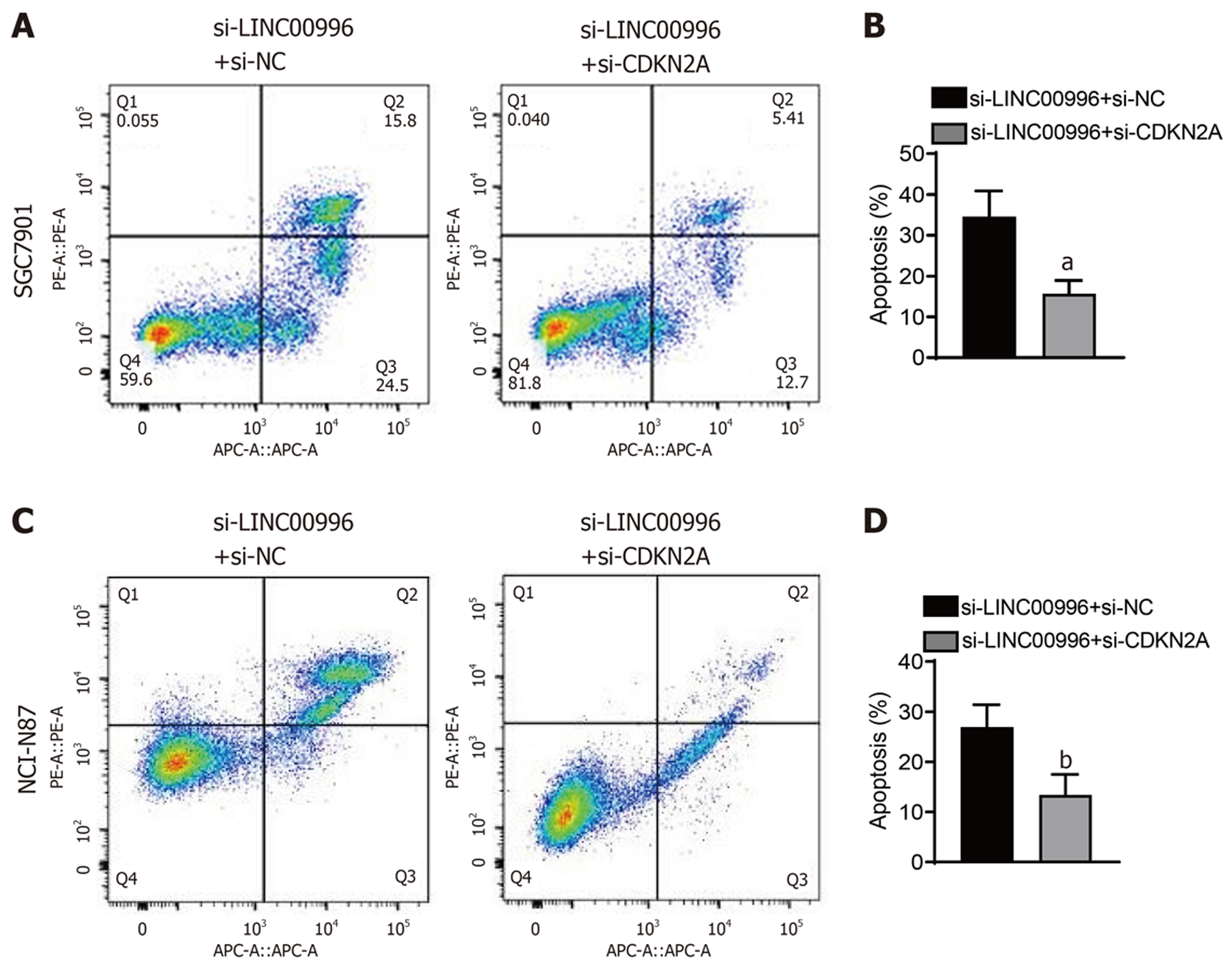
用EDU染色和CCK-8法评估敲降LINC00996抑制胃癌细胞增殖的影响, 如图2A-F所示, 与si-NC组比较, si-LINC00996组SGC7901细胞(图2A-C)和NCI-N87细胞(图2D-F)的EDU阳性细胞数和细胞活力均显著下降(P<0.01), 说明敲降LINC00996抑制胃癌细胞增殖. 进一步检测了敲降LINC00996对胃癌细胞周期的影响, 如图2G-J所示, 与si-NC组比较, si-LINC00996组SGC7901细胞(图2G和I)和NCI-N87细胞(图2H和J)的G1期比例明显增高(P<0.01), S期比例明显降低(P<0.01), 说明敲降LINC00996能使得胃癌细胞周期阻滞于G1期. 用划痕实验研究了敲降LINC00996对胃癌细胞迁移的影响, 如图3A-D所示, 与si-NC组比较, si-LINC00996组SGC7901细胞(图3A和B)和NCI-N87细胞(图3C和D)的细胞迁移能力明显降低(P<0.01). 用Transwell实验研究了敲降LINC00996对胃癌细胞侵袭的影响, 如图3E-H所示, 与si-NC组比较, si-LINC00996组SGC7901细胞(图3E和F)和NCI-N87细胞(图3G和H)的细胞侵袭能力明显降低(P<0.01). 用流式细胞术法检测了敲降LINC00996对胃癌细胞凋亡的影响, 如图4所示, 与si-NC组比较, si-LINC00996组SGC7901细胞(图4A和B)和NCI-N87细胞(图4C和D)的细胞凋亡明显增高(P<0.01).
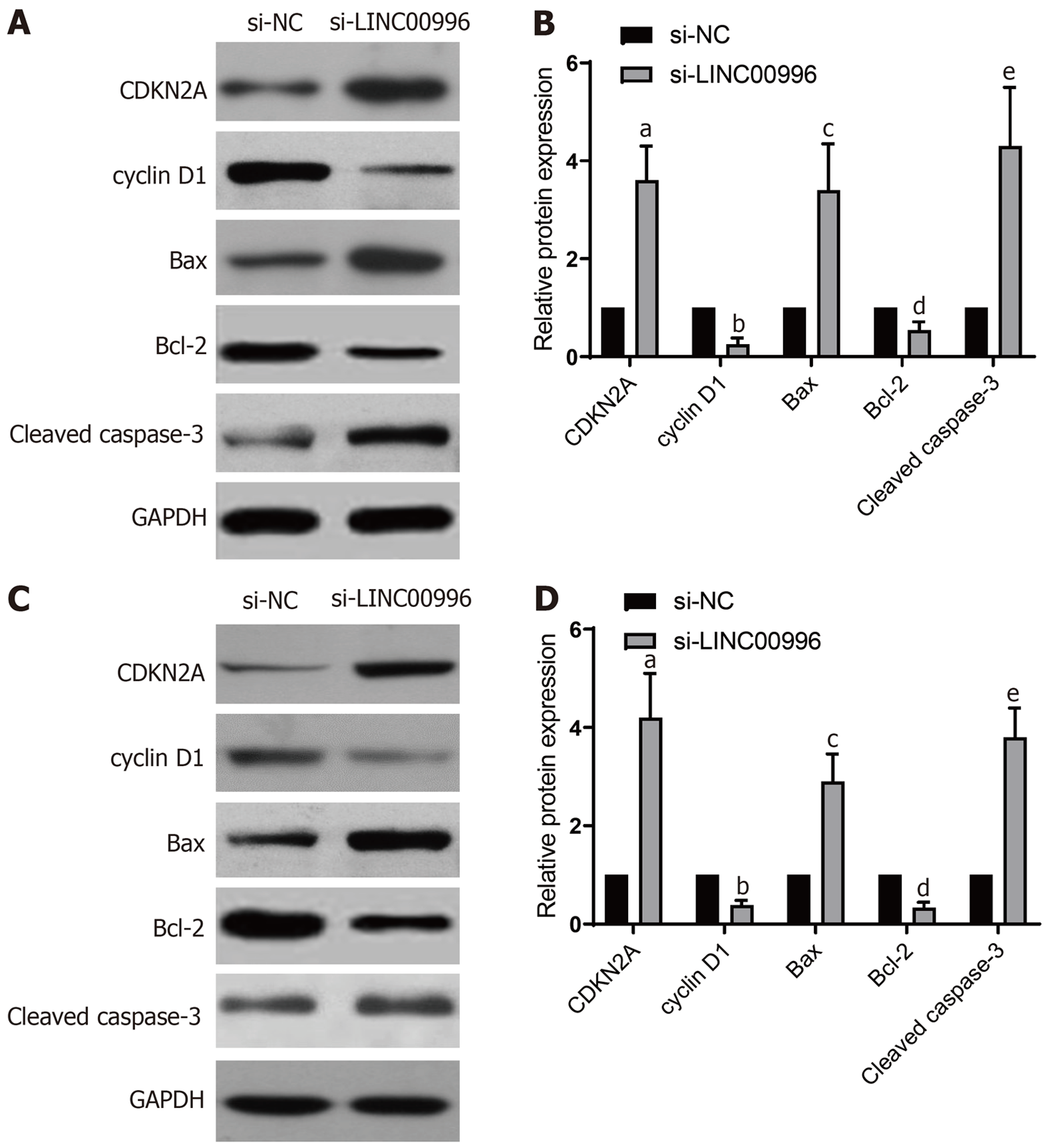
用Western blot法检测敲降LINC00996抑制胃癌细胞中CDKN2A、cyclin D1、Bax、Bcl-2和Cleaved caspase-3表达的影响, 如图5所示, 与si-NC组比较, si-LINC00996组SGC7901细胞(图5A和B)和NCI-N87细胞(图5C和D)中均CDKN2A、Bax和Cleaved caspase-3表达显著上调(P<0.01), 而cyclin D1和Bcl-2表达显著下调(P<0.01).
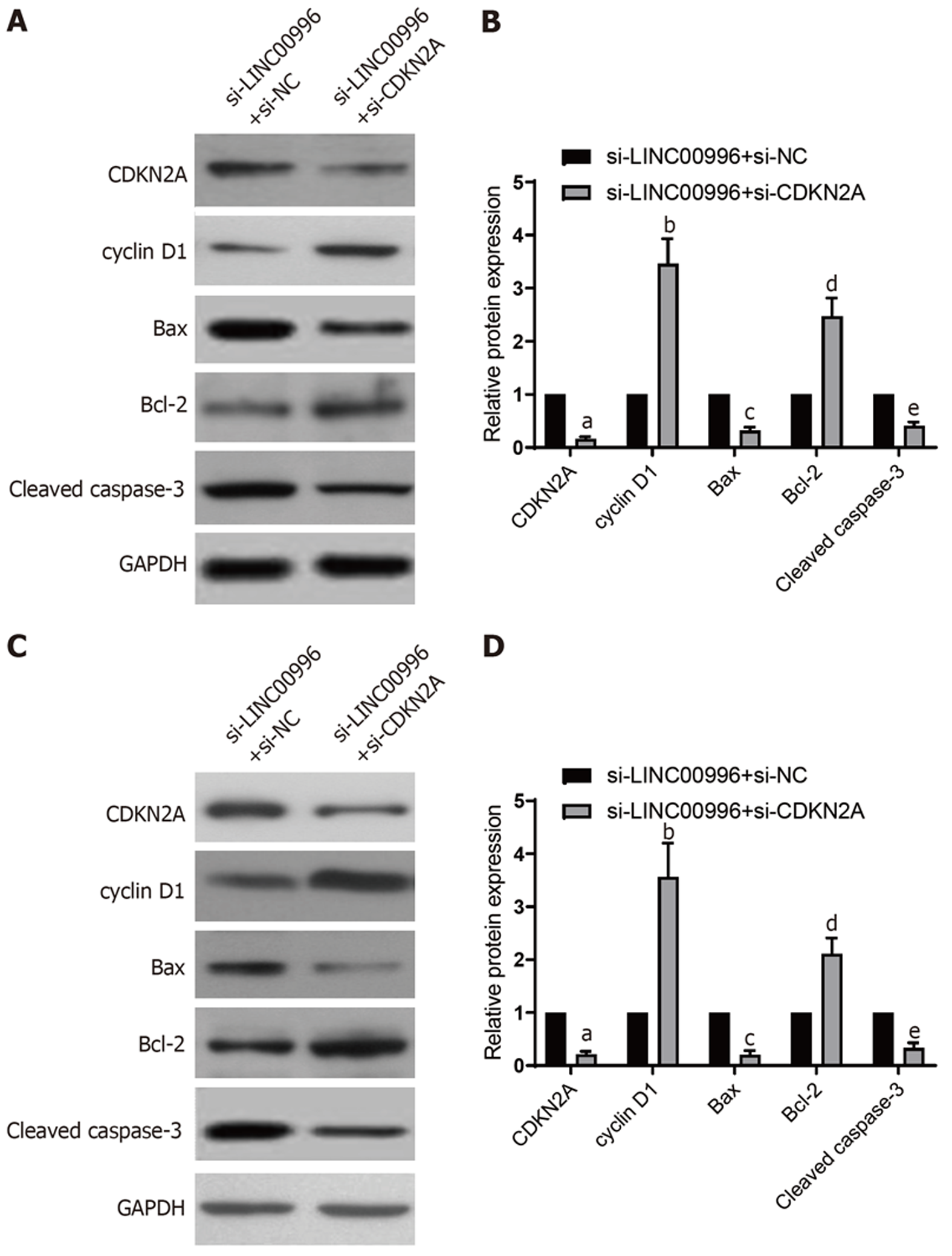
首先检测胃癌细胞增殖, 如图6A-F所示, 与si-LINC00996+si-NC组比较, si-LINC00996+si-CDKN2A组SGC7901细胞(图6A-C)和NCI-N87细胞(图6D-F)的EDU阳性细胞数和细胞活力均显著增加(P<0.01), 说明敲降CDKN2A能逆转敲降LINC00996对胃癌细胞增殖的抑制作用. 细胞周期检测结果如图6G-J所示, 与si-LINC00996+si-NC组比较, si-LINC00996+si-CDKN2A组SGC7901细胞(图6G和I)和NCI-N87细胞(图6H和J)的G1期比例明显降低(P<0.01), S期比例明显增高(P<0.01). 细胞迁移和细胞侵袭检测结果如图7所示, 与si-LINC00996+si-NC组比较, si-LINC00996+si-CDKN2A组SGC7901细胞(图7A、7B、7E和7F)和NCI-N87细胞(图7C、7D、7G和7H)的细胞迁移和细胞侵袭能力明显增加(P<0.01). 细胞凋亡检测结果如图8所示, 与si-LINC00996+si-NC组比较, si-LINC00996+si-CDKN2A组SGC7901细胞(图8A和B)和NCI-N87细胞(图8C和D)的细胞凋亡明显降低(P<0.01).
Western blot结果如图9所示, 与si-LINC00996+si-NC组比较, si-LINC00996+si-CDKN2A组SGC7901细胞(图9A和B)和NCI-N87细胞(图9C和D)中均CDKN2A、Bax和Cleaved caspase-3表达显著下调(P<0.01), 而cyclin D1和Bcl-2表达显著上调(P<0.01).
探索影响胃癌进展的关键靶点有助于制定胃癌的生物靶向治疗策略[12]. 本研究显示, LINC00996在胃癌中表达上调, 且其与淋巴结转移、TNM分期较高以及淋巴管血管侵袭有关; 进一步研究显示, 敲降LINC00996能抑制胃癌细胞增殖、迁移与侵袭; 这些结果说明LINC00996在胃癌发生促癌因子作用, 靶向敲降LINC00996表达可能是潜在的胃癌生物靶向治疗策略.
本研究进一步探讨了LINC00996在胃癌中发挥促癌因子作用的机制. 已有研究表明LINC00996是细胞周期调节点P16(也称为CDKN2A)的调节性LncRNA[13]. 本研究显示, 敲降LINC00996能上调胃癌细胞中CDKN2A的表达. 大量研究证实[14-16], CDKN2A能与cyclin D1竞争性结合细胞周期蛋白依赖性激酶4, 使得视网膜母细胞瘤蛋白磷酸化降低, 抑制细胞周期由G1期向S期转化, 导致细胞增殖降低. 另外, 上调CDKN2A能抑制多种肿瘤细胞迁移与侵袭并诱导其凋亡[17,18]. 本研究结果也显示, 敲降LINC00996在上调CDKN2A的同时, 伴随着下调cyclin D1, 胃癌细胞G1期进入S期受阻和增殖受到抑制, 且抑制细胞迁移与侵袭, 同时胃癌细胞中Bax表达上调, 而Bcl-2表达下调, Caspase 3剪切, 细胞凋亡增多. 拒此推测, 敲降LINC00996抑制胃癌细胞增殖、细胞周期运行、迁移和侵袭并促凋亡可能与上调CDKN2A表达有关. 为证明这样假设, 本研究通过共转si-LINC00996+si-CDKN2A以下调CDKN2A表达, 结果说明下调CDKN2A能逆转敲降LINC00996对胃癌细胞的抗肿瘤效应, 提示LINC00996在胃癌中可通过调节CDKN2A发挥促癌作用. 当然本研究依然存在一定的不足, 比如LINC00996是通过什么信号途径调节CDKN2A的表达的, 以及CDKN2A通过下游什么信号途径发挥生物学效应的; 另外, LINC00996是否还能通过其他信号途径在胃癌中发挥作用?这些未知的信息仍需要大量研究来探索.
综上所述, 本研究发现LINC00996在胃癌中表达上调, 且其表达量与淋巴结转移、TNM分期较高以及淋巴管血管侵袭有关, 敲降LINC00996能抑制胃癌细胞增殖、迁移与侵袭并促进凋亡, 另外敲降LINC00996的功能可能与上调CDKN2A有关. 另外, 本研究还提示LINC00996可能为胃癌的潜在分子靶标, 且敲降LINC00996可能为胃癌今后诊治的潜在策略.
长链非编码RNA(long non-coding RNA, lncRNA)在肿瘤的发生和进展中发挥重要作用, 但其种类和数量繁多, 需要筛选和鉴别出功能性lncRNA, 并研究其调控肿瘤进展的作用机制.
长基因间非蛋白编码RNA 996(long intergenic non-protein coding RNA 996, LINC00996)是一种新近鉴定的lncRNA, 在多种肿瘤类型中具有抑制或促进癌症发展的功能, 而其在胃癌中的作用和机制尚不清楚.
探索LINC00996在胃癌中的生物学功能及机制.
通过生物信息学法和临床样本检测LINC00996在胃癌中表达, 用细胞学实验检测敲降LINC00996对胃癌细胞活性、增殖、细胞周期、凋亡、迁移与侵袭及凋亡相关蛋白的影响, 并分析其与细胞周期蛋白依赖性激酶抑制蛋白2A(cyclin-dependent kinase inhibitor 2A, CDKN2A)的关系.
LINC00996在胃癌中上调表达, 其与总生存期较短、淋巴结转移、TNM分期较高以及淋巴管血管侵袭有关. 敲降其表达可能通过上调CDKN2A来发挥抑制胃癌细胞增殖、细胞周期运行、迁移与侵袭.
LINC00996在胃癌中发挥促癌作用.
LINC00996可能是胃癌诊断与不良预后的潜在标记物, 靶向LINC00996也可能是胃癌治疗的潜在策略.
学科分类: 胃肠病学和肝病学
手稿来源地: 浙江省
同行评议报告学术质量分类
A级 (优秀): 0
B级 (非常好): 0
C级 (良好): C, C, C, C
D级 (一般): 0
E级 (差): 0
科学编辑:张砚梁 制作编辑:张砚梁
| 1. | Smyth EC, Nilsson M, Grabsch HI, van Grieken NC, Lordick F. Gastric cancer. Lancet. 2020;396:635-648. [PubMed] [DOI] |
| 2. | Machlowska J, Baj J, Sitarz M, Maciejewski R, Sitarz R. Gastric Cancer: Epidemiology, Risk Factors, Classification, Genomic Characteristics and Treatment Strategies. Int J Mol Sci. 2020;21. [PubMed] [DOI] |
| 3. | Thrift AP, El-Serag HB. Burden of Gastric Cancer. Clin Gastroenterol Hepatol. 2020;18:534-542. [PubMed] [DOI] |
| 4. | Guan WL, He Y, Xu RH. Gastric cancer treatment: recent progress and future perspectives. J Hematol Oncol. 2023;16:57. [PubMed] [DOI] |
| 5. | Zeng Y, Jin RU. Molecular pathogenesis, targeted therapies, and future perspectives for gastric cancer. Semin Cancer Biol. 2022;86:566-582. [PubMed] [DOI] |
| 6. | 布力布• 吉力斯汉, 赛福丁• 柯尤木, 刘春晖, 王培红. 长链非编码RNA核富集转录子1对胃癌SGC7901细胞迁移、侵袭、自噬的影响及机制研究. 陕西医学杂志. 2023;52:1749-1753. [DOI] |
| 7. | Xing C, Sun SG, Yue ZQ, Bai F. Role of lncRNA LUCAT1 in cancer. Biomed Pharmacother. 2021;134:111158. [PubMed] [DOI] |
| 8. | Shen Z, Li X, Hu Z, Yang Y, Yang Z, Li S, Zhou Y, Ma J, Li H, Liu X, Cai J, Pu L, Wang X, Huang Y. Linc00996 is a favorable prognostic factor in LUAD: Results from bioinformatics analysis and experimental validation. Front Genet. 2022;13:932973. [PubMed] [DOI] |
| 9. | Ge H, Yan Y, Wu D, Huang Y, Tian F. Potential role of LINC00996 in colorectal cancer: a study based on data mining and bioinformatics. Onco Targets Ther. 2018;11:4845-4855. [PubMed] [DOI] |
| 10. | Zhou S, Fang J, Sun Y, Li H. Integrated Analysis of a Risk Score System Predicting Prognosis and a ceRNA Network for Differentially Expressed lncRNAs in Multiple Myeloma. Front Genet. 2020;11:934. [PubMed] [DOI] |
| 11. | Cai Z, Tang B, Chen L, Lei W. Mast cell marker gene signature in head and neck squamous cell carcinoma. BMC Cancer. 2022;22:577. [PubMed] [DOI] |
| 12. | Park JY, Herrero R. Recent progress in gastric cancer prevention. Best Pract Res Clin Gastroenterol. 2021;50-51:101733. [PubMed] [DOI] |
| 13. | Zeng T, Jiang S, Wang Y, Sun G, Cao J, Hu D, Wang G, Liang X, Ding J, Du J. Identification and validation of a cellular senescence-related lncRNA signature for prognostic prediction in patients with multiple myeloma. Cell Cycle. 2023;22:1434-1449. [PubMed] [DOI] |
| 14. | Knudsen ES, Kumarasamy V, Nambiar R, Pearson JD, Vail P, Rosenheck H, Wang J, Eng K, Bremner R, Schramek D, Rubin SM, Welm AL, Witkiewicz AK. CDK/cyclin dependencies define extreme cancer cell-cycle heterogeneity and collateral vulnerabilities. Cell Rep. 2022;38:110448. [PubMed] [DOI] |
| 15. | Safwan-Zaiter H, Wagner N, Wagner KD. P16INK4A-More Than a Senescence Marker. Life (Basel). 2022;12:1332. [PubMed] [DOI] |
| 16. | Knudsen ES, Witkiewicz AK, Rubin SM. Cancer takes many paths through G1/S. Trends Cell Biol. 2023. [PubMed] [DOI] |
| 17. | Shi WK, Li YH, Bai XS, Lin GL. The Cell Cycle-Associated Protein CDKN2A May Promotes Colorectal Cancer Cell Metastasis by Inducing Epithelial-Mesenchymal Transition. Front Oncol. 2022;12:834235. [PubMed] [DOI] |