修回日期: 2017-03-28
接受日期: 2017-04-05
在线出版日期: 2017-06-18
转录组是特定的细胞或组织在特定的时间或状态下转录出来的RNA集合, 转录组研究能够从整体水平研究基因功能以及基因结构, 并能很好的显示处于表达状态的基因数量和活跃程度. 作为转录组学新一代高通量测序技术之一, RNA-Seq技术能够更为快速、准确地为人们提供更多的生物体转录信息, 在生物医学研究中已经得到广泛应用. 随着全球胃肠肿瘤发病率的逐年提高, RNA-Seq技术在胃肠肿瘤研究领域进行全转录组测序分析的应用越来越多, 并取得了一些新的进展. 本文将就RNA-Seq技术原理、优势及其在胃肠肿瘤研究中的具体应用进行论述.
核心提要: 随着全球胃肠肿瘤发病率的逐年提高, 近年来应用RNA-Seq技术在胃肠肿瘤进行全转录组测序分析方面, 取得了一些新的进展. 本文将就RNA-Seq技术原理、优势及其在胃肠肿瘤研究中的具体应用进行论述.
引文著录: 江海丽, 李平, 张梅, 张锋利, 苏丽. RNA-Seq技术及其在胃肠肿瘤研究中的应用现状. 世界华人消化杂志 2017; 25(17): 1564-1571
Revised: March 28, 2017
Accepted: April 5, 2017
Published online: June 18, 2017
Transcriptome is an RNA set transcribed by a specific cell or tissue in a specific time or state, which allows to study gene function and structure from the overall level and can well display the number and activity of expressed genes. RNA-Seq, as a new high-throughput sequencing technology for transcriptome analysis, has been widely used in biomedical research, because it can provide organism transcription information more quickly and accurately. As the incidence of gastrointestinal cancer increases worldwide, the RNA-Seq technology has achieved some new progress in whole transcriptome resequencing of gastrointestinal tumors. In this article, we will discuss the principle and advantages of RNA-Seq technology as well as its application in the research of gastrointestinal tumors.
- Citation: Jiang HL, Li P, Zhang M, Zhang FL, Su L. RNA-Seq technology and its application in research of gastrointestinal tumors. Shijie Huaren Xiaohua Zazhi 2017; 25(17): 1564-1571
- URL: https://www.wjgnet.com/1009-3079/full/v25/i17/1564.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v25.i17.1564
随着后基因组时代的来临, 转录组学技术作为率先发展起来的新技术已得到广泛应用[1,2], 其为研究基因功能及基因结构的重要手段, 可以将基因组遗传信息与生物功能蛋白组联系起来[3], 研究的主要目的: 对转录本整个发育不同阶段的表达水平进行量化; 确定基因的剪接模式和其他转录后的修饰; 并且可以对转录产物进行分类[4]. 而RNA-Seq技术作为转录组学重要技术手段, 可利用高通量测序技术对组织或细胞中所有RNA反转录而成cDNA文库进行测序, 无需预先设计探针, 即可对任意物种的整体转录活动进行检测, 提供更精确的数字化信号, 更高的检测通量以及更广泛的检测范围[5-7]. RNA-Seq技术利用新一代高通量测序技术的优势, 已经广泛应用于肿瘤医学研究领域, 并取得了一定的进展.
一般来说, 所有高通量测序技术都能进行RNA测序. 以Illumina公司的Solexa技术、Roche公司的454技术和ABI公司的SOLiD技术为标志的新一代测序, 相对于传统的Sanger测序而言, 新一代测序主要特点是测序通量高, 测序时间和成本显著下降, 相应技术平台各有其特点[8,9].
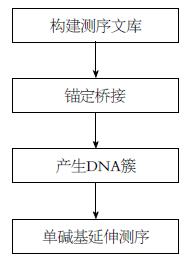
近两年来, Illumina/Solexa测序平台不断升级, 相继推出了GA IIx、HiSeq 2000等测序仪[10,11], GA(Genome Analyzer)核心技术是"DNA簇"和"可逆性末端终结", 采用边合成边测序的原理, 利用桥式PCR扩增的方法, 将DNA片段固定到流动池中, 每次加入不同荧光素标记的4种碱基中的一种, SBS, 用高分辨率电荷耦合装置采集数据, 将荧光颜色信号转换为DNA序列[12,13]. Illumina测序技术与其他3种测序技术相比具有的优点: (1)在单核苷酸水平下, 能检测任何物种的转录活动; (2)能够分析转录本的结构和表达水平, 同时最重要的是可以找到稀有的以及以前不曾发现的转录本; (3)能够高度精确的识别单核苷酸的多态性和可变剪切位点的编码序列, 对转录组信息能进行全面的分析; (4)测序成本最低. 其测序流程如图1[14].
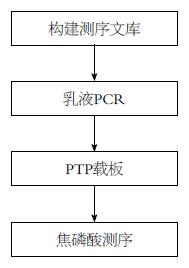
454技术平台是基于焦磷酸测序法的超高通量基因组测序系统, 其推出的GS FLX(Genome Sequencer FLX System)测序系统, 利用磁珠吸附DNA分子, 再乳化成微滴进行PCR扩增, 连接着扩增产物的磁珠被加到PTP板中, 测序开始后每次反应加入4种碱基中的一种, 当配对的碱基合成到引物上时, 释放焦磷酸分子, 将荧光素氧化发出荧光, 通过检测荧光信号的有无达到测序的目的. 其突出优势是性能优, 读片长, 但准确率较低, 成本高[15], 其测序流程如图2.
SOLiD(supported oligo ligation detection)系统在文库构建和PCR扩增方面与GS FLX系统类似, 微珠通过接头捕获DNA片段, 并进行乳液PCR扩增, 扩增结束后磁珠共价结合到SOLiD玻片表面, 随后加入4种荧光染料标记的8种碱基单链探针, 每次反应向引物上连接一种探针, 仪器记录下该探针的荧光信号后, 酶切掉后面的3个碱基和荧光基团, 为下一个探针的掺入做好准备. 由于SOLiD采用了"双碱基编码技术", 在测序过程中对每个碱基都检测了两遍, 从而提高了原始数据的质量. SOLiD系统的主要优势在于具有很高的序列读取精确度和数据输出量, 相同数据量的测序价格略低Solexa测序的价格[16,17].
把上述高通量测序技术应用到由mRNA逆转录生成的cDNA上, 从而获得特定样本中不同基因的mRNA片段的含量, 这就是mRNA测序或mRNA-Seq, 同样原理, 各种类型的转录本都可以用深度测序技术进行高通量检测, 统称作RNA-Seq. RNA-Seq技术可以将细胞或组织中的转录本逆转录成cDNA, 随后cDNA文库中的DNA被打成片段, 在cDNA两端加上接头, 开始利用上述高通量测序技术检测. RNA-Seq无需预先设计探针, 即可对任意物种的整体转录活动进行检测, 提供更精确的数字化信号、更高的检测通量以及更广泛的检测范围[18].
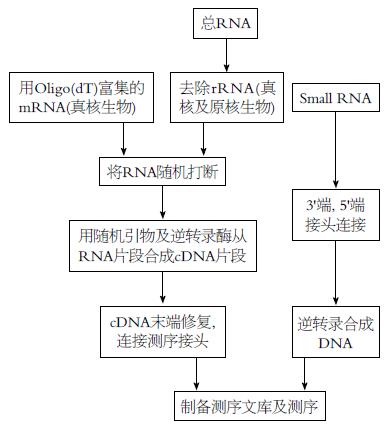
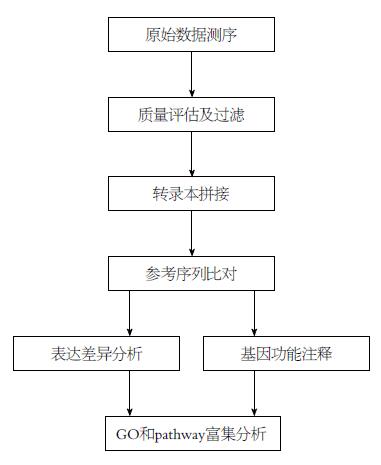
RNA-Seq技术测序后会产生海量数据, 需对数据进行有效比对和分析, 才能有最新科学发现. 以Illumina公司的HiSeq 2000技术流程为例, 全过程包括样品质检、建库、上机测序以及获取最终序列分析数据[19-21]: (1)样品总RNA质检: 利用琼脂糖凝胶电泳观察RNA完整性及是否污染; 精确定量RNA浓度; 利用Agilent 2100精确检测RNA的完整性; (2)构建转录组测序文库: 利用Oligo dT与ploy A进行A-T碱基配对, 可以从总RNA中分离出mRNA, 用于分析转录组信息; 利用金属离子, 将mRNA随机断裂成200 bp左右的小片段, 使mRNA片段化; 在逆转录酶的作用下, 利用随机引物, 以mRNA为模板反转录合成cDNA; 加入End Repair Mix将cDNA补成平末端, 连接adaptor; (3)Illumina Hiseq2500上机测序: PCR扩增15个cycles; 利用2%琼脂糖对目的条带进行回收; 将目的条带定量后, 按数据比例混合上机; cBot上进行Bridge PCR扩增, 生成clusters; 利用Illumina Hiseq2500进行2*125bp测序; (4)具体实验流程图及数据信息分析流程如下(图3, 图4).
目前转录组相关技术主要包括芯片技术、SAGE技术、MPSS技术及最新提出的RNA-Seq技术等. 基因芯片作为目前应用较为广泛的转基因组检测技术, 优点是成本适中, 数据分析软件较多, 方法成熟, 缺点是限用于已知序列, 无法检测新的RNA, 灵敏度有限, 难以检测低丰度的目标、重复序列以及异常转录产物. SAGE技术被用于分析细胞基因表达状态, 优点是在无基因序列信息的条件下, 就能够检测到所有基因的表达水平, 对未知基因的发现, 其中包括低拷贝基因, 具有很大的推动作用. MPSS技术是在一款在SAGE技术的基础进行改进的新技术, 该技术简化了一些测序操作流程, 精确度也得到了很大的提高, 这使得该款测序技术的工作难度得到了加强[22,23].
最新提出的RNA-Seq技术, 可以通过测定细胞全部转录产物的序列以及序列的比对就能得到转录组, 具有独特的优势: (1)数字化信号: 直接测定每个转录本片段序列, 单核苷酸分辨率的精确度高, 可以检测单个碱基差异、基因家族中相似基因以及可变剪接造成的不同转录本的表达, 同时不存在传统微阵列杂交的荧光模拟信号带来的交叉反应和背景噪音问题; (2)高灵敏度: 能够检测到细胞中少至几个拷贝的稀有转录本[24-27]; (3)任意物种的全转录组分析: 无需预先设计特异性探针, 能够直接对任何物种进行转录组分析, 同时能够检测新的转录本, 发现未知基因; (4)更广的检测范围: 高于6个数量级的动态检测范围, 能够同时鉴定和定量稀有转录本和正常转录本[28-30].
随着系统生物学的到来, 转录组学技术得到了不断发展和应用, RNA-Seq技术在医学研究领域得到较为广泛的应用, 尤其在胃肠道肿瘤研究领域, 取得了一些新的进展.
大肠癌(colorectal cancer, CRC)分为结肠癌和直肠癌, 是常见的消化道恶性肿瘤之一. 2014 年全球CRC的预计新发及死亡病例居常见恶性肿瘤的第3位[31]. 《2015年中国癌症生死数据报告》显示, 我国男性CRC发病率和死亡率均位居各型肿瘤第5位, 女性CRC发病率和死亡率分别位居各型肿瘤的第4和第5位[32], CRC已经成为不容忽视的疾病. 近年来通过对CRC标本行转录组学测序, 为结肠癌深入研究提供了高效途径, 尤其是RNA-Seq测序技术已经广泛应用CRC研究, 并取得了一些进展.
以奥沙利铂为基础的化疗方案, 如FOLFOX, 是CRC以及转移性CRC的一线化疗方案, 然而奥沙利铂的耐药性以及周围神经毒性, 迫使我们探究对奥沙利铂敏感的特异生物标志物以提高疗效. RNA-Seq技术已经被广泛应用在探究生物标志物上[33], Li等[34]利用RNA-Seq技术对奥沙利铂特异敏感的21个结肠癌患者血液标本进行检测, 将对奥沙利铂不敏感的基础mRNA表达水平和敏感的mRNA表达水平进行比对分析, 结果显示这21个对奥沙利铂特异敏感的结肠癌病例中有58个相关上调下调基因. 进一步数据分析显示, 当奥沙利铂常规剂量作用时, log2基因突变, 而当奥沙利铂IC50作用剂量时, log10突变, 这对奥沙利铂的特异敏感标志物的进一步探究, 以及对奥沙利铂为基础的联合化疗方案的实施具有指导意义.
以伊立替康为基础的化疗方案, 如FOLFIRE, 为复发转移性CRC的一线或者二线治疗方案. 虽然伊立替康在复发转移性CRC中展现出很多优势, 但是特异敏感性较低以及不良反应较大, 探究伊立替康敏感的特异标志物就成为必然, 以提高伊立替康疗效并减轻副反应. Li等[35]以伊立替康特异敏感的20个结肠癌患者为研究对象, 利用RNA-Seq技术探究对伊立替康敏感或耐受的相关基因的应答. 发现与MYC相互作用的转录调节剂CITED2(Glu/Asp-rich carboxy-terminal domain 2)可作为伊立替康耐药的蛋白标志物[36,37]. 并且发现伊立替康联合铂类在乳腺癌的转移以及结肠癌的侵袭等方面的抗肿瘤作用或许与CITED2的变化密切相关[38,39].
利用RNA-Seq技术探究CRC以及转移性CRC的一线化疗方案中具体化疗药物敏感的特异生物标志物, 对于提高CRC化疗药物的疗效具有重要意义, 为进一步深入RNA-Seq技术在CRC领域的研究具有指导意义.
肝转移是结直肠癌患者的主要死亡原因之一, 早期预测、发现肝转移对提高患者预后有重要意义. 林国生等[40]结合RNA-Seq、GO和KEGG 3种分析显示直肠癌原发灶肿瘤组织中REG基因家族的下调可能是直肠癌异时性肝转移最主要的预警事件之一, REG1A、REG3A、REG1B表达缺失可能是直肠癌异时性肝转移潜在的预测指标. 这是首次应用RNA-Seq技术描述与直肠癌异时性肝转移相关的转录谱改变, 有助于直肠癌异时性肝转移潜在预警因子的发现, 并为直肠癌异时性肝转移的治疗提供潜在治疗靶点.
细胞增殖、细胞分化以及细胞凋亡导致基因突变以及分子机制改变可导致结肠癌的发生, 为了探究CRC特异性表达的基因、突变基因以及融合基因, Liu等[41]从序列读档下载三期结肠癌患者瘤组织、癌旁组织以及远端正常组织相关数据, 通过RNA-Seq技术对数据进行分析处理, 发现一些异常调节的转录因子和细胞凋亡密切相关, 而且Trp53和BCL6与癌组织的错义突变相关联. COL1A1和PDGFB以及COL1A2和PLAG1和基因的融合相关联. 此结果对我们进一步研究结肠癌相关致瘤机制具有指导意义.
据世界卫生组织最新研究显示[42], 全球范围内胃癌新发病例数居恶性肿瘤发病谱第4位. 其中超过70%的胃癌新发病例发生在发展中国家, 约50%发生在中国. 胃癌是我国最常见的恶性肿瘤之一, 尤其在我国西北地区胃癌的发病率及死亡率均处于较高水平[43,44], 我国每年新发胃癌病例40万, 占世界总发病例数的42%. 胃癌的发生是一个多阶段、多因素的复杂致病过程[45-47], 严重威胁人类健康. 在胃癌的分子生物学领域采用的研究手段很多, 但是仍缺乏快速、准确、有效的方法. 近些年来, RNA-Seq技术在胃癌的分子机制、基因突变以及融合基因的致瘤机制等方面取得了突出进展.
为了研究胃癌的分子机制, Yon等[48]利用RNA-Seq技术研究胃癌组织以及正常组织, 得到了显示胃癌全转录组特点的6.8亿短读信息(包括mRNAs和microRNAs), 利用多层(包括全转录组、小RNA-Seq图书馆、SOLiD序列)分析证实各种转录突变和不同阶段胃癌的相关关系, 包括特异性mRNAs的表达, 复合体的突变以及microRNAs的特异性表达. 最后发现中心代谢调控因子AMPK-α也许是胃癌发生发展的功能性靶蛋白, 这为早期胃癌的靶蛋白治疗提供依据, 在一定程度上诠释了胃癌的发生发展机制.
虽然胃癌的发生率很高, 其中基因突变在胃癌的发生机制中占据重要地位, 但是具体机制尚未完全清楚. Liu等[49]利用全外显子序列技术、RNA-Seq技术、单核苷酸多态矩阵技术发现了相对正常组织胃癌组织中ZAK激酶同工酶TV1表达上调. 如果胃癌中TV1过表达, 许多肿瘤相关的全转录组因子受体将被激活, 相反, 如果TV1表达下调, 将抑制肿瘤细胞的增殖, 这是首次利用RNA-Seq技术充分显示了ZAK激酶同工酶TV1的致瘤作用. 卫星不稳定因子引起基因突变是胃癌形成的一个重要因素. Yoon等[50]利用RNA-Seq技术、全基因组序列和高通量的RNA序列证实胃癌重复编码区和非翻译区中有18377个微卫星(microsatellite, MS)突变, 其中有139个特异表达下调的基因和非翻译区MS的突变相关联. 有90.5%的MS突变发生在非翻译区, 揭示了卫星不稳定性在胃癌的重要作用, 这对胃癌的临床治疗具有指导意义.
基因融合是恶性肿瘤的重要致病因素, 融合基因是由两个相邻基因的全部或一部分序列相互融合, 通过基因间剪接从而形成的一种新的转录物. 随着第二代测序技术的发展, 在这一领域的研究成为热点. 为了探究胃癌基因融合的相关机制, Kim等[51]用高效探测基因融合的全转录组测序技术, 对23个融合本包括嵌合体RNAs和融合基因进行研究, 发现融合基因包括细胞生长因子受体、周期依赖性蛋白受体和成纤维生长因子受体2, 均可作为药物作用的重要靶点. 同时发现转录通读的二氢尿嘧啶激酶4、B细胞受体相关蛋白29有致瘤作用. 通过RNA-Seq技术发现了很多胃癌相关的基因融合本, 提高了我们对胃癌致瘤中相关融合基因的认识.
目前关于RNA-Seq在胃肠肿瘤领域的应用, 主要集中在化疗药物敏感的特异生物标志物、胃肠肿瘤发生发展的分子机制、特异表达的基因、突变基因以及融合基因等, 使得对胃肠肿瘤特定化疗药物敏感的分子标志物越来越多被发现, 从而在一定程度上提高了疗效, 并且越来越多的突变基因被发现和胃肠肿瘤的发生机制有关, 特异基因的上调和下调可作为胃肠肿瘤转移倾向的标志.
胃肠肿瘤从本质上来说是一种遗传病, 并且基因组体细胞水平分子改变的积累是肿瘤演进过程的基础. RNA-Seq技术是对已知基因组序列的物种进行不同个体的基因组测序, 并在此基础上对个体或群体进行差异性分析, 其优势在于能够通过新一代高通量测序及生物信息学比对, 快速准确地对肿瘤基因组及转录组进行测序分析, 找到与肿瘤发生发展密切相关的变异及改变, 从而为快速定位肿瘤分子靶标及完善肿瘤个体化医疗带来曙光. 基因芯片技术作为另一种在胃肠道肿瘤分子生物学领域应用较为广泛的技术, 虽然同样具有高通量、快速、灵敏等特点, 但该技术检测的是芯片上已有的基因, 而RNA-Seq技术可以全谱筛查胃肠肿瘤转录基因, 能够从整体水平研究基因功能以及基因结构, 并能很好地显示处于表达状态的基因数量和活跃程度. 在胃肠肿瘤的基础研究领域RNA-Seq技术重复性好, 起始样品比芯片技术要少得多, 尤其适用于来源极为有限的生物样品分析, 如胃肠肿瘤干细胞研究等.
RNA-Seq技术作为刚起步的技术, 已经以惊人的速度应用于各种生物组织研究, 极大的促进肿瘤研究的深入. 虽然RNA-Seq技术本身已经显示出其他分析技术无可比拟的优势, 但RNA-Seq技术仍然存在问题, 测序虽然得到了大量的数据, 但基于有限的生物信息学分析软件, 经常导致研究者对所要分析的信息得不到很好的结果, 造成测序数据的浪费. 因此建立系统、完善的信息分析数据库, 减少实验数据及资源浪费是我们面临的重大挑战, 同时需不断改进测序技术和降低测序费用, 相信随着RNA-Seq技术不断发展, 其将为医学深入研究提供便捷.
近几年RNA-Seq测序技术虽然在胃肠肿瘤研究领域有关化疗药物敏感的特异生物标志物、胃肠肿瘤发生发展的分子机制、特异表达的基因、突变基因以及融合基因等方面取得进展, 但研究尚还不够全面和深入, 肿瘤的发生发展, 除了跟基因突变、转录异常有关外, 还涉及翻译、表观遗传学等方面的改变, 因此还需要各类数据的有效整合. 相信随着分子生物学技术的发展和测序技术的逐步成熟, RNA-Seq技术在胃肠道肿瘤的相关分子机制以及药物研发中将得到更广泛的应用.
随着后基因组时代的来临, 转录组学技术作为率先发展起来的新技术已被广泛应用. 转录组学新一代高通量测序技术, 如基因芯片、RNA-Seq等, 已经广泛应用于医学研究领域, 并取得了一定的进展.
近几年RNA-Seq测序技术虽然使越来越多的胃肠道相关致癌基因被发现, 但肿瘤的发生发展, 除了跟基因突变、转录异常有关外, 发病还涉及翻译、表观遗传学等方面的改变, 因此还需要各类数据的有效整合. 相信随着分子生物学技术的发展和测序技术的逐步成熟, RNA-Seq在胃肠道肿瘤的相关分子机制以及药物研发中将得到更广泛的应用.
陈竺认为已经到了系统生物学时代, 林国生等结合RNA-Seq、GO和KEGG 3种分析显示直肠癌原发灶肿瘤组织中REG基因家族的下调可能是直肠癌异时性肝转移最主要的预警事件之一, REG1A、REG3A、REG1B表达缺失可能是直肠癌异时性肝转移潜在的预测指标.
利用RNA-Seq技术探究大肠癌(colorectal cancer, CRC)以及转移性CRC的一线化疗方案中具体化疗药物敏感的特异生物标志物, 并首次应用RNA-Seq技术描述与直肠癌异时性肝转移相关的转录谱改变以及CRC致瘤机制. RNA-Seq技术在胃癌的分子机制、基因突变以及融合基因的致瘤机制等方面取得了一些新的研究进展.
本文就RNA-Seq技术原理、优势及其在胃肠肿瘤研究中的具体应用进行论述, 为进一步RNA-Seq技术在胃肠道肿瘤的相关分子机制以及药物研发研究中打下基础.
李忠武, 副教授, 副主任医师, 北京大学肿瘤医院病理科; 杨秋蒙, 副主任医师, 上海交通大学医学院附属瑞金医院; 周春祥, 教授, 南京中医药大学基础医学院伤寒教研室
该文行文流畅, 主题明确, 立意新颖, 具有一定的学术指导价值.
手稿来源: 自由投稿
学科分类: 胃肠病学和肝病学
手稿来源地: 安徽省
同行评议报告分类
A级 (优秀): 0
B级 (非常好): 0
C级 (良好): C, C, C
D级 (一般): 0
E级 (差): 0
编辑:马亚娟 电编:李瑞芳
| 1. | Bawa P, Zackaria S, Verma M, Gupta S, Srivatsan R, Chaudhary B, Srinivasan S. Integrative Analysis of Normal Long Intergenic Non-Coding RNAs in Prostate Cancer. PLoS One. 2015;10:e0122143. [PubMed] [DOI] |
| 2. | Ament SA, Pearl JR, Grindeland A, St Claire J, Earls JC, Kovalenko M, Gillis T, Mysore J, Gusella JF, Lee JM. High resolution time-course mapping of early transcriptomic, molecular and cellular phenotypes in Huntington's disease CAG knock-in mice across multiple genetic backgrounds. Hum Mol Genet. 2017;26:913-922. [PubMed] [DOI] |
| 3. | Mills JD, Janitz M. Alternative splicing of mRNA in the molecular pathology of neurodegenerative diseases. Neurobiol Aging. 2012;33:1012.e11-1012.e24. [PubMed] [DOI] |
| 4. | Li M, Xie X, Zhou J, Sheng M, Yin X, Ko EA, Zhou T, Gu W. Quantifying circular RNA expression from RNA-seq data using model-based framework. Bioinformatics. 2017; Mar 8 [Epub ahead of print]. [PubMed] [DOI] |
| 5. | Tsai KY. The preneoplastic genome: transcriptomic drivers of squamous cell carcinoma development. Dermatol Online J. 2016;22:57-68. |
| 6. | Eirin A, Zhu XY, Puranik AS, Woollard JR, Tang H, Dasari S, Lerman A, van Wijnen AJ, Lerman LO. Integrated transcriptomic and proteomic analysis of the molecular cargo of extracellular vesicles derived from porcine adipose tissue-derived mesenchymal stem cells. PLoS One. 2017;12:e0174303. [PubMed] [DOI] |
| 9. | 林 燕敏, 门 振华, 陈 业强, 兰 文军. 基因测序技术发展及生物医学应用. 齐鲁工业大学学报(自然科学版). 2016;30:24-28. |
| 10. | Huang W, Goldfischer M, Babyeva S, Mao Y, Volyanskyy K, Dimitrova N, Fallon JT, Zhong M. Identification of a novel PARP14-TFE3 gene fusion from 10-year-old FFPE tissue by RNA-seq. Genes Chromosomes Cancer. 2015; May 29 [Epub ahead of print]. [PubMed] [DOI] |
| 11. | Mardis ER. The impact of next-generation sequencing technology on genetics. Trends Genet. 2008;24:133-141. [PubMed] [DOI] |
| 13. | Margulies M, Egholm M, Altman WE, Attiya S, Bader JS, Bemben LA, Berka J, Braverman MS, Chen YJ, Chen Z. Genome sequencing in microfabricated high-density picolitre reactors. Nature. 2005;437:376-380. [PubMed] [DOI] |
| 14. | Smith DR, Quinlan AR, Peckham HE, Makowsky K, Tao W, Woolf B, Shen L, Donahue WF, Tusneem N, Stromberg MP. Rapid whole-genome mutational profiling using next-generation sequencing technologies. Genome Res. 2008;18:1638-1642. [PubMed] [DOI] |
| 16. | 陈 子江, 颜 军昊. 迎接新一代测序时代的到来. 中国实用妇科与产科杂志. 2016;32:13-15. |
| 18. | 王 兴春, 谭 河林, 陈 钊, 孟 令芝, 王 文斌, 范 圣此. 基于RNA-Seq技术的连翘转录组组装与分析及SSR分子标记的开发. 中国科学: 生命科学. 2015;45:301-310. |
| 19. | Cock PJ, Fields CJ, Goto N, Heuer ML, Rice PM. The Sanger FASTQ file format for sequences with quality scores, and the Solexa/Illumina FASTQ variants. Nucleic Acids Res. 2010;38:1767-1771. [PubMed] [DOI] |
| 20. | Erlich Y, Mitra PP, delaBastide M, McCombie WR, Hannon GJ. Alta-Cyclic: a self-optimizing base caller for next-generation sequencing. Nat Methods. 2008;5:679-682. [PubMed] [DOI] |
| 21. | Grabherr MG, Haas BJ, Yassour M, Levin JZ, Thompson DA, Amit I, Adiconis X, Fan L, Raychowdhury R, Zeng Q. Full-length transcriptome assembly from RNA-Seq data without a reference genome. Nat Biotechnol. 2011;29:644-652. [PubMed] [DOI] |
| 22. | Okoniewski MJ, Miller CJ. Hybridization interactions between probesets in short oligo microarrays lead to spurious correlations. BMC Bioinformatics. 2006;7:276. [PubMed] [DOI] |
| 23. | Royce TE, Rozowsky JS, Gerstein MB. Toward a universal microarray: prediction of gene expression through nearest-neighbor probe sequence identification. Nucleic Acids Res. 2007;35:e99. [PubMed] [DOI] |
| 24. | Tariq MA, Kim HJ, Jejelowo O, Pourmand N. Whole-transcriptome RNAseq analysis from minute amount of total RNA. Nucleic Acids Res. 2011;39:e120. [PubMed] [DOI] |
| 25. | Richard H, Schulz MH, Sultan M, Nürnberger A, Schrinner S, Balzereit D, Dagand E, Rasche A, Lehrach H, Vingron M. Prediction of alternative isoforms from exon expression levels in RNA-Seq experiments. Nucleic Acids Res. 2010;38:e112. [PubMed] [DOI] |
| 26. | Lee MC, Lopez-Diaz FJ, Khan SY, Tariq MA, Dayn Y, Vaske CJ, Radenbaugh AJ, Kim HJ, Emerson BM, Pourmand N. Single-cell analyses of transcriptional heterogeneity during drug tolerance transition in cancer cells by RNA sequencing. Proc Natl Acad Sci USA. 2014;111:E4726-E4735. [PubMed] [DOI] |
| 27. | Ding X, Zhu L, Ji T, Zhang X, Wang F, Gan S, Zhao M, Yang H. Long intergenic non-coding RNAs (LincRNAs) identified by RNA-seq in breast cancer. PLoS One. 2014;9:e103270. [PubMed] [DOI] |
| 28. | Iyer MK, Niknafs YS, Malik R, Singhal U, Sahu A, Hosono Y, Barrette TR, Prensner JR, Evans JR, Zhao S. The landscape of long noncoding RNAs in the human transcriptome. Nat Genet. 2015;47:199-208. [PubMed] [DOI] |
| 29. | Jiang YZ, Li QH, Zhao JQ, Lv JJ. Identification of a novel fusion gene (HLA-E and HLA-B) by RNA-seq analysis in esophageal squamous cell carcinoma. Asian Pac J Cancer Prev. 2014;15:2309-2312. [PubMed] [DOI] |
| 30. | Qian X, Ba Y, Zhuang Q, Zhong G. RNA-Seq technology and its application in fish transcriptomics. OMICS. 2014;18:98-110. [PubMed] [DOI] |
| 31. | Jemal A, Bray F, Center MM, Ferlay J, Ward E, Forman D. Global cancer statistics. CA Cancer J Clin. 2011;61:69-90. [PubMed] [DOI] |
| 32. | Chen W, Zheng R, Baade PD, Zhang S, Zeng H, Bray F, Jemal A, Yu XQ, He J. Cancer statistics in China, 2015. CA Cancer J Clin. 2016;66:115-132. [PubMed] [DOI] |
| 33. | Lin KT, Shann YJ, Chau GY, Hsu CN, Huang CY. Identification of latent biomarkers in hepatocellular carcinoma by ultra-deep whole-transcriptome sequencing. Oncogene. 2016;35:5078. [PubMed] [DOI] |
| 34. | Li XX, Peng JJ, Liang L, Huang LY, Li DW, Shi DB, Zheng HT, Cai SJ. RNA-seq identifies determinants of oxaliplatin sensitivity in colorectal cancer cell lines. Int J Clin Exp Pathol. 2014;7:3763-3770. [PubMed] |
| 35. | Li XX, Zheng HT, Peng JJ, Huang LY, Shi DB, Liang L, Cai SJ. RNA-seq reveals determinants for irinotecan sensitivity/resistance in colorectal cancer cell lines. Int J Clin Exp Pathol. 2014;7:2729-2736. [PubMed] |
| 36. | Wu ZZ, Sun NK, Chao CC. Knockdown of CITED2 using short-hairpin RNA sensitizes cancer cells to cisplatin through stabilization of p53 and enhancement of p53-dependent apoptosis. J Cell Physiol. 2011;226:2415-2428. [PubMed] [DOI] |
| 37. | Wu ZZ, Lu HP, Chao CC. Identification and functional analysis of genes which confer resistance to cisplatin in tumor cells. Biochem Pharmacol. 2010;80:262-276. [PubMed] [DOI] |
| 38. | van Agthoven T, Sieuwerts AM, Veldscholte J, Meijer-van Gelder ME, Smid M, Brinkman A, den Dekker AT, Leroy IM, van Ijcken WF, Sleijfer S. CITED2 and NCOR2 in anti-oestrogen resistance and progression of breast cancer. Br J Cancer. 2009;101:1824-1832. [PubMed] [DOI] |
| 39. | Bai L, Merchant JL. A role for CITED2, a CBP/p300 interacting protein, in colon cancer cell invasion. FEBS Lett. 2007;581:5904-5910. [PubMed] [DOI] |
| 40. | Lin GS, ZhouYJ . Investigation of warning genes involved in hepatic metastasis of rectal carcinoma based on RNA-Seq. Fujian: Fujian Medical University 2014; 1-16. |
| 41. | Liu F, Ji F, Ji Y, Jiang Y, Sun X, Lu Y, Zhang L, Han Y, Liu X. Dissecting the mechanism of colorectal tumorigenesis based on RNA-sequencing data. Exp Mol Pathol. 2015;98:246-253. [PubMed] [DOI] |
| 43. | Dong Y, Ma S, Yang S, Luo F, Wang Z, Guo F. Non-curative surgery for patients with gastric cancer with local peritoneal metastasis: A retrospective cohort study. Medicine (Baltimore). 2016;95:e5607. [PubMed] [DOI] |
| 44. | Liu Z, Feng F, Guo M, Liu S, Zheng G, Xu G, Lian X, Fan D, Zhang H. Distal gastrectomy versus total gastrectomy for distal gastric cancer. Medicine (Baltimore). 2017;96:e6003. [PubMed] [DOI] |
| 45. | Ferlay J, Shin HR, Bray F, Forman D, Mathers C, Parkin DM. Estimates of worldwide burden of cancer in 2008: GLOBOCAN 2008. Int J Cancer. 2010;127:2893-2917. [PubMed] [DOI] |
| 46. | Wadhwa R, Song S, Lee JS, Yao Y, Wei Q, Ajani JA. Gastric cancer-molecular and clinical dimensions. Nat Rev Clin Oncol. 2013;10:643-655. [PubMed] [DOI] |
| 47. | Akhavan-Niaki H, Samadani AA. Molecular insight in gastric cancer induction: an overview of cancer stemness genes. Cell Biochem Biophys. 2014;68:463-473. [PubMed] [DOI] |
| 48. | Kim YH, Liang H, Liu X, Lee JS, Cho JY, Cheong JH, Kim H, Li M, Downey TJ, Dyer MD. AMPKα modulation in cancer progression: multilayer integrative analysis of the whole transcriptome in Asian gastric cancer. Cancer Res. 2012;72:2512-2521. [PubMed] [DOI] |
| 49. | Liu J, McCleland M, Stawiski EW, Gnad F, Mayba O, Haverty PM, Durinck S, Chen YJ, Klijn C, Jhunjhunwala S. Integrated exome and transcriptome sequencing reveals ZAK isoform usage in gastric cancer. Nat Commun. 2014;5:3830. [PubMed] [DOI] |
| 50. | Yoon K, Lee S, Han TS, Moon SY, Yun SM, Kong SH, Jho S, Choe J, Yu J, Lee HJ. Comprehensive genome- and transcriptome-wide analyses of mutations associated with microsatellite instability in Korean gastric cancers. Genome Res. 2013;23:1109-1117. [PubMed] [DOI] |