修回日期: 2019-12-10
接受日期: 2019-12-20
在线出版日期: 2019-12-28
肝细胞癌(hepatocellular carcinoma, HCC)是消化系统常见恶性肿瘤, 预后较差. 本文拟采用生物信息学方法探讨CCNA2基因在HCC中的表达情况, 及其作为HCC患者预后分子标志物的可行性. 同时, 采用免疫组织化学法对CCNA2生物信息分析结果进行验证.
探讨CCNA2在HCC中的表达, 相关信号通路及与患者预后关系.
应用生物信息分析工具比对TCGA数据库中CCNA2基因mRNA在HCC组织和癌旁组织中表达水平; 在STRING数据库中构建CCNA2蛋白-蛋白相互作用网络, 并对相关蛋白功能和KEGG信号通路进行富集. 根据CCNA2表达水平分为高低表达组, 比较CCNA2高低组患者总生存(overall survival, OS)和无疾病进展生存(disease free survival, DFS)是否不同. 回顾性分析72例手术治疗的HCC患者, 采用免疫组织化学法检测患者癌组织中CCNA2蛋白表达水平与患者临床病理特征的关系, 对生物信息分析结果进行验证.
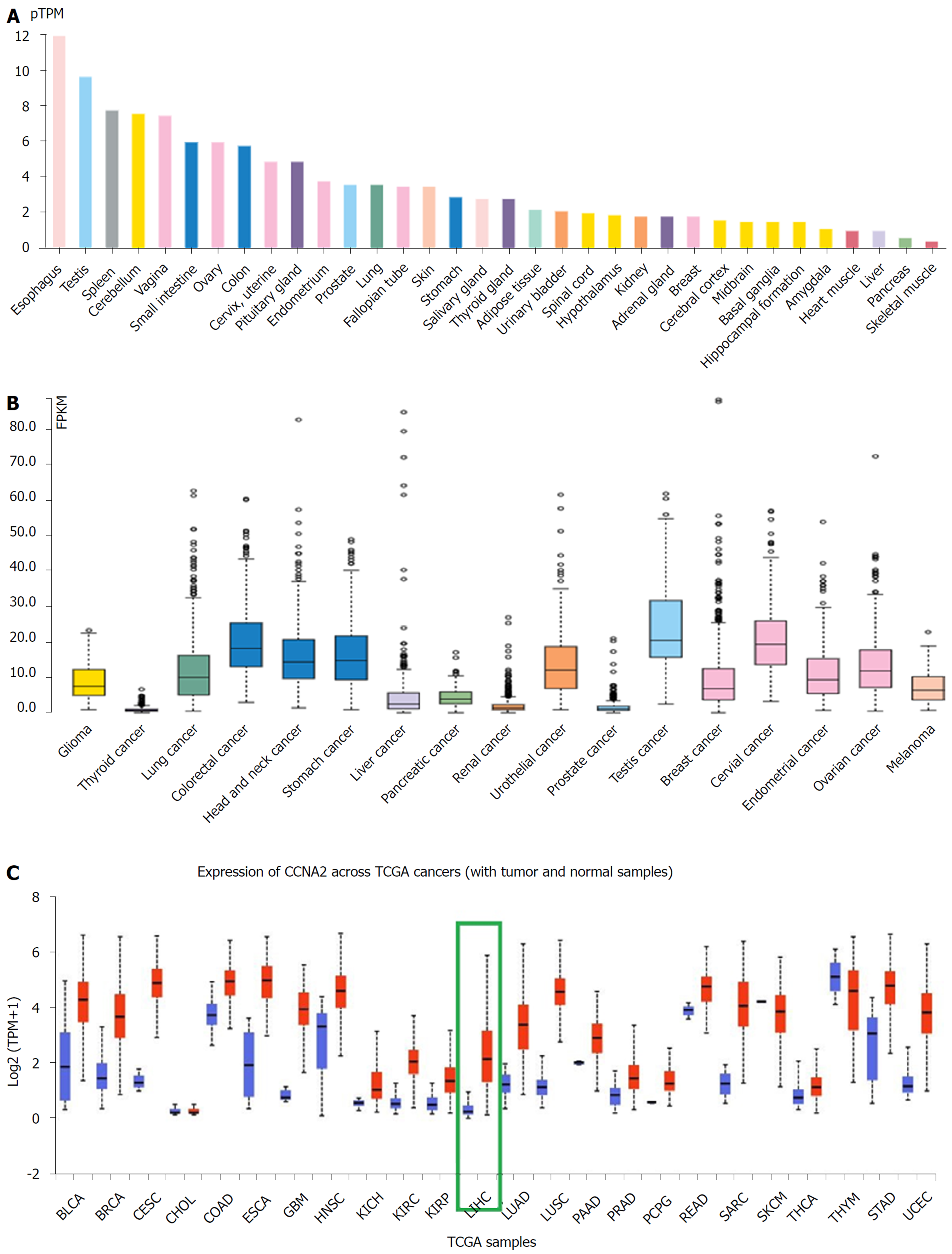
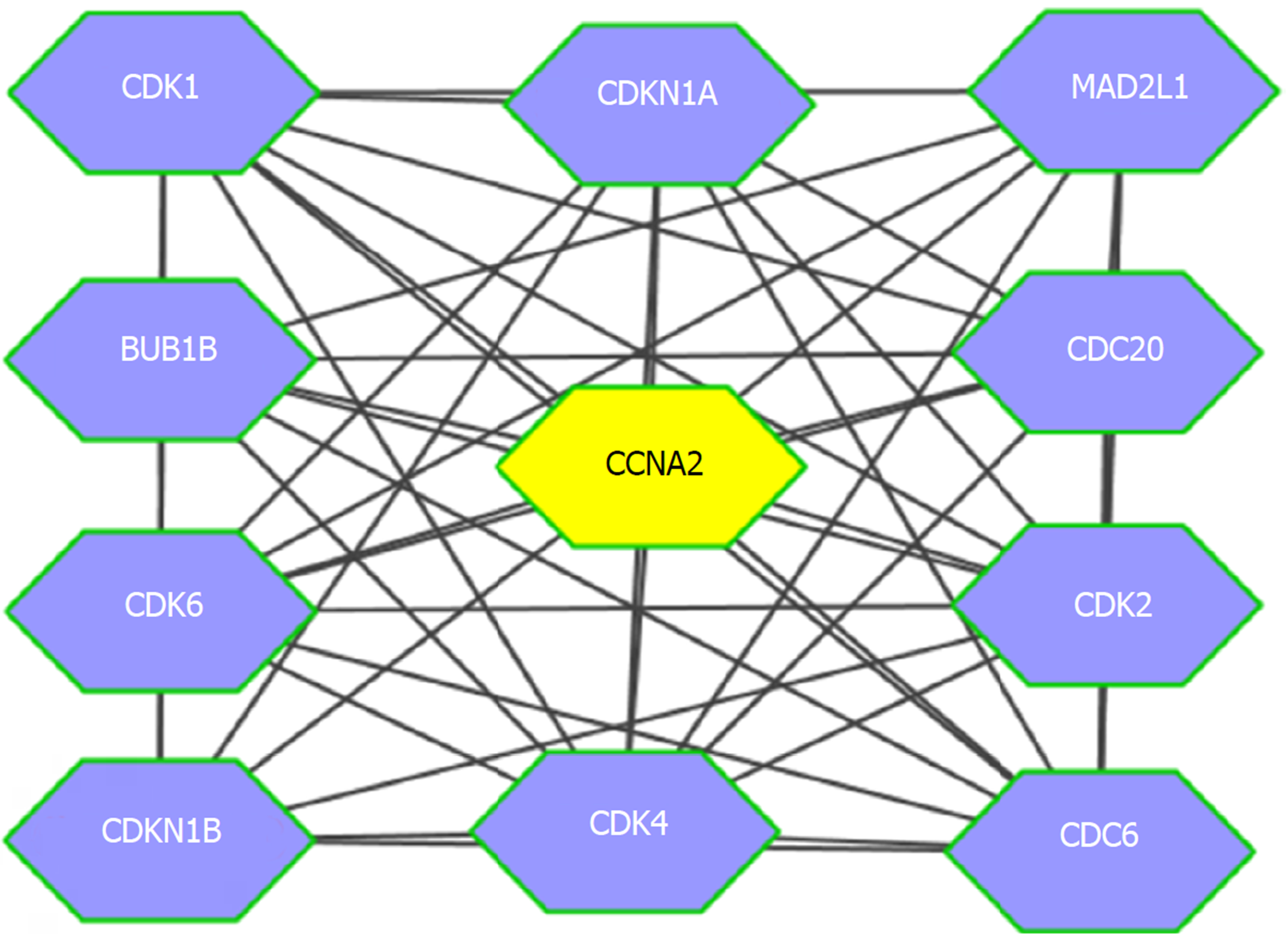
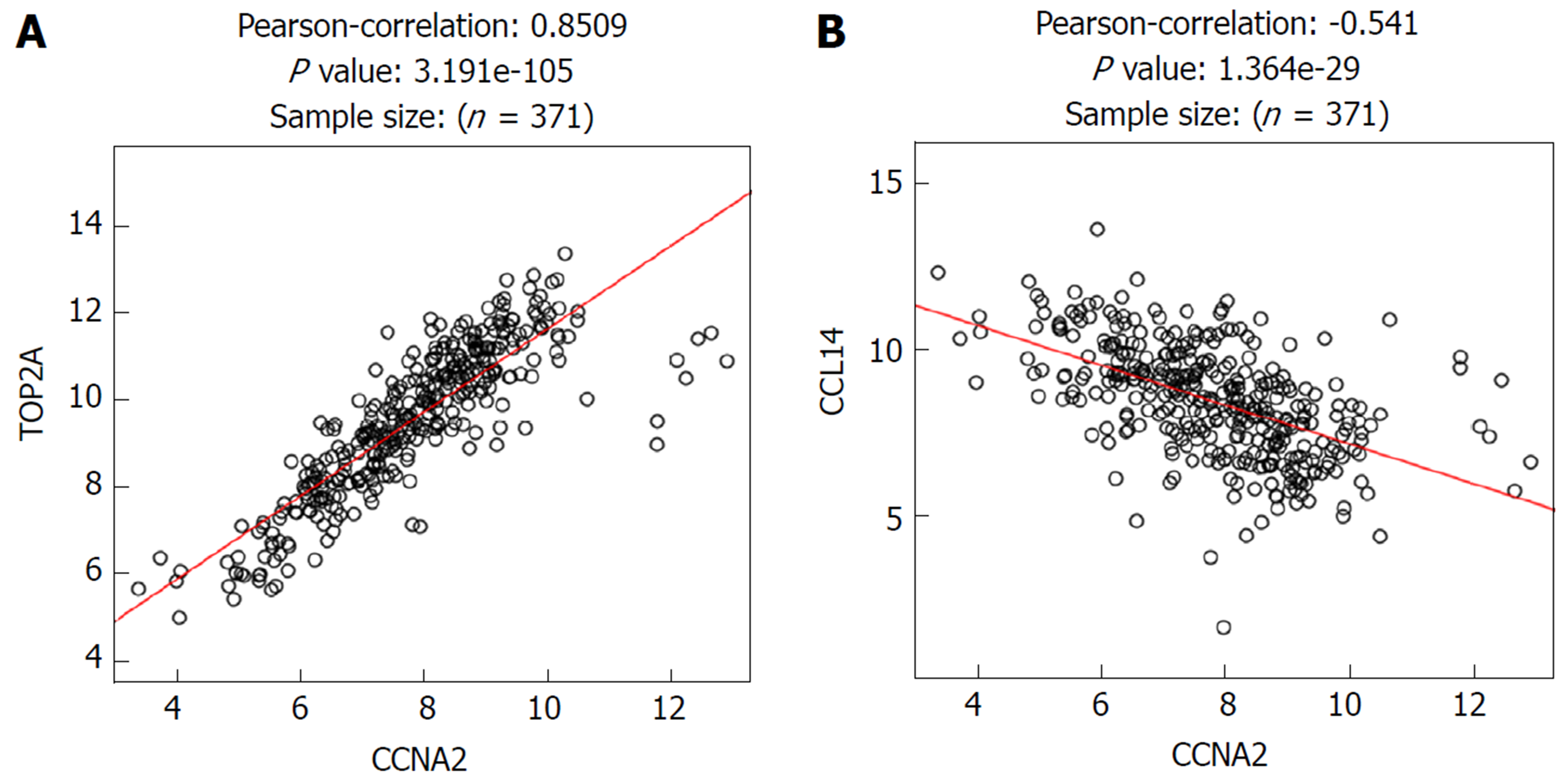
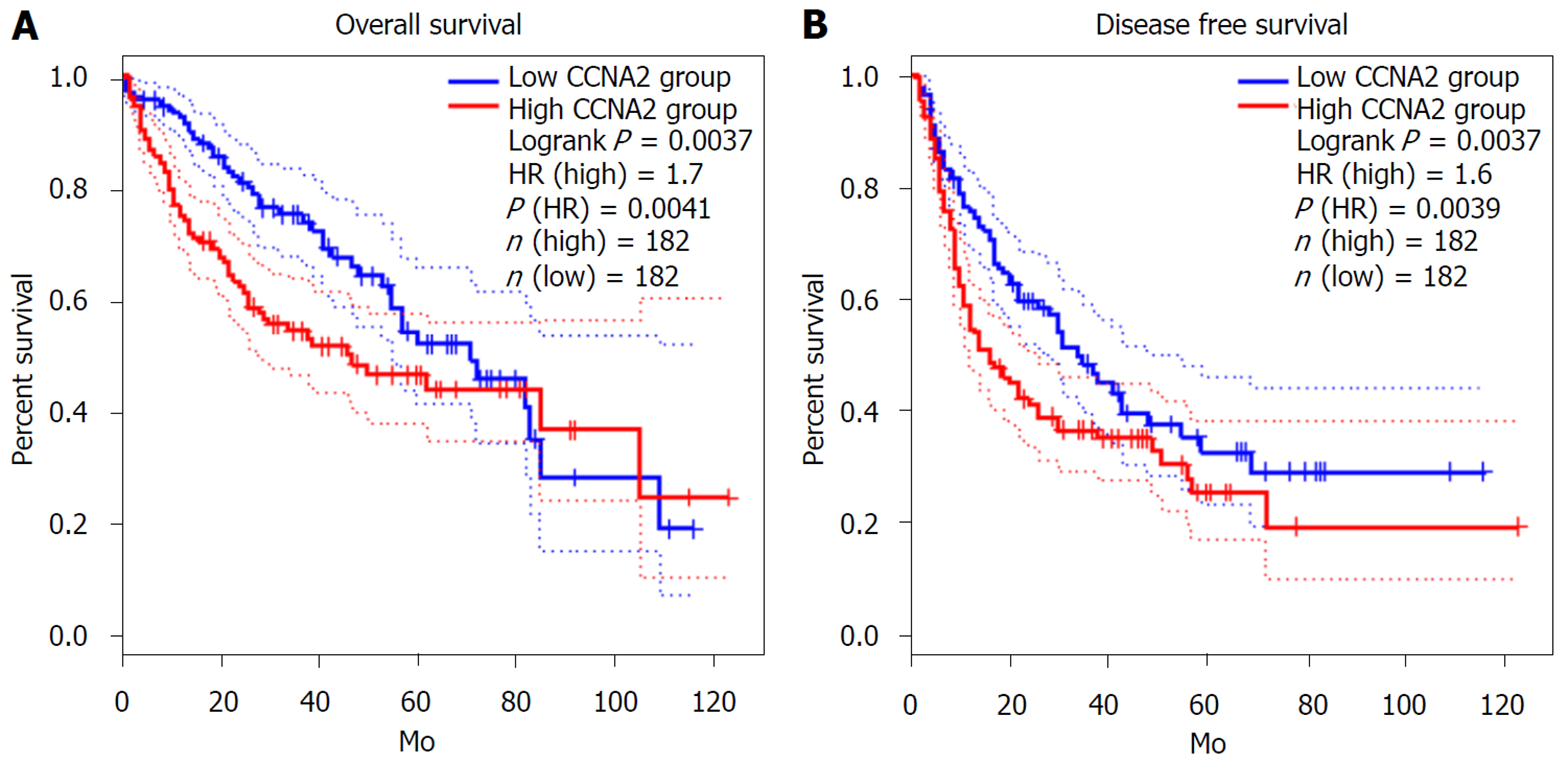
在HCC中, CCNA2中mRNA表达水平明显高于癌旁正常肝组织; 与CCNA2蛋白相互作用较为紧密的蛋白10个, 蛋白间相互作用关系edge = 50, 区域聚类指数为0.931, 与CCNA2蛋白相互作用较为紧密的10蛋白相互作用网络富集显著(P<0.05); TOP2基因mRNA与CCNA2正向相关表达(r = 0.85, P<0.05), 而CCL14基因与CCNA2负相关表达(r = -0.54, P<0.05). CCNA2基因相关信号通路主要富集于细胞周期、病毒致癌、乙型肝炎、p53信号通路和PI3K-Akt信号通路等. 预后分析提示CCNA2基因mRNA高表达患者OS(HR = 1.7, P = 0.0037)和DFS低于低表达组(HR = 1.6, P = 0.0037). 免疫组化显示CCNA2蛋白在HCC患者癌组织中的高表达率为34.7%(25/72). CCNA2蛋白高表达与HCC患者肿瘤直径(P<0.05)、DC浸润(P<0.05)、术后2年复发/转移(P<0.05)有关.
CCNA2基因在HCC患者癌组织中表达水平上调, 并可作为HCC预后不良的分子标记物.
核心提要:CCNA2基因在肝细胞癌(hepatocellular carcinoma, HCC)患者癌组织中表达水平显著高于癌旁正常组织, CCNA2蛋白高表达与HCC患者肿瘤直径(P<0.05)、DC浸润(P<0.05)、术后2年复发/转移(P<0.05)和预后不良有关. CCNA2高表达可作为HCC预后不良的分子标志物.
引文著录: 杨结, 刘树业, 刘运德. CCNA2基因在肝细胞癌中的表达、信号通路和预后关系生物信息分析及验证. 世界华人消化杂志 2019; 27(24): 1490-1501
Revised: December 10, 2019
Accepted: December 20, 2019
Published online: December 28, 2019
Hepatocellular carcinoma (HCC) is one of the most diagnosed malignant carcinomas of the digestive system with a poor prognosis. In this study, the expression of CCNA2 gene expression in HCC was investigated by bioinformatics analysis and the feasibility of CCNA2 gene as a molecular marker for prognosis in HCC was assessed. The results of bioinformatics analysis were verified by immunohistochemistry assay.
To investigate the expression of CCNA2 in HCC, related signal pathways, and its relationship with prognosis.
The expression levels of CCNA2 mRNA in HCC and paracancerous tissues were compared based on the TCCA database. CCNA2 protein interaction network was constructed based on the String database, and the related protein function and KEGG signal pathways were enriched. According to the expression level of CCNA2, the patients were divided into high and low expression groups, and the difference in overall survival (OS) and disease free survival (DFS) was compared between the two groups. The relationship between the expression of CCNA2 protein analyzed by immunohistochemistry and the clinicopathological features of 72 patients with HCC who underwent surgical treatment was analyzed.
The expression level of CCNA2 mRNA was significantly higher in HCC than in normal liver tissues. There are ten proteins that have a close interaction with CCNA2 protein, with edge = 50, and the regional clustering index is 0.931. The interaction network of the ten proteins that have a close interaction with CCNA2 protein was significantly enriched (P < 0.05). The expression of TOP2 mRNA was positively correlated with CCNA2 expression (r = 0.85, P < 0.05), while CCL14 expression was negatively correlated with CCNA2 expression (r = -0.54, P < 0.05). CCNA2 gene related signaling pathways are mainly enriched in cell cycle, viral carcinogenesis, hepatitis B, p53 signaling pathway, and PI3K-Akt signaling pathway. The prognosis analysis indicated that the OS (HR = 1.7, P = 0.0037) and DFS rates were significantly lower in the high expression group than in the low expression group (HR = 1.6, P = 0.0037). Immunohistochemistry showed that the high expression rate of CCNA2 in HCC was 34.7% (25/72). High expression of CCNA2 protein was significantly associated with tumor diameter (P < 0.05), DC infiltration (P < 0.05), and recurrence/metastasis 2 years after operation (P < 0.05).
CCNA2 gene is up-regulated in HCC, which can be used as a molecular marker for poor prognosis in HCC.
- Citation: Yang J, Liu SY, Liu YD. Integrated bioinformatics analysis of expression, related signal pathways, and prognostic significance of CCNA2 in hepatocellular carcinoma. Shijie Huaren Xiaohua Zazhi 2019; 27(24): 1490-1501
- URL: https://www.wjgnet.com/1009-3079/full/v27/i24/1490.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v27.i24.1490
肝细胞癌(hepatocellular carcinoma, HCC)是最常见的原发性肝癌[1]. HCC在欧美等发达国家高加索人种中发病率较低, 但在东南亚, 尤其是我国发病率较高[2]. 虽然年轻人群甚至儿童肝癌临床中偶尔可见, 但HCC主要影响50岁以上的男性. 肝也是胆管癌(肝内胆管癌)的主要部位, 胆管癌相对少见, 占所有原发性肝癌的10%-20%. 目前HCC的危险因素较为明确, 包括: 乙肝、丙肝病毒感染, 肝硬化、黄曲霉毒素暴露和先天性代谢异常如α-1抗胰蛋白酶缺乏症等[3-6]. HCC的一个共同的特点是肝纤维化和炎症的存在, 低分化的肿瘤表现出明显的多形性, 巨细胞与正常肝细胞几乎没有相似之处.
已有研究显示CCNA2基因在多种肿瘤中呈现高表达[7,8], 而CNNA2在HCC的研究未见报道. 本研究首先采用生物信息学分析CNNA2在HCC中的表达及相关信号通路. 同时, 采用免疫组织化学法检测72例HCC患者癌组织中CNNA2的表达及其与HCC患者预后的关系对生物信息分析结果进行验证.
1.1.1 CCNA2表达水平分析: 检索TCGA数据库, 对比各组织及实体肿瘤组织中CCNA2基因的表达水平. 检索分析条件为"肝细胞癌", "肝癌", "CCNA2", 物种为人类. 同时比较CCNA2基因在HCC患者癌组织和癌旁组织中是否存在差异. 存在差异表达的条件为CCNA2基因mRNA上调或下调表达超过2倍(|Log2FC|>1), 且P<0.05[9].
1.1.2 CCNA2蛋白-蛋白相互作用网络: 应用STRING数据库构建CCNA2蛋白相互作用的蛋白-蛋白网络, CCNA2蛋白-蛋白相互作用网络构建条件为置信度大于0.7, 相互作用来源为共表达、基因功能和比邻关系[10].
1.1.3 CCNA2基因共表达及生存分析: 在TCGA数据库中, 依据与CCNA2基因共表达关系, 对与CCNA2基因存在相关性的基因进行聚类. 同时选取正相关和负相关表达最为明显的2个基因进行分析, 计算spearman相关系数; 根据CCNA2基因mRNA在HCC患者癌组织中表达的中位数, 分为高低表达组, 绘制生存分析曲线, 并进行Log-rank检验比较高低表达组患者总生存(overall survival, OS)和无疾病进展生存(disease free survival, DFS)是否存在差异.
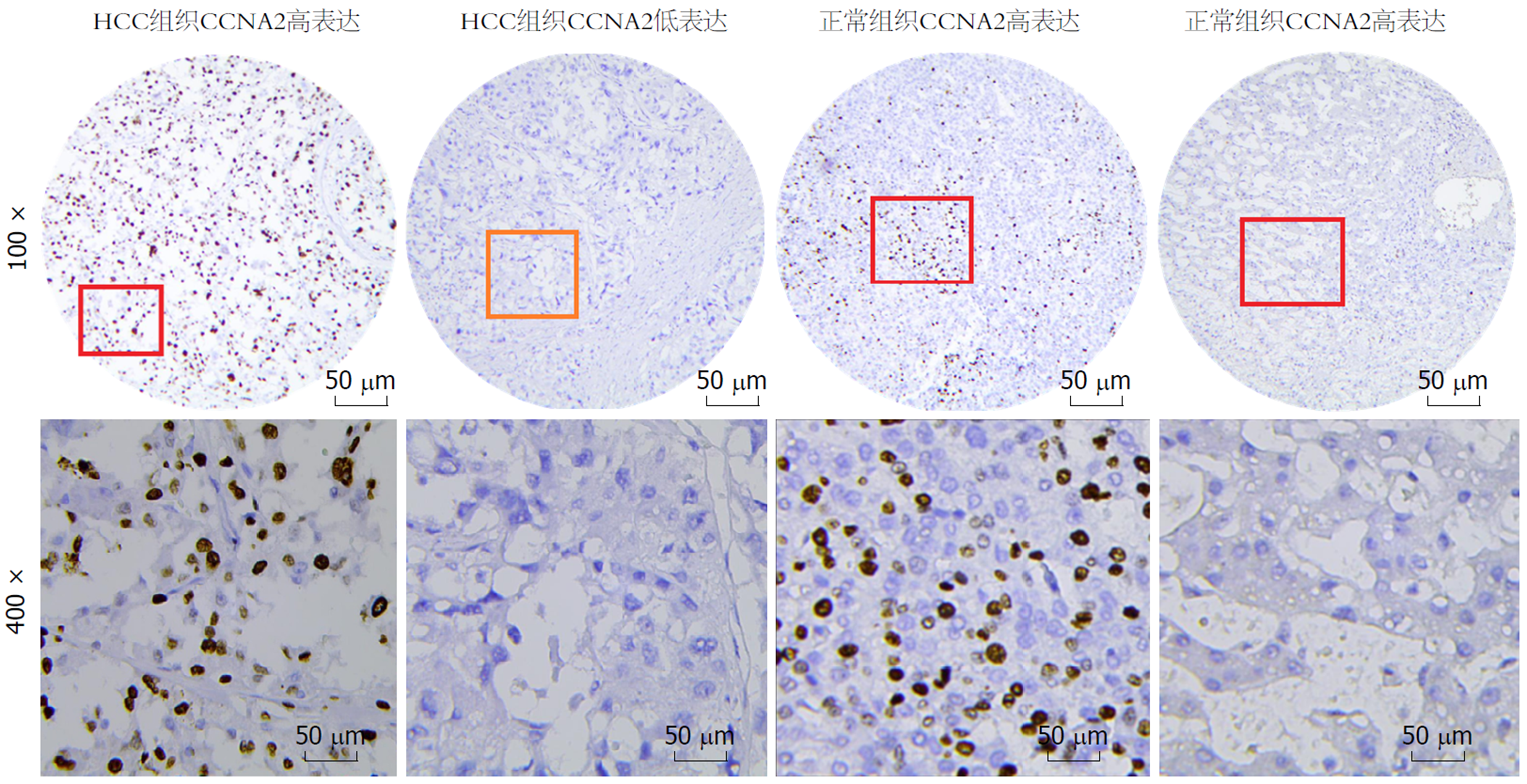
免疫组化检测CCNA2蛋白表达验证生物信息结果: 同时回顾性分析我院收治手术之类的HCC患者72例, 采用免疫组织化学法检测肿瘤组织中CCNA2蛋白表达水平, 分析比较CCNA2蛋白表达水平与患者临床病理特征的关系. 免疫组织化学检测CCNA2蛋白表达按试剂盒操作说明进行. CCNA2蛋白表达高低表达标注: 在显微镜下随机选取3个高倍视野(SP×400)下进行观察. 染色强度评分: 不着色(0分)、浅黄(1分)、棕黄(2分)、棕褐色(3分); 阳性细胞比例评分: 阳性<10%(0分); 阳性10到25%(1分); 阳性26-50%(2分); 阳性51-75%(3分)及阳性>75%(4分). 总体评分值=阳性细胞比例评分×染色强度评分; 最终评判: <6分为低表达, ≥6分为高表达.
统计学处理 Stata 12.0软件进行分析, 计量资料应用Mean±SD表示, t检验; 计数资料采用n表示, χ2检验, CCNA2基因高低表达组生存期采用Log-rank检验, P<0.05为存在统计学差异.
在正常组织中CCNA2中mRNA在食管组织、睾丸组织和脾脏中表达最高, 而在肝脏、胰腺和骨骼肌中表达最低(图1A). 在肿瘤组织中, 睾丸癌、HCC和宫颈癌中表达水平相对较高, 而甲状腺癌、前列腺癌和肾癌表达水平较低(图1B). 在HCC中, CCNA2中mRNA表达水平明显高于癌旁正常肝组织(图1C).
通过STRING数据库, 构建CCNA2蛋白-蛋白相互作网, 与CCNA2蛋白相互作用较为紧密的蛋白10个, 蛋白间相互作用关系edge = 50, 区域聚类指数为0.931, 与CCNA2蛋白相互作用较为紧密的10蛋白相互作用网络富集显著(P<0.05)(图2).
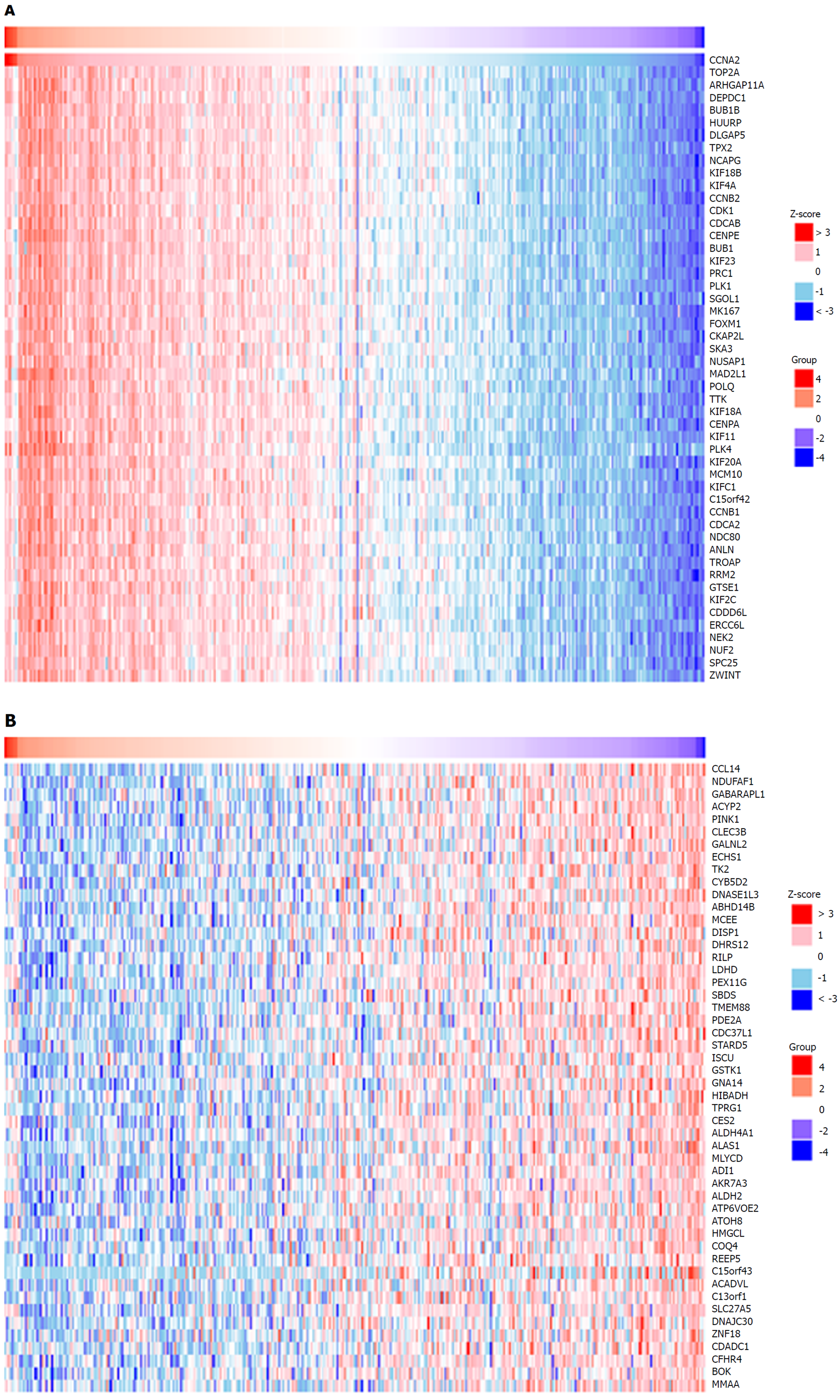
对与CCNA2基因正负相关表达的基因进行了聚类分析(图3), TOP2基因mRNA与CCNA2正向相关表达(r = 0.85, P<0.05)(图4A). 而CCL14基因与CCNA2负相关表达(r = -0.54, P<0.05)最为显著[11](图4B).
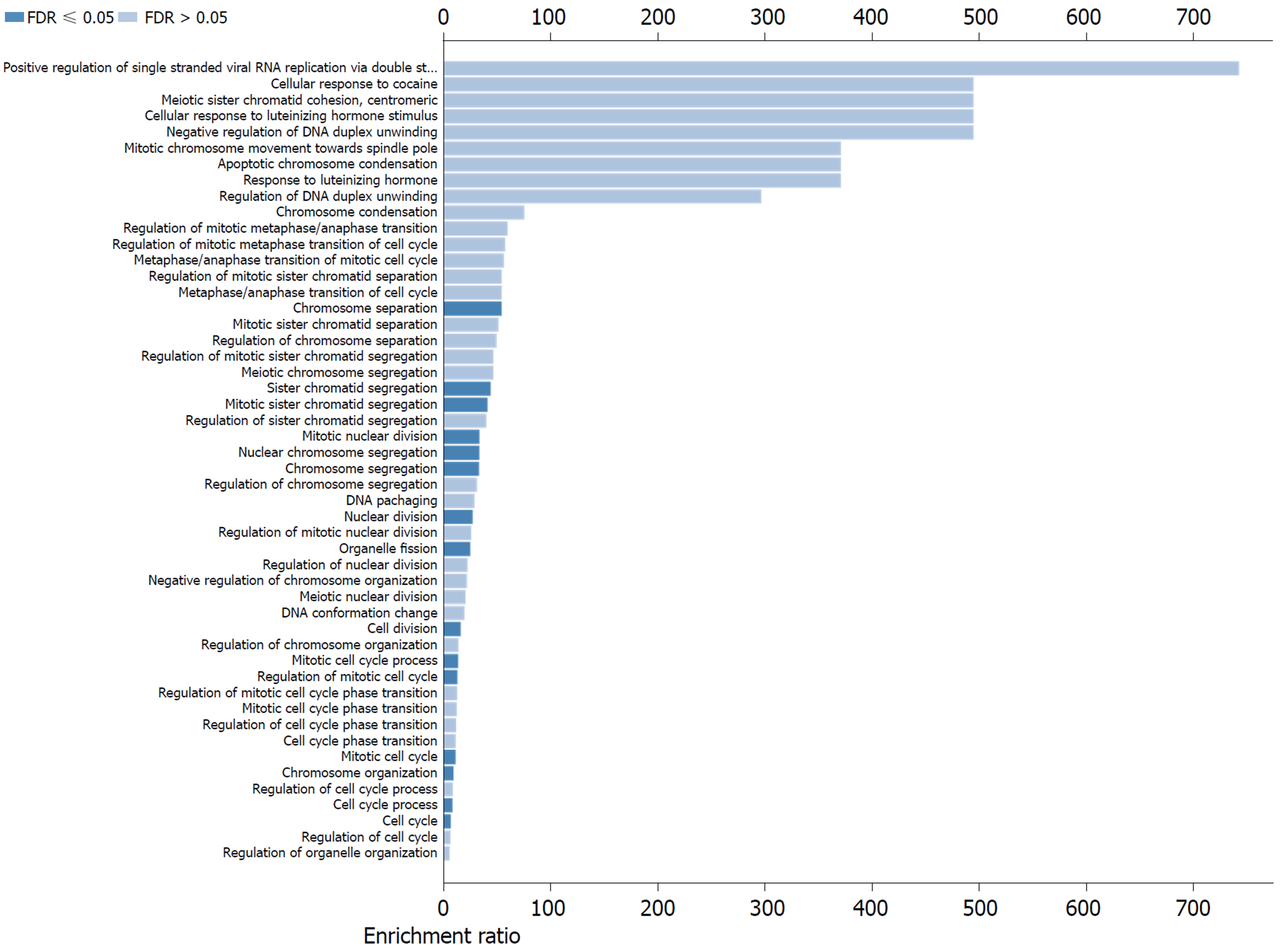
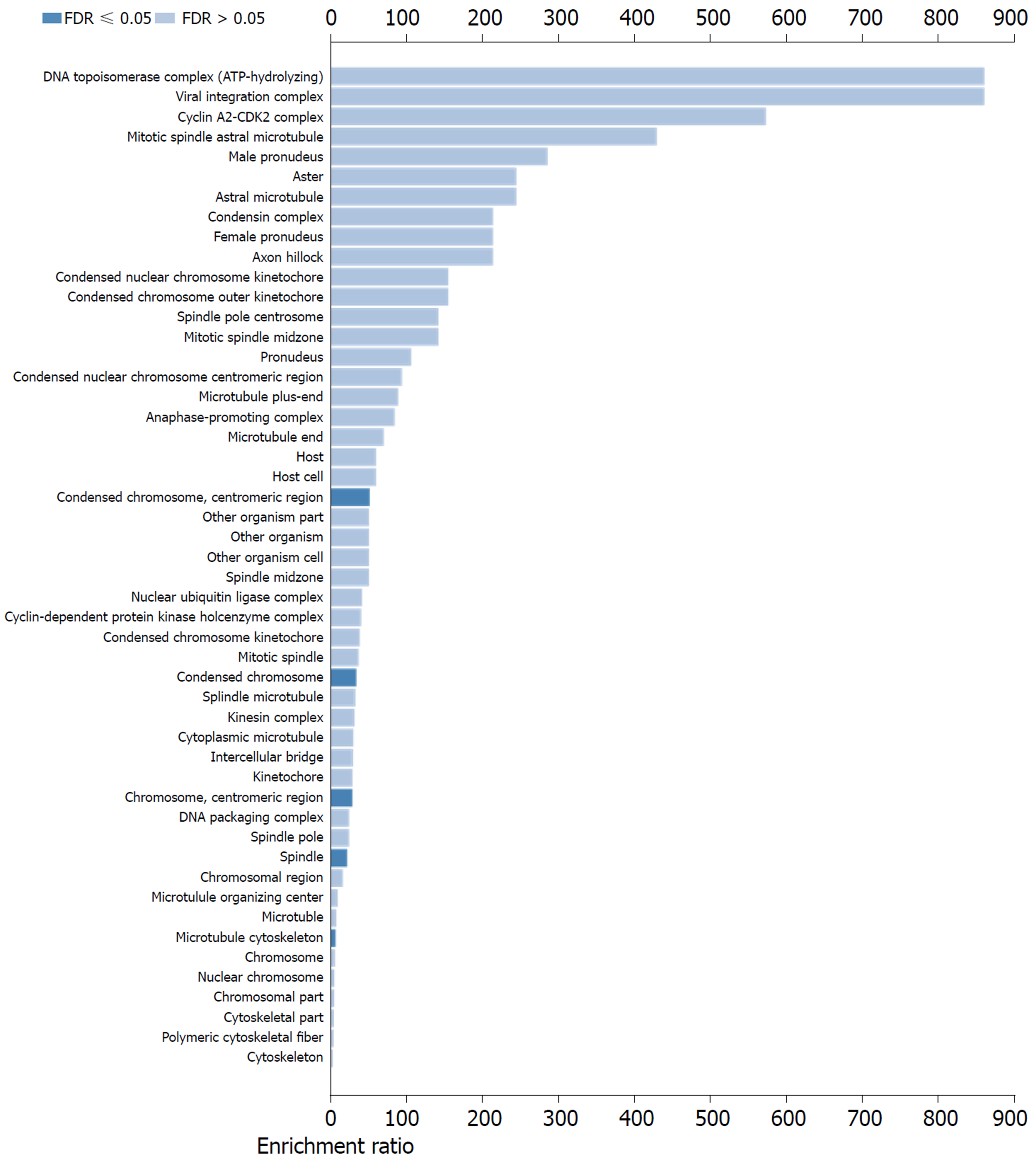
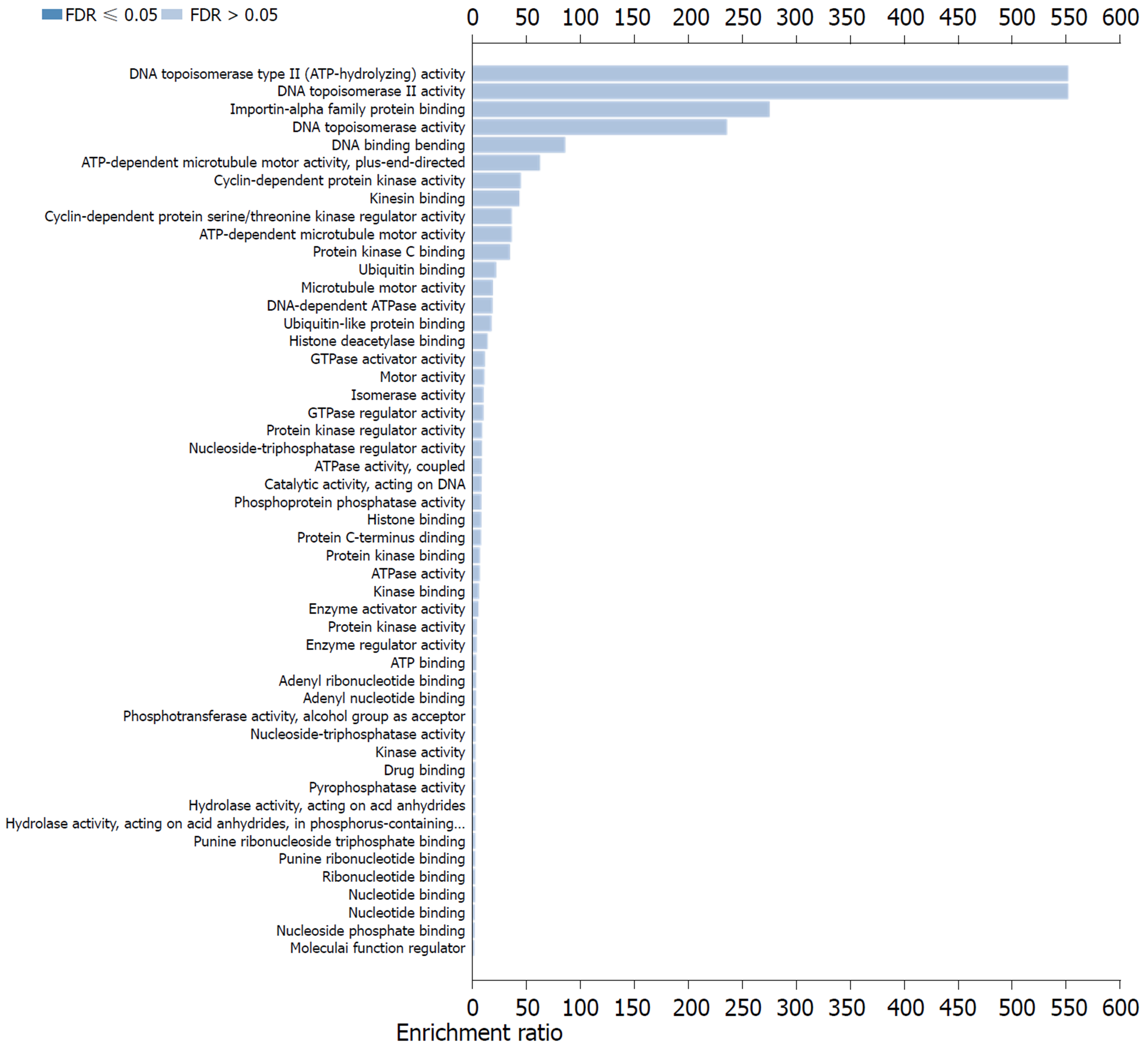
CCNA2生物学过程主要富集于核分裂、染色体分离、有丝分裂细胞周期过程等(图5); 细胞成分主要富集于凝聚染色体、染色体、着丝粒区和微管细胞骨架等(图6). 而分子功能分别主要富集于APT酶活性、DNA拓扑异构酶Ⅱ型(DNA水解酶)活性和DNA拓扑异构酶活性等(图7).
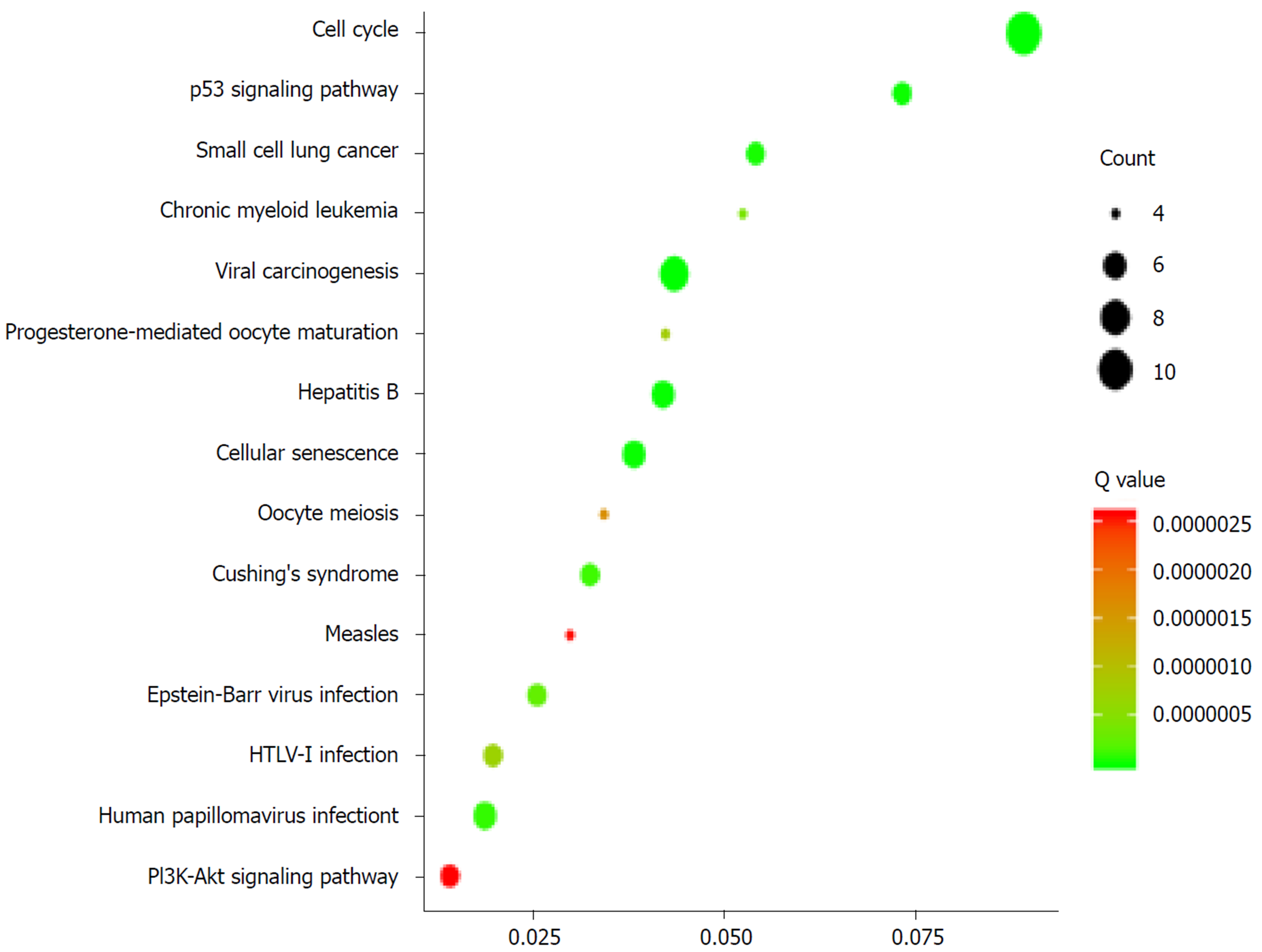
CCNA2基因相关信号通路主要富集于细胞周期、病毒致癌、乙型肝炎、p53信号通路和PI3K-Akt信号通路等(图8, 表1).
| 信号通路 | 基因数 | P值 | 基因比例 |
| 细胞周期 | 11 | 4.98E-23 | 0.089431 |
| 病毒致癌 | 8 | 2.79E-13 | 0.043716 |
| 乙型肝炎 | 6 | 1.23E-09 | 0.042254 |
| 细胞衰老 | 6 | 1.60E-09 | 0.038462 |
| p53信号通路 | 5 | 2.79E-09 | 0.073529 |
| 小细胞肺癌 | 5 | 9.92E-09 | 0.054348 |
| 人乳头瘤病毒感染 | 6 | 5.81E-08 | 0.018927 |
| 库欣综合征 | 5 | 8.74E-08 | 0.03268 |
| EB病毒感染 | 5 | 2.47E-07 | 0.025773 |
| 慢性粒细胞白血病 | 4 | 4.09E-07 | 0.052632 |
| HTLV-I感染 | 5 | 6.96E-07 | 0.02 |
| 孕酮介导的卵母细胞成熟 | 4 | 7.74E-07 | 0.042553 |
| 卵母细胞减数分裂 | 4 | 1.62E-06 | 0.034483 |
| PI3K-Akt信号通路 | 5 | 2.56E-06 | 0.014368 |
依据CCNA2基因mRNA在HCC患者癌组织中表达的中位数分为高低表达组, 预后分析提示CCNA2基因mRNA高表患者OS(HR = 1.7, P = 0.0037)(图9A)和DFS低于低表达组(HR = 1.6, P = 0.0037)(图9B).
免疫组化显示CCNA2蛋白主要表达于HCC细胞核, 呈现棕褐色颗粒(图10). CCNA2蛋白在HCC患者癌组织中的高表达率为34.7%(25/72). CCNA2蛋白高表达与HCC患者肿瘤直径(P<0.05)、DC浸润(P<0.05)和术后2年复发/转移(P<0.05)有关(表2).
| 特征 | n = 72 | CCNA2蛋白 | χ2 | P值 | |
| 高表达(n = 25) | 低表达(n = 47) | ||||
| 性别 | 0.94 | 0.33 | |||
| 男性 | 66 | 24 | 42 | ||
| 女性 | 6 | 1 | 5 | ||
| 年龄(岁) | 0.12 | 0.73 | |||
| ≤60 | 50 | 18 | 32 | ||
| >60 | 22 | 7 | 15 | ||
| 肿瘤大小(cm) | |||||
| ≤5 | 24 | 4 | 20 | 5.18 | 0.02 |
| >5 | 48 | 21 | 27 | ||
| 分化程度 | 1.00 | 0.32 | |||
| 高中分化 | 54 | 17 | 37 | ||
| 低分化 | 18 | 8 | 10 | ||
| DC浸润 | 4.23 | 0.03 | |||
| 少量 | 35 | 8 | 27 | ||
| 大量 | 37 | 17 | 20 | ||
| AFP(μg/L) | 0.88 | 0.35 | |||
| ≤400 | 40 | 12 | 28 | ||
| >400 | 32 | 13 | 19 | ||
| 癌栓 | 1.43 | 0.23 | |||
| 有 | 7 | 1 | 6 | ||
| 无 | 65 | 24 | 41 | ||
| 包膜完整 | 2.32 | 0.12 | |||
| 是 | 29 | 8 | 21 | ||
| 否 | 43 | 17 | 26 | ||
| 术后2年复发/转移 | 5.04 | 0.02 | |||
| 是 | 25 | 13 | 12 | ||
| 否 | 47 | 12 | 35 | ||
HCC患者的总中位生存期为4 mo左右, 总5年生存率为3%. HCC的预后较差, 相关预后因素包括肿瘤大小、是否存在癌栓等[12]. 近年来随着分子生物学技术和生物信息学的发展, 越来越多的证据显示, HCC的发生发展是一个多基因多步骤的过程, 由肝纤维化逐渐发展为肝硬化进而发展为HCC. 同时也有研究显示[13], 从肝纤维化到HCC转化的过程中, 肝组织中的基因表达谱呈现明显的改变, 检测这些基因表达普的改变, 有助于判断HCC的病理进程, 同时也对HCC的诊断和预后具有一定的预测价值.
已有研究显示[8]CCNA2基因在多种肿瘤中呈现高表达, 而CNNA2在HCC的研究未见报道. CCNA2定位于人4号染色体, q27区, 基因全长7489 bp, 该基因编码蛋白属于高度保守的细胞周期蛋白家族, 在人体几乎所有组织中均有表达[14]. 它在G1/S期和G2/M期的细胞周期控制中起着关键作用. 同时又研究报道, miR-22在HCC中对其具有调控作用[15,16]. 来自人类蛋白质图谱的数据显示[17], CCNA2在人体多种恶性肿瘤中呈现过度表达, 表明其在癌症转化和进展中的潜在作用. 也有报道称, CCNA2可能参与上皮-间质转化和转移的过程[18]. 然而, CCNA2在HCC中的研究未见报道, 其在HCC组织和癌旁正常组织中的表达水平、生物学功能及与患者预后的关系并不清楚. 在本研究中, 首先采用生物信息学分析CNNA2在HCC中的表达及相关信号通路. 我们发现在HCC中, CCNA2中mRNA表达水平明显高于癌旁正常肝组织, 提示CCNA2在HCC的发生发展中可能发挥重要作用, 这与既往CCNA2在其他肿瘤中的研究结果基本一致; CCNA2基因相关信号通路主要富集于细胞周期、病毒致癌、乙型肝炎、p53信号通路和PI3K-Akt信号通路等, 上述信号通路符合HCC已知在HCC中的作用机制. 共表达分析显示, TOP2基因mRNA与CCNA2正向相关表达(r = 0.85, P<0.05), 而CCL14基因与CCNA2负相关表达(r = -0.54, P<0.05)最为显著. 提示TOP2基因可能在生物学功能方面与CCNA2具有协同效应, 而CCL14与CCNA2具有拮抗作用. 但上述协同和拮抗作用有待相关实验进一步证实.
预后分析提示CCNA2基因mRNA高表患者OS(HR = 1.7, P = 0.0037); 和DFS低于低表达组(HR = 1.6, P = 0.0037). 免疫组化显示CCNA2蛋白在HCC患者癌组织中的高表达率为34.7%. CCNA2蛋白高表达者肿瘤直径较大、DC浸润较为严重及术后复发率较高. 上述因素佐证了CCNA2高表达是HCC患者预后不良的危险因素.
CCNA2基因在HCC患者癌组织中表达水平上调, 并可作为HCC预后不良的分子标记物. 同时, CCNA2也可作为HCC靶向药物开发的潜在靶点, 进一步对其调控HCC相关分子生物学功能开展相关研究, 有望开发出抑制HCC的靶向药物.
已有研究显示CCNA2基因在多种肿瘤中呈现高表达, 而CNNA2在肝细胞癌(hepatocellular carcinoma, HCC)的研究未见报道, 其生物学功能及其作为HCC预后分子标志物可行性不清.
通过生物信息学分析和免疫组织化学实验, 分析CCNA2在HCC中的表达情况, 判断其表达与HCC患者临床病理特征的关系及其作为HCC预后分子标志物的可行性.
探讨CCNA2在HCC中的表达及其作为HCC预后分子标志物的临床价值.
比对TCGA数据库中CCNA2基因mRNA在HCC组织和癌旁组织中表达水平; 构建CCNA2蛋白-蛋白相互作用网络, 并对相关蛋白功能和KEGG信号通路进行富集. 比较CCNA2高低组患者总生存和无疾病进展生存是否不同. 采用免疫组织化学法检测患者癌组织中CCNA2蛋白表达水平与患者临床病理特征的关系, 对生物信息分析结果进行验证.
CCNA2基因在HCC患者癌组织中表达水平显著高于癌旁正常组织, CCNA2蛋白高表达与HCC患者肿瘤直径(P<0.05)、DC浸润(P<0.05)、术后2年复发/转移(P<0.05)和预后不良有关.
CCNA2基因在HCC患者癌组织中表达水平上调, 并可作为HCC预后不良的分子标记物.
CCNA2在干细胞癌患者癌组织中高表达, 且高表达与预后不良有关. 研究结果为开发抑制HCC中CCNA2表达靶向治疗药物提供了新的潜在靶点.
学科分类: 胃肠病学和肝病学
手稿来源地: 天津市
同行评议报告分类
A级 (优秀): 0
B级 (非常好): B, B, B
C级 (良好): 0
D级 (一般): D
E级 (差): 0
编辑:王禹乔 电编:刘继红
| 1. | Siegel RL, Miller KD, Jemal A. Cancer statistics, 2019. CA Cancer J Clin. 2019;69:7-34. [PubMed] [DOI] |
| 2. | Chen W, Zheng R, Baade PD, Zhang S, Zeng H, Bray F, Jemal A, Yu XQ, He J. Cancer statistics in China, 2015. CA Cancer J Clin. 2016;66:115-132. [PubMed] [DOI] |
| 3. | Serrano PE, Gu CS, Husien M, Jalink D, Ritter A, Martel G, Tsang ME, Law CH, Hallet J, McAlister V, Sela N, Solomon H, Moulton CA, Gallinger S, Levine M. Risk factors for survival following recurrence after first liver resection for colorectal cancer liver metastases. J Surg Oncol. 2019;120:1420-1426. [PubMed] [DOI] |
| 4. | Smith JW, Kroker-Lobos MF, Lazo M, Rivera-Andrade A, Egner PA, Wedemeyer H, Torres O, Freedman ND, McGlynn KA, Guallar E, Groopman JD, Ramirez-Zea M. Aflatoxin and viral hepatitis exposures in Guatemala: Molecular biomarkers reveal a unique profile of risk factors in a region of high liver cancer incidence. PLoS One. 2017;12:e0189255. [PubMed] [DOI] |
| 5. | Suh JK, Lee J, Lee JH, Shin S, Tchoe HJ, Kwon JW. Risk factors for developing liver cancer in people with and without liver disease. PLoS One. 2018;13:e0206374. [PubMed] [DOI] |
| 6. | VoPham T. Environmental risk factors for liver cancer and nonalcoholic fatty liver disease. Curr Epidemiol Rep. 2019;6:50-66. [PubMed] [DOI] |
| 7. | Gan Y, Li Y, Li T, Shu G, Yin G. CCNA2 acts as a novel biomarker in regulating the growth and apoptosis of colorectal cancer. Cancer Manag Res. 2018;10:5113-5124. [PubMed] [DOI] |
| 8. | Gao T, Han Y, Yu L, Ao S, Li Z, Ji J. CCNA2 is a prognostic biomarker for ER+ breast cancer and tamoxifen resistance. PLoS One. 2014;9:e91771. [PubMed] [DOI] |
| 9. | Wu K, Yin X, Jin Y, Liu F, Gao J. Identification of aberrantly methylated differentially expressed genes in prostate carcinoma using integrated bioinformatics. Cancer Cell Int. 2019;19:51. [PubMed] [DOI] |
| 10. | Hoffmann R, Valencia A. A gene network for navigating the literature. Nat Genet. 2004;36:664. [PubMed] [DOI] |
| 11. | Vasaikar SV, Straub P, Wang J, Zhang B. LinkedOmics: analyzing multi-omics data within and across 32 cancer types. Nucleic Acids Res. 2018;46:D956-D963. [PubMed] [DOI] |
| 12. | Hiraoka A, Kumada T, Atsukawa M, Hirooka M, Tsuji K, Ishikawa T, Takaguchi K, Kariyama K, Itobayashi E, Tajiri K, Shimada N, Shibata H, Ochi H, Tada T, Toyoda H, Nouso K, Tsutsui A, Nagano T, Itokawa N, Hayama K, Imai M, Joko K, Koizumi Y, Hiasa Y, Michitaka K, Kudo M; Real-life Practice Experts for HCC (RELPEC) Study Group, HCC 48 Group (hepatocellular carcinoma experts from 48 clinics in Japan). Prognostic factor of lenvatinib for unresectable hepatocellular carcinoma in real-world conditions-Multicenter analysis. Cancer Med. 2019;8:3719-3728. [PubMed] [DOI] |
| 13. | Ogawa S, Kumada T, Toyoda H, Ichikawa H, Kawachi T, Otobe K, Hibi T, Takeshima K, Kiriyama S, Sone Y, Tanikawa M, Hisanaga Y, Yamaguchi A, Isogai M, Kaneoka Y, Washizu J. Evaluation of pathological features of hepatocellular carcinoma by contrast-enhanced ultrasonography: comparison with pathology on resected specimen. Eur J Radiol. 2006;59:74-81. [PubMed] [DOI] |
| 14. | Ko E, Kim Y, Cho EY, Han J, Shim YM, Park J, Kim DH. Synergistic effect of Bcl-2 and cyclin A2 on adverse recurrence-free survival in stage I non-small cell lung cancer. Ann Surg Oncol. 2013;20:1005-1012. [PubMed] [DOI] |
| 15. | Yang F, Hu Y, Liu HX, Wan YJ. MiR-22-silenced cyclin A expression in colon and liver cancer cells is regulated by bile acid receptor. J Biol Chem. 2015;290:6507-6515. [PubMed] [DOI] |
| 16. | Yang F, Gong J, Wang G, Chen P, Yang L, Wang Z. Waltonitone inhibits proliferation of hepatoma cells and tumorigenesis via FXR-miR-22-CCNA2 signaling pathway. Oncotarget. 2016;7:75165-75175. [PubMed] [DOI] |
| 17. | Saviranta P, Kallioniemi O. Human Protein Atlas charts a diverse terrain. Trends Biotechnol. 2006;24:195-197. [PubMed] [DOI] |
| 18. | Bendris N, Arsic N, Lemmers B, Blanchard JM. Cyclin A2, Rho GTPases and EMT. Small GTPases. 2012;3:225-228. [PubMed] [DOI] |