修回日期: 2015-07-07
接受日期: 2015-07-14
在线出版日期: 2015-08-18
目的: 探索CRISPR干扰(CRISPR interference, CRISPRi)特异性抑制肝癌细胞HepG2内源性miR-122表达.
方法: 设计靶向miR-122启动子区TATA盒和转录起始位点(transcription start site, TSS)的sgRNA(sgT1和sgT2), 与无DNA内切酶活性仅保留识别特性的dCas9-KRAB载体共转染HepG2细胞, 通过实时定量PCR(quantitative Real-time PCR, qRT-PCR)方法检测miR-122的表达并确立有效的sgRNA; 设计不同的sgRNA浓度, 探索CRISPRi抑制作用的剂量依赖性; 通过qRT-PCR及Western blot方法检测miR-122靶分子HOMX1和CyclinG1的表达变化.
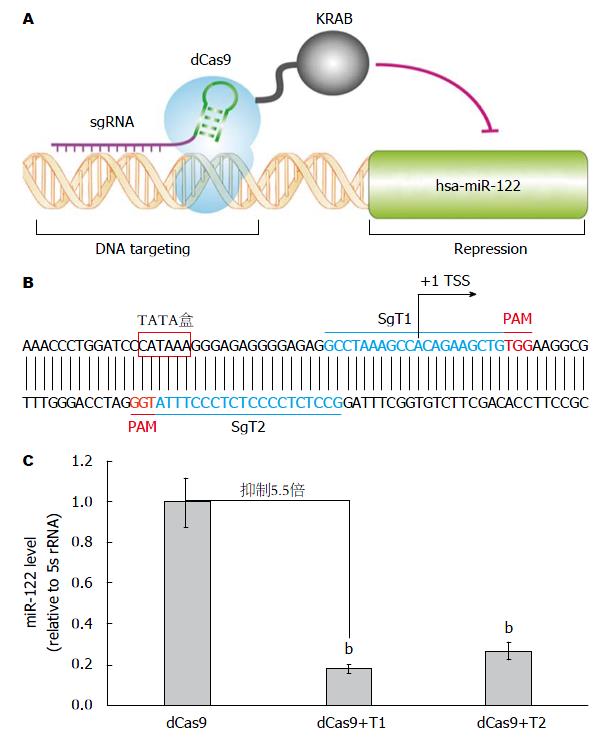
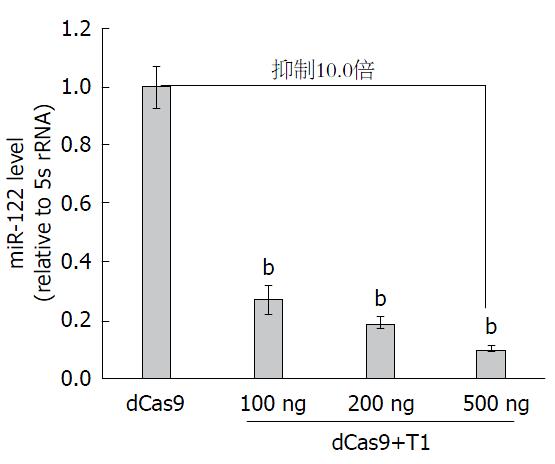
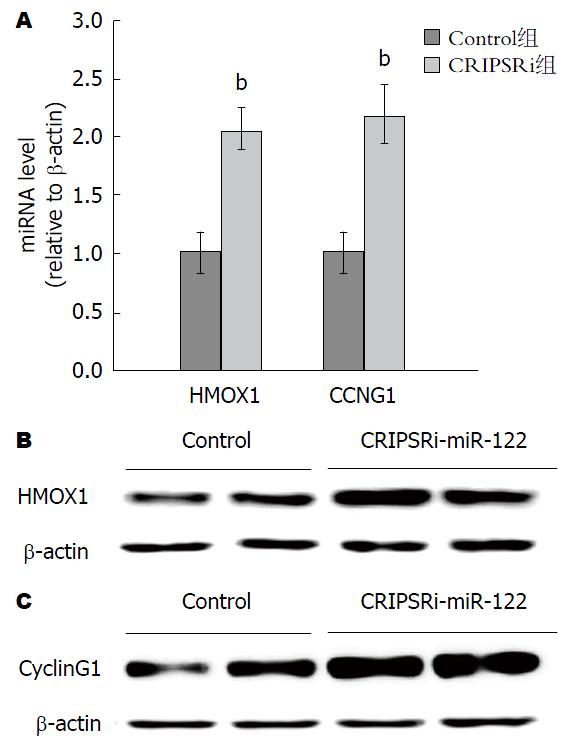
结果: sgT1和sgT2介导的CRISPRi分别将肝癌细胞HepG2内源性miR-122的表达水平抑制5.5倍(P<0.01)和3.7倍(P<0.01); CRIPSRi抑制效应是sgRNA剂量依赖性的, 当lentiGuide-Puro-sgT1质粒为500 ng时, 可将miR-122的表达抑制10.0倍(P<0.01); CRIPSRi抑制肝癌细胞HepG2内源性miR-122表达的同时, 上调了miR-122下游靶分子HMOX1和CyclinG1的表达.
结论: 本研究利用CRISPRi技术实现了对肝癌细胞HepG2内源性miR-122表达的特异性抑制, 为抗丙型肝炎病毒(hepatitis C virus, HCV)提供了全新的策略.
核心提示: 利用CRISPR干扰技术实现了对肝癌细胞HepG2内源性miR-122表达的靶向抑制, 并上调了miR-122下游靶分子HMOX1和CyclinG1的表达, 为抗丙型肝炎病毒(hepatitis C virus)提供了全新的策略.
引文著录: 赵洪礼, 李洪运, 毛德华, 李桂玲, 黄勇. CRISPRi靶向调控HCV复制相关miR-122的表达. 世界华人消化杂志 2015; 23(23): 3742-3748
Revised: July 7, 2015
Accepted: July 14, 2015
Published online: August 18, 2015
AIM: To investigate whether clustered regularly interspaced short palindromic repeat (CRISPR) interference (CRISPRi) specifically represses endogenous miR-122 expression in HepG2 cells.
METHODS: Single-guide RNAs (sgRNAs; sgT1 and sgT2) targeting transcription start site (TSS) and TATA box in the region of miR-122 promoter were designed. After co-transfecting HepG2 cells with sgRNA and catalytically inactive dCas9-KRAB, the expression of miR-122 was determined by quantitative Real-time PCR (qRT-PCR) to determine the more effective sgRNA. Different concentrations of sgRNA were tested in order to address whether CRISPRi was concentration dependent. The expression of miR-122 downstream target genes HOMX1 and CyclinG1 was assessed by qRT-PCR and Western blot.
RESULTS: Compared to the control group, CRISPRi mediated by sgT1 and sgT2 could repress endogenous miR-122 expression by 5.5-fold (P < 0.01) and 3.7-fold (P < 0.01) in HepG2 cells, respectively. The effect of CRISPRi was enhanced with increased concentration of sgRNA, and the miR-122 expression was inhibited by 10.0-fold (P < 0.01) when the amount of lentiGuide-Puro-sgT1 was 500 ng. After endogenous miR-122 expression was inhibited by CRISPRi, the expression of miR-122 downstream target genes HMOX1 and CyclinG1 was upregulated.
CONCLUSION: CRISPRi can specifically repress endogenous miR-122 expression, which provides a novel therapeutic strategy against hepatitis C virus infection.
- Citation: Zhao HL, Li HY, Mao DH, Li GL, Huang Y. Regulation of expression of HCV replication associated miR-122 by CRISPR interference. Shijie Huaren Xiaohua Zazhi 2015; 23(23): 3742-3748
- URL: https://www.wjgnet.com/1009-3079/full/v23/i23/3742.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v23.i23.3742
丙型肝炎是一种致命的血液传染病, 世界卫生组织(World Health Organization, WHO)估计全球目前约有1.7亿人受丙型肝炎病毒(hepatitis C virus, HCV)感染, 其中, 约70%会发展成肝硬化和肝癌等疾病. HCV引起的肝脏疾病已经成为公众健康的主要挑战之一. HCV是单链的RNA病毒, 存在多种基因型和亚型, 这对抗HCV病毒治疗极具挑战. 尽管目前的抗病毒药物如聚乙二醇化干扰素和利巴韦林具有一定的疗效, 然而这些药物的低耐药屏障, 导致在长期治疗过程中耐药性病毒突变体的产生[1-3].
微小RNA(microRNA, miRNA)是一种内源性的小片段非编码RNA, 通过与靶分子mRNA 3'端非翻译区(3' untranslated region, 3'UTR)结合, 介导靶分子转录后水平的调节, 导致mRNA降解或翻译抑制[4]. miRNA广泛参与多种重要生物过程的调节, 包括细胞增殖、分化、发育、凋亡以及病毒感染的宿主反应调节. miR-122是肝脏特异性表达的miRNA, 占肝组织中miRNA的70%, 参与HCV RNA的稳定和复制[5,6]. miR-122与HCV RNA的5'UTR的两个靶位点结合, 形成miR-122-HCV复合体, 保护HCV RNA不被核酸内切酶水解或者不被宿主免疫反应清除[6,7]. miR-122结合位点在HCV RNA的所有基因型及亚型中高度保守, 这使得miR-122成为抗HCV病毒治疗的宿主靶点.
CRISPR(clustered regularly interspaced short palindromic repeat sequences)/CRISPR相关基因(CRISPR-associated genes, Cas)系统广泛分布于细菌和古生菌基因组中, 是在进化过程中形成的一种适应性免疫系统, 可以降解入侵病毒或质粒DNA. 2012年, Jinek等[8]首先利用CRISPR/Cas9系统对外源DNA靶序列进行了精确切割, 开辟了RNA(tracrRNA与crRNA)介导的基因编辑时代. Cas9含有2种核酸酶结构域: RuvC1结构域及HNH结构域, HNH结构域切割与crRNA互补的模板链, RuvC1结构域对另一条链进行切割[9]. Qi等[10]将Cas9的核酸内切酶结构域突变(RuvC1, D10A; HNH, H841A), 产生dCas9, 只有DNA识别能力, 没有内切酶活性, 当dCas9与sgRNA共表达时, 形成识别特定位点的复合体, 该复合体可以特异性的干扰RNA聚合酶结合或者转录因子结合, 有效抑制靶分子的表达, 将此过程称为CRISPR干扰(CRISPR interference, CRISPRi). 本研究利用CRISPRi技术实现了HepG2细胞内miR-122的特异性表达抑制, 为抗HCV病毒提供了全新的策略.
HepG2细胞购自上海细胞库, 载体构建用的BsmBⅠ酶、碱性磷酸酶FastAP、T4连接酶购自Fermentas公司; dCas9-KRAB质粒(#46911)、lentiGuide-Puro质粒(#52963)购自Addgene; 核酸纯化试剂盒(EasyPure Quick Gel Extraction Kit, EG101-01)、反转录试剂盒购自北京全式金生物公司; 引物由上海生工合成; 细胞培养用胎牛血清购自Hyclone公司; VigoFect转染试剂购自威格拉斯; 定量PCR试剂盒购自大连宝生物; TRIzol(Invitrogen), polyA聚合酶购自NEB; SYBR Premix Ex Tag试剂盒(TaKaRa, code: DRR041A); anti-HMOX1(heme oxygenase 1)一抗(Abcam cat. ab137749); anti-Cyclin G1一抗(sc-7865, Santa Cruz); anti-β-actin一抗(Abcam cat. 6276); RIPA Buffer裂解液、考马斯亮蓝(R-250)购自碧云天生物; Western显色液购自Thermo公司.
1.2.1 载体构建: 靶向miR-122启动子sgRNA载体的构建: 采用BsmBⅠ酶将lentiGuide-Puro载体线性化, 并通过碱性磷酸酶FastAP去磷酸化, 利用EasyPure Quick Gel Extraction Kit进行核酸纯化. 将合成的sgT1(sgT1-F: 5'-CACCGCCTAAAGCCACAGAAGCTG-3'和sgT1-R: 5'-AAACCAGCTTCTGTGGCTTTAGGC-3')、sgT2(sgT2-F: 5'-CACCGCCTCTCCCCTCTCCCTTTA-3'和sgT2-R: 5'-AAACTAAAGGGAGAGGGGAGAGGC-3')分别进行退火与磷酸化, 并与线性化的lentiGuide-Puro载体进行连接, 分别构建靶向miR-122启动子区不同位点的sgRNA载体lentiGuide-Puro-sgT1和lentiGuide-Puro-sgT2.
1.2.2 HepG2细胞培养及转染: HepG2细胞培养按常规方法传代培养. HepG2肝癌细胞在含有10%的胎牛血清、终浓度为1×105 U/L的青霉素和链霉素的RPMI 1640培养基中37 ℃, 50 mL/L CO2条件下培养数天, 每2 d换液1次, 4 d传代. 按照VigoFect转染试剂说明书进行HepG2肝癌细胞转染, (1)筛选sgRNA: 共同转染dCas9-KRAB与lentiGuide-Puro(空载体)或lentiGuide-Puro-sgT1或lentiGuide-Puro-sgT2, 其中dCas9-KRAB质粒250 ng/孔(24孔板), lentiGuide-Puro(空载体)或lentiGuide-Puro-sgT1或lentiGuide-Puro-sgT2 200 ng/孔(24孔板), 收集细胞检测miR-122的表达; (2)筛选sgRNA的浓度: 共同转染dCas9-KRAB与lentiGuide-Puro-sgT1, 其中转染dCas9-KRAB质粒250 ng/孔(24孔板), 转染lentiGuide-Puro-sgT1分别为100 ng、200 ng或500 ng/孔(24孔板), 收集细胞检测miR-122的表达; (3)miR122靶分子表达水平检测: 转染dCas9-KRAB质粒1.0 μg/孔与lentiGuide-Puro-sgT1质粒2.0 μg/孔(6孔板), 收集细胞检测HMOX1, CyclinG1 mRNA及蛋白表达.
1.2.3 总RNA提取及反转录: (1)总RNA提取: 用1 mL TRIzol裂解细胞, 室温放置5 min, 加入0.2 mL氯仿, 盖紧管盖, 手动剧烈震摇15 s, 然后室温静置2-3 min, 4 ℃ 12000 g离心15 min, 小心吸取上层水相加入到新EP管中, 加入0.5 mL异丙醇颠倒混匀, 室温静置10 min, 4 ℃, 12000 g离心10 min, 后弃上清, 加入1 mL 75 750 mL/L乙醇(DEPC H2O配制)润洗, 4 ℃, 7000 g离心5 min, 弃上清, 将RNA沉淀溶解于适量的无RNase水中, 吸出少量测定浓度, RNA溶液可用于进一步实验, 或者-80 ℃保存; (2)反转录: 取1 μg RNA, 加入1 μL Oligo(dT)20, 无RNase水配成10 μL体系, 65 ℃ 5 min进行变性, 后立即置于冰上, 随后加入2 μL dNTP Mixture、1 μL RNase Inhibitor、1 μL反转录酶以及Buffer和水, 配成20 μL体系, 42 ℃ 1 h, 99 ℃ 5 min, 4 ℃ 5 min, 瞬时离心, -20 ℃保存, 以备逆转录-聚合酶链反应(Real-time polymerase chain reaction, RT-PCR)使用.
1.2.4 miRNA定量检测: 采用polyA聚合酶加A法[11,12]检测miR-122的表达. (1)加A反应: 取1 μg RNA, 加入1 μL ATP、1 μL PolyA polymerase以及Buffer和无RNase水, 配成10 μL体系, 37 ℃ 1 h, 65 ℃ 20 min, 4 ℃ 5 min; (2)反转录: 加A后, 加入1 μL RNase Inhibitor、1 μL反转录酶、0.5 μL Poly(T) adapter(10 μmol)[序列为GCGAGCACAGAATTAATACGACTCACTATAGG(T)12VN]、2 μL dNTP Mixture以及Buffer和水, 配成20 μL体系, 42 ℃ 1 h, 99 ℃ 5 min, 4 ℃ 5 min; (3)RT-PCR PCR检测miR-122.
1.2.5 定量PCR: 使用SYBR Premix Ex Tag试剂盒, 引物序列如表1, 使用ABI 7500仪器, 采用两步法PCR反应程序, Stage1: 预变性, 95 ℃ 30 s; Stage 2: PCR反应(40 cycles): 95 ℃ 5 s, 60 ℃ 34 s; Stage 3: Dissociation stage: 95 ℃ 15 s, 60 ℃ 1 min, 95 ℃ 15 s. 后进行数据分析.
| 引物名称 | 序列 |
| miR-122-F | 5'-TGGAGTGTGACAATGGTGTTTG-3' |
| miR-122-R | 5'-GCGAGCACAGAATTAATACGAC-3' |
| 5s rRNA-F | 5'-CCGCCTGGGAATACCGGGTGCTGTAGGCTTT-3' |
| 5s rRNA-R | 5'-GCGAGCACAGAATTAATACGAC-3' |
| HOMX1-F | 5'-CGGGCCAGCAACAAAGTG-3' |
| HOMX1-R | 5'-AGTGTAAGGACCCATCGGAGAA-3' |
| CCNG1-F | 5'-AGCTGCAGTCTCTG TCAAG-3' |
| CCNG1-R | 5'-ATGTCTCTGTGTCAAAGCCA-3' |
| β-actin-F | 5'-CTGGAACGGTGAAGGTGACA-3' |
| β-actin-R | 5'-AAGGGACTTCCTGTAACAATGCA-3' |
1.2.6 Western blot: 转染48 h后, 收集细胞, 并利用RIPA Buffer裂解细胞提取总蛋白, 考马斯亮蓝(R-250)测定蛋白浓度, 上样30 μg, 电泳, 转膜, 奶粉封闭, 一抗孵育4 ℃过夜, 洗膜, 二抗孵育(HRP标记, 1:10000), 洗膜, 显色, 采集图像.
统计学处理 采用SPSS12.0统计学处理软件进行t检验分析, 各项检测结果以mean±SD记录. 以P<0.05为差异有统计学意义.
本研究利用CRIPSRi原理(图1), 即利用dCas9仅保留DNA识别能力而无内切酶活性的特点, 并将dCas9与KRAB(Kruppel-associated box)转录抑制结构域融合, 当靶向miR-122启动子区的sgRNA存在时, 将抑制miR-122的转录. 核心启动子元件TATA盒(TATA box)和转录起始位点(transcription start site, TSS)在转录起始过程中发挥重要作用, 因此我们分别设计了靶向TSS和TATA盒的sgRNA,分别命名为T1和T2(图1). 研究结果显示, sgT1和sgT2介导的CRISPRi均能够显著性降低miR-122的表达. 与对照组相比, sgT1和sgT2介导的CRISPRi分别将miR-122的表达水平抑制5.5倍(P<0.01)和3.7倍(P<0.01). 研究结果揭示, CRISPRi可有效地调节miR-122的表达(图1).
在前期结果的基础上, 进一步探索了sgRNA剂量对miR-122抑制效果的影响. 我们选择了抑制效果较为明显的sgT1, 分别采用100 ng、200 ng和500 ng的质粒lentiGuide-Puro-sgT1. 研究结果表明, 随着sgRNA浓度的增加, CRISPRi对miR-122的抑制作用显著性提高. 与对照组相比, 当lentiGuide-Puro-sgT1质粒为500 ng时, 可将miR-122的表达抑制10.0倍(P<0.01). 因此, CRIPSRi抑制肝细胞miR-122表达依赖于sgRNA的剂量, sgRNA剂量越高, 抑制效果越显著(图2).
miR-122通过下调HCV复制抑制分子HMOX1和CyclinG1的表达, 而间接促进HCV复制[13,14]. 为探索CRIPSRi在抑制肝细胞miR-122表达的同时, 能否进一步实现对miR-122下游靶分子的表达调控, 本研究在共转染dCas9-KRAB和lentiGuide-Puro-sgT1后, 检测了HepG2细胞的HMOX1和CyclinG1的mRNA及蛋白表达, 结果显示, 在转染dCas9-KRAB和lentiGuide-Puro-sgT1后HMOX1 mRNA和CCNG1 mRNA水平均显著升高, 同时HMOX1和CyclinG1蛋白水平也有明显升高. 因此, CRIPSRi在抑制肝细胞miR-122表达的同时, 并实现上调miR-122下游靶分子HMOX1和CyclinG1的表达(图3).
本研究首次利用CRIPSRi抑制了肝细胞miR-122表达, 并且实现上调miR-122下游靶分子HMOX1和CyclinG1的表达, 为CRISPRi应用于抗HCV病毒的治疗提供了理论依据. HCV的生活周期分进入靶细胞、复制、包装和释放新病毒三个阶段. HCV的持续感染率和聚乙二醇化干扰素和利巴韦林联合抗病毒治疗的局限性, 使得研发HCV新的治疗策略显得尤为重要. Jopling等[6]首次证实, miR-122与HCV的复制有关, 当miR-122失去活性时, HCV的RNA复制降低约80%. 与多数miRNA作用于靶分子的mRNA的3'UTR的方式不同的是, miR-122通过2个结合位点作用于HCV RNA 5'UTR, 从而促进病毒复制[15-17]. 由于miR-122由宿主细胞所控制, 病毒无法将其改变, 而miR-122是病毒复制所必需, 这使得miR-122成为治疗丙型肝炎的新的抗病毒靶点. Lanford等[17]利用RNA干扰(RNA interference, RNAi)的原理, 采用miR-122的反义锁核酸(locked-nucleic acid, LNA)miravirsen(SPC3469)治疗基因Ⅰ型HCV慢性感染的黑猩猩, 结果显示SPC3469可持久抑制HCV病毒, 并且未出现病毒耐药, 开启了靶向miR-122治疗丙型肝炎的先河. CRISPR作为一种高效而又易于操作的基因编辑技术, 引起科学界的广泛关注, 成为全球科学家的新宠, CRISPR/Cas9系统已经被广泛应用于模式细胞及模式动物的基因编辑, 包括人细胞、小鼠、大鼠及斑马鱼等. Qi等[10]将CRISPR/Cas9改造为CRISPRi体系, CRISPRi通过干扰DNA的转录实现对靶分子的表达抑制. Gilbert等[18]研究结果揭示, 在人类细胞系中通过CRISPRi可将靶基因的表达抑制5.0-15.0倍, 在酵母中则高达50.0倍. 此外, Gilbert等[18]还发现CRISPRi具有高效且特异地转录抑制作用, 几乎不发生脱靶效应; 由于CRISPRi作用于DNA水平, CRISPRi可应用于非编码RNA(noncoding RNA, ncRNA)、miRNA、转录物的反义序列、核定位的RNA以及聚合酶Ⅲ转录产物. RNAi是目前广泛应用于基因转录抑制的方法[19]. RNAi和CRISPRi在许多研究中成为互补的研究方法, 然而CRISPRi更具有潜在的优势. Miravirsen作为新的抗HCV宿主靶向药物, 他具有较高的基因屏障、泛基因型的抗病毒活性, 将应用于丙型肝炎临床治疗. 但是, 相对于锁核酸标记的寡核苷酸成本高、易降解的特点, 利用CRISPRi技术具有其自身的优势: 成本低, 易操作, 高效. 本研究利用CRISPRi技术实现了HepG2细胞内miR-122的特异性表达抑制. 同时, CRIPSRi抑制肝细胞miR-122表达依赖于sgRNA的剂量, sgRNA剂量越高, 抑制效果越显著. 此外, CRISPRi可以同时抑制过个基因的表达, 并且抑制作用是可逆的[10]. 因此, 将CRISPRi应用于抗HCV病毒的治疗具有更加广阔的前景.
值得注意的是, 除了直接作用方式, miR-122通过下调HCV复制抑制分子HMOX1和CyclinG1的表达, 而间接促进HCV复制[3,13,14]. Shan等[13]研究发现, miR-122的反义互补序列antagomir通过上调HMOX1的表达进而抑制HCV病毒复制. Hou等[14]研究发现, siRNA介导的CyclinG1的沉默显著增加了细胞内HCV RNA水平, 说明CyclinG1在HCV复制中发挥重要作用; 而miR122的抑制剂增加了CyclinG1的表达, 并且阻止了乙醇诱导的HCV RNA水平. 本研究利用CRISPRi技术实现了HepG2细胞内miR-122的特异性表达抑制, 同时, miR-122下游靶分子HMOX1和CyclinG1均表达升高, 这进一步说明了CRISPRi技术在针对miR-122宿主靶点的应用为抗HCV治疗开拓了新的思路.
丙型肝炎是一种致命的血液传染病, 全球约有1.7亿人感染丙型肝炎病毒(hepatitis C virus, HCV), 其中约70%会发展成肝硬化和肝癌等疾病. 然而到目前为止仍没有一种有效的治疗方式. 已有研究发现微小RNA(microRNA, miRNA)-122是HCV复制所必需的, 因此miR-122有可能成为治疗丙型肝炎的新的抗病毒靶点.
秦建民, 主任医师, 上海中医药大学附属普陀医院普外科
miR-122是HCV病毒复制所必需的, 而且miR-122由宿主细胞所控制, 病毒无法将其改变, 因此靶向抑制miR-122成为治疗丙型肝炎的重点, 而探索特异性强、效率高的抑制方式则成为丙型肝炎治疗的关键.
Lanford等利用RNA干扰的原理, 采用miR-122的反义锁核酸miravirsen(SPC3469)治疗基因Ⅰ型HCV慢性感染的黑猩猩, 结果显示SPC3469可持久抑制HCV病毒, 并且未出现病毒耐药, 开启了靶向miR-122治疗丙型肝炎的先河.
相对于锁核酸标记的寡核苷酸成本高、易降解的特点, CRISPR干扰(CRISPR interference, CRISPRi)技术具有成本低、易操作、高效等特点. 本研究首次利用CRISPRi技术实现了HepG2细胞内miR-122的特异性表达抑制, 在抗HCV病毒方面具有更加广阔的应用前景.
CRISPRi通过靶向miR-122抑制HCV病毒的复制, 为分子靶向药物治疗丙型肝炎提供了理论基础.
CRISPRi: 天然CRISPR相关基因9(CRISPR-associated gene 9, Cas9)含有2种核酸酶结构域: RuvC1结构域及HNH结构域, HNH结构域切割与crRNA互补的模板链, RuvC1结构域切割非互补链. 将Cas9的核酸内切酶结构域突变(RuvC1, D10A; HNH, H841A)产生dCas9, 仅保留DNA识别能力而无内切酶活性, 当dCas9与sgRNA共表达时, 形成识别特定位点的复合体, 该复合体可以特异性的干扰RNA聚合酶或者转录因子与DNA序列的结合, 有效抑制靶分子的表达, 将此过程称为CRISPRi.
本研究利用了CRISPRi技术靶向抑制肝癌细胞与HCV病毒复制相关小RNAmiR-122, 进而上调抑制HCV复制相关的miR-122下游靶分子HMOX1和CyclinG1 mRNA和蛋白表达, 为分子靶向药物治疗HCV肝炎提供了理论基础.
编辑:韦元涛 电编:都珍珍
| 1. | Poordad F, McCone J, Bacon BR, Bruno S, Manns MP, Sulkowski MS, Jacobson IM, Reddy KR, Goodman ZD, Boparai N. Boceprevir for untreated chronic HCV genotype 1 infection. N Engl J Med. 2011;364:1195-1206. [PubMed] [DOI] |
| 2. | Sherman KE, Flamm SL, Afdhal NH, Nelson DR, Sulkowski MS, Everson GT, Fried MW, Adler M, Reesink HW, Martin M. Response-guided telaprevir combination treatment for hepatitis C virus infection. N Engl J Med. 2011;365:1014-1024. [PubMed] [DOI] |
| 3. | Bandiera S, Pfeffer S, Baumert TF, Zeisel MB. miR-122--a key factor and therapeutic target in liver disease. J Hepatol. 2015;62:448-457. [PubMed] [DOI] |
| 5. | Henke JI, Goergen D, Zheng J, Song Y, Schüttler CG, Fehr C, Jünemann C, Niepmann M. microRNA-122 stimulates translation of hepatitis C virus RNA. EMBO J. 2008;27:3300-3310. [PubMed] [DOI] |
| 6. | Jopling CL, Yi M, Lancaster AM, Lemon SM, Sarnow P. Modulation of hepatitis C virus RNA abundance by a liver-specific MicroRNA. Science. 2005;309:1577-1581. [PubMed] [DOI] |
| 7. | Machlin ES, Sarnow P, Sagan SM. Masking the 5' terminal nucleotides of the hepatitis C virus genome by an unconventional microRNA-target RNA complex. Proc Natl Acad Sci U S A. 2011;108:3193-3198. [PubMed] [DOI] |
| 8. | Jinek M, Chylinski K, Fonfara I, Hauer M, Doudna JA, Charpentier E. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 2012;337:816-821. [PubMed] [DOI] |
| 9. | Doudna JA, Charpentier E. Genome editing. The new frontier of genome engineering with CRISPR-Cas9. Science. 2014;346:1258096. [PubMed] [DOI] |
| 10. | Qi LS, Larson MH, Gilbert LA, Doudna JA, Weissman JS, Arkin AP, Lim WA. Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression. Cell. 2013;152:1173-1183. [PubMed] [DOI] |
| 11. | Shi R, Chiang VL. Facile means for quantifying microRNA expression by real-time PCR. Biotechniques. 2005;39:519-525. [PubMed] [DOI] |
| 12. | Carlsbecker A, Lee JY, Roberts CJ, Dettmer J, Lehesranta S, Zhou J, Lindgren O, Moreno-Risueno MA, Vatén A, Thitamadee S. Cell signalling by microRNA165/6 directs gene dose-dependent root cell fate. Nature. 2010;465:316-321. [PubMed] [DOI] |
| 13. | Shan Y, Zheng J, Lambrecht RW, Bonkovsky HL. Reciprocal effects of micro-RNA-122 on expression of heme oxygenase-1 and hepatitis C virus genes in human hepatocytes. Gastroenterology. 2007;133:1166-1174. [PubMed] [DOI] |
| 14. | Hou W, Bukong TN, Kodys K, Szabo G. Alcohol facilitates HCV RNA replication via up-regulation of miR-122 expression and inhibition of cyclin G1 in human hepatoma cells. Alcohol Clin Exp Res. 2013;37:599-608. [PubMed] [DOI] |
| 15. | Pedersen IM, Cheng G, Wieland S, Volinia S, Croce CM, Chisari FV, David M. Interferon modulation of cellular microRNAs as an antiviral mechanism. Nature. 2007;449:919-922. [PubMed] [DOI] |
| 16. | Jopling CL, Schütz S, Sarnow P. Position-dependent function for a tandem microRNA miR-122-binding site located in the hepatitis C virus RNA genome. Cell Host Microbe. 2008;4:77-85. [PubMed] [DOI] |
| 17. | Lanford RE, Hildebrandt-Eriksen ES, Petri A, Persson R, Lindow M, Munk ME, Kauppinen S, Ørum H. Therapeutic silencing of microRNA-122 in primates with chronic hepatitis C virus infection. Science. 2010;327:198-201. [PubMed] [DOI] |