修回日期: 2006-02-07
接受日期: 2006-02-21
在线出版日期: 2006-03-28
聚腺苷酸二磷酸核糖转移酶-1[poly (ADP-ribose) polymerase-1, PARP-1]是存在于真核细胞中催化聚ADP核糖化的细胞核酶, 他参与的聚ADP核糖化是真核细胞中蛋白质翻译后的重要修饰方式之一. PARP-1在DNA修复和细胞凋亡中发挥至关重要的作用. PARP-1的缺失使细胞对DNA损伤因子易感, 可能参与肿瘤的发生. 体外和体内研究表明抑制PARP-1则可降低DNA修复功能, 增强放疗和化疗对肿瘤的治疗效果. 目前PARP-1抑制剂已进入抗肿瘤药物Ⅰ期临床研究, PARP-1有望成为肿瘤治疗的一个新靶点.
引文著录: 黄胜辉, 黄志勇. PARP-1: 一个肿瘤治疗的新靶点. 世界华人消化杂志 2006; 14(9): 841-847
Revised: February 7, 2006
Accepted: February 21, 2006
Published online: March 28, 2006
N/A
- Citation: N/A. N/A. Shijie Huaren Xiaohua Zazhi 2006; 14(9): 841-847
- URL: https://www.wjgnet.com/1009-3079/full/v14/i9/841.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v14.i9.841
聚腺苷酸二磷酸核糖转移酶[poly(ADP-ribose) polymerase, PARP]是存在于真核细胞中催化聚ADP核糖化的细胞核酶. 1963年法国斯特拉斯堡Mandel小组发现肝细胞核中尼克酰胺单核苷酸(NMN)促进已标记的ATP合成一种难溶于酸的片段. Sugimura et al推测该片段是一种小分子代谢物合成的高聚物, 于1966年证实为聚腺苷酸二磷酸核糖, 并从肝细胞核中提取一种新的酶, 即PARP(Masutani et al[1]提及). PARP自发现40多年来, 其在DNA损伤修复和维持基因组的稳定性方面的重要作用引起广大学者的关注, 尤其是近年来对PARP与肿瘤发生的关系及利用调节PARP来提高肿瘤治疗效果的研究取得了令人鼓舞的成绩. 而在PARP家族成员中以PARP-1结构最典型,研究较多.
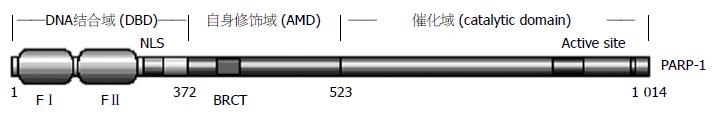
PARP-1是一种分子质量116 ku的核酶, 他包括DNA结合域(DNA-binding domain, DBD), 自身修饰域(the automodification domain, AMD)和C-端催化域(图1). 其中DBD分子质量为46 ku, 他含有2个锌指结构(FⅠ和FⅡ)及核定位信号(nuclear localization signal, NLS), 可以结合到DNA单链或双链缺口, NLS中含有凋亡相关的DEVD模体; AMD含有15个保守的谷氨酸残基作为自身聚ADP核糖基化靶位, 含有与DNA修复有关的乳腺癌感受蛋白C端模序(BRCT); C-端催化域分子质量为54 ku, 含有NAD结合位点, 在PARP家族中高度保守[2-3].
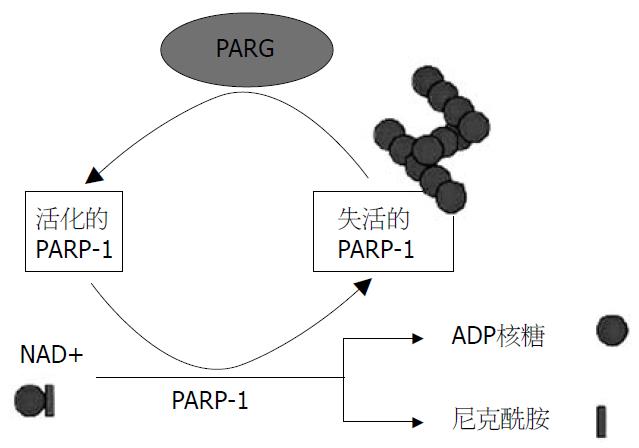
PARP-1主要参与细胞内聚腺苷酸二磷酸核糖(PAR)的代谢. 识别DNA缺口后活化的PARP-1形成同型二聚体并催化尼克酰二核苷酸(NAD)降解为烟酸和ADP, 然后以ADP为底物在受体蛋白(包括自身)上合成聚腺苷酸二磷酸核糖基(PAR), 从而改变其功能. 这些受体蛋白包括PARP-1自身、组蛋白、各种转录因子、DNA修复子以及信号转导子(包括NF-κB, AP-2, Oct-1, YY1, B-MYB, DNA依赖型蛋白激酶, P53等)等[3]. 而PAR的分解依赖于2种酶: 聚ADP核糖水解酶[poly (ADP-ribose) glycohydrolase, PARG]和裂解酶. PARG可以清除PAR线状和分枝的核糖与核糖基, 而裂解酶则裂解最近端的单ADP核糖基, 裂解后的ADP核糖可以与烟酸再合成NAD, 实现再利用[2,4]. PARG可裂解PARP-1上的聚ADP核糖基使其从DNA缺口上分离, 有利于PARP-1活性恢复(图2). PARP与PARG的活性平衡维持着细胞内聚ADP核糖基化的水平, 从而保持受体蛋白活性的稳定.
PARP-1参与DNA缺口的识别. DNA缺口主要包括单链DNA缺口(single-strand break, SSB)和双链DNA缺口(double-strand break, DSB). PARP-1作为DNA缺口感受器(DNA nick sensor), 依赖N-端DBD的两个锌指模序结合到单链DNA缺口(SSB)或双链DNA缺口(DSB). 其中, 锌指模序Ⅰ是SSB和DSB依赖的PARP-1活化所必需的, 而锌指模序Ⅱ主要在SSB依赖的PARP-1活化中发挥作用. 而锌指模序下游的氨基酸残基与非特异DNA结合有关[1,5]. 识别DNA缺口后活化的PARP-1形成同型二聚体并催化NAD分解成尼克酰胺和ADP核糖, 利用后者为原料使核受体蛋白聚ADP核糖化, 从而影响这些修复相关蛋白的功能. PARP-1可参与DNA修复过程. DNA的修复主要有碱基切除修复, 核苷酸切除修复, 错配修复, 重组修复和非同源末端连接, 其中单链DNA缺口(SSB)的碱基切除修复(BER)中聚ADP核糖化起着重要作用. Haince et al[6-7]认为, PARP-1参与SSB修复有两个主要机制: 首先, PARP-1自身和组蛋白的聚ADP核糖化加速他们与DNA链分离, 从而促进染色质的松弛, 以利于修复蛋白在缺口处的集结. PARP-1自身以及组蛋白的聚ADP核糖化改变了其电荷从而促使其与DNA链排斥分离. 其次, 聚ADP核糖(PAR)和聚ADP核糖化的PARP-1促进X线修复交叉互补组合-1(X-ray repair cross-complementing group 1, XRCC1)和DNA连接酶Ⅲ募集到SSBs以及DNA聚合酶beta的活化[8-10]. 另外, PAR可能与募集的蛋白中特异性PAR结合模序非共价结合从而改变了这些蛋白的功能[11].
双链DNA缺口(DSB)是一种致命的DNA损伤, 可以由电离辐射, 化学物质造成, 亦可由于DNA复制叉的延伸停滞所致. Susse et al[12]发现, 全长PARP-1或仅DNA结合域的短暂过度表达可促使DSB修复下调, 不依赖PARP的酶活性, 并与P53在抑制染色体重排方面起协同作用. 哺乳动物细胞的DSB修复主要利用两条修复旁路进行: 一是同源重组(homologous recombination, HR), 另一种是非同源末端连接(nonhomologous end-joining, NHEJ). 许多研究发现PARP-1可能影响HR. PARP-1基因缺失或活性抑制的小鼠容易发生姐妹染色单体交换(SCE)和基因扩增(gene amplification), 而SCE和基因扩增被认为是同源重组引起的, 这表明PARP-1可能影响同源重组(HR)[13-15]. Dominguez et al[15]发现PARP-1抑制剂3-AB可以促使BrdU(5-溴脱氧尿苷)处理后的中国仓鼠卵巢细胞姐妹染色单体交换(SCE)频率增加, 但并不同步增加易位的频率. Waldman et al[16]研究发现, 抑制小鼠成纤维细胞PARP-1活性可影响染色体内HR, 但并不影响染色体外HR. 而Schultz et al[14]抑制中国仓鼠卵巢细胞PARP-1并未见HR频率改变. PARP-1的作用究竟是使引发HR的DNA损伤数量改变还是直接参与影响HR曾存在争议. 同源重组需要一组叫RAD52的族蛋白, 包括Rad51, Rad52, Rad54, BRCA1和BRCA2. 这些蛋白聚集在DNA损伤处形成亚核结构称为聚集点(foci), 其中Rad51是foci动态结构的相关核心组分[17]. 绝大多数参与同源重组的蛋白位于Rad51聚集点, 而同源重组缺陷的细胞不能形成Rad51聚集点. 然而Schultz et al[14]发现, PARP-1-/-细胞仍可形成Rad51聚集点, 故推测PARP-1主要并不位于Rad51聚集点. PARP-1抑制或缺失可以增加自发的Rad51聚集点的形成, 而在PARP-1抑制的细胞中, 针对位点特异性DSB的同源定向修复正常. 因此, 目前认为PARP-1控制着HR识别的DNA损伤而非直接影响HR的执行.
DSB的非同源末端连接(NHEJ)修复机制主要依赖Ku70/80异二聚体, Ku70/80可能作为DNA锚定亚单位与DNA依赖的蛋白激酶催化亚单位(DNA-PKcs)结合形成DNA-PK全酶, 后者募集XRCC4和DNA连接酶Ⅳ完成NHEJ修复. 在NHEJ方面, PARP-1可能通过紧密结合到DSB与DNA-PKcs或Ku70/80相互作用[18]. Tong et al[19]亦发现, PARP-1与Ku80可能存在一定的协同作用. 另外, Audebert et al[20]认为, PARP-1与XRCC1/DNA连接酶Ⅰ亦可通过不依赖于DNA-PKcs, XRCC4和连接酶Ⅳ的途径参与NHEJ.
细胞凋亡过程中, 半胱-天冬蛋白酶Casepase-7和Casepase-3可以通过识别PARP-1核定位信号(NLS)的DEVD模序, 促使DNA结合域与催化域分离导致PARP-1分裂成p89和p24两个片段, 后者可不可逆结合于DNA缺口, 抑制PARP-1与DNA缺口结合能力, 导致PARP-1损伤修复作用丧失, 诱发凋亡[21]. Casepase介导PARP-1失活的正反馈控制环存在表明PARP-1的活性阻滞对于维持正常细胞凋亡至关重要. 另外, PARP-1在凋亡诱导因子(AIF)介导的半胱-天冬蛋白酶非依赖型的细胞凋亡中发挥重要作用[21-22]. 然而缺乏PARP基因的小鼠表型正常, 仅在老年时易发生表皮增生和肥胖. PARP-1-/-小鼠表现出正常DNA切除修复、V(D)J重组和免疫球蛋白转型, 但对大剂量的g射线高度敏感, 表现出姐妹染色体交换频率增加和DNA破坏诱导的微核形成. 缺乏PARP基因的细胞在给予TNF-a, g射线和地塞米松仍可以发生凋亡. Wang et al[23-24]因此推测PARP-1在多数凋亡中是可有可无的. 尽管如此, 许多学者仍认为PARP-1的裂解在调亡机制中发挥着重要作用.
PARP-1在维持基因组的稳定性方面至关重要, 而PARP-1的抑制可能使机体细胞对烷化剂, 电离辐射等DNA损伤因子易感, 可能导致更容易发生肿瘤. 目前PARP-1的抑制模型主要有三种: 一是PARP-1基因敲除: 这种方式缺乏PARP-1结合的SSB是暴露的, SSB可形成停止复制叉, 推测SSB可能通过非PARP-1介导的修复机制参与修复. 二是利用抑制剂抑制PARP-1活性, 这阻碍PARP-1从单链DNA缺口(SSB)分离, 阻碍了BER修复蛋白募集到SSB上, 因此在细胞分裂S期未修复的SSB的复制叉将停滞. 这种停滞复制叉有赖于重组修复. 三是PARP-1基因部分损耗(depletion): 利用siRNA干扰PARP-1, 从而改变PARP和PARG的平衡, PARG活性相对增强因而使PAR降解过多. 这种作用相当于PARP抑制剂[7]. 这三种途径在肿瘤发生和治疗方面研究以前两种应用较多. 许多研究发现, PARP-1的基因敲除的小鼠经其他的DNA损伤因子处理后可导致肿瘤发生, 而单纯PARP-1缺失的小鼠观察至9月龄却未发现任何器官发生肿瘤[25]. Morrison et al[26]发现PARP-1-/-SCID小鼠胸腺淋巴瘤发生率明显高于PARP-1+/+SCID组. Tsutsumi et al[27]通过破坏PARP-1外显子1产生PARP-1-/-小鼠, 然后经BHP(N-二亚硝基-2-羟丙胺)处理后, 其肝血管瘤, 血管肉瘤和肺腺瘤发生率明显高于PARP-1+/+组. Nozaki et al[25]通过破坏PARP外显子1产生PARP-1-/-小鼠, 给予AOM(氧化偶氮甲烷)后PARP-1-/-小鼠结肠腺瘤和腺癌、肝脏结节发生率均明显高于PARP+/+组, 然而从出生至9月龄的PARP-1-/-小鼠各器官并未发生自发的肿瘤. 而Conde et al[28]发现, 破坏PARP-1外显子4所产生的PARP-1和p53共缺失(PARP-1-/- p53-/-)的小鼠和单纯p53完全缺失组鼠(PARP-1+/+ p53-/-)几乎都死于肿瘤(主要是淋巴瘤, 肉瘤, 表皮肿瘤和脑肿瘤), 然而前组无瘤生存率却明显高于后组. Tong et al[29-30]发现PARP-1无效性突变(null mutation)会使p53缺失的小鼠在8周龄时髓母细胞瘤发生率增高, 破坏PARP-1外显子2产生的PARP-1-/- p53-/-小鼠淋巴瘤、乳腺和结肠肿瘤发生率明显增高. Dandri et al[31]发现PARP抑制剂可以明显增加过氧化氢诱导HepG2肝癌细胞的HBV整合, 提示PARP-1可以限制HBV整合到宿主DNA, 而HBV-DNA整合是肝癌的高危因素之一, 这似乎表明PARP-1与HBV相关的肝癌发生存在一定联系. 而Nomura et al[32]发现, 人肝癌组织中PARP-1比邻近非肿瘤组织表达增高且活性增强, 其具体机制尚待进一步阐明. Tong et al[19]研究发现, PARP-1-/- Ku80-/-小鼠约在胚胎至少8.5 d之前即发生生长迟滞, 并于9.5 d死亡, 提示PARP-1和Ku80双缺失对小鼠是致命的. 而且在PARP-1-/-小鼠中Ku80的单个等位基因缺失将增加肝腺瘤和肝癌的发生率, 可见PARP-1与参与细胞NHEJ机制的Ku80可能存在一定的协同作用.
从分子水平而言, 恶性肿瘤的治疗策略多数是利用损伤肿瘤细胞DNA的方法, 如放疗和化疗等, 来杀伤肿瘤细胞. 因此针对参与DNA损伤识别、反应和修复的分子靶向治疗探索一直是近年来研究的热点. PARP-1在DNA修复、细胞死亡、增殖分化等方面发挥着重要作用, 推测通过抑制PARP-1活性可抑制PARP-1介导的DNA修复机制, 提高放疗和化疗对肿瘤细胞DNA的损伤, 因而对肿瘤可能具有潜在治疗价值. 目前已有多种PARP-1抑制剂应用于临床药物研究, 例如已开发的NU1025, GPI15427, AG014699, CEP-6800, AG14361, INO1001等. 无论从分子水平、细胞水平, 还是从整体水平, 这些PARP-1抑制剂在联合化疗、放疗或单独化疗方面的治疗研究都取得一些重要进展. 许多研究发现PARP-1抑制剂可加强多种抗肿瘤药(如烷化剂、拓扑异构酶抑制剂)的疗效. Tentori et al[33]发现NU1025可延长脑淋巴瘤荷瘤小鼠的生存期. Delaney et al[34]采用12个人类肿瘤细胞株(包括肺, 结肠, 卵巢, 乳腺的肿瘤细胞系各3个), 发现PARP-1抑制剂NU1025或NU1085与抗肿瘤药temozolomide(TMZ, 烷化剂)或topotecan(TP, 喜树碱类)联用可增强后二者对瘤细胞的生长抑制作用, NU1025和NU1085的抑瘤增强作用与二者对PARP的抑制强度有关. Guilford公司开发的PARP-1抑制剂GPI15427可增强TMZ的作用, 明显延长多形性胶质母细胞瘤、脑淋巴瘤或颅内恶性黑色素瘤荷瘤小鼠的生存期[35]. Calabrese et al[36]研究皮下移植结肠癌细胞的小鼠发现, AG14361会增强TMZ的抗肿瘤作用. Miknyoczki et al[37]用PARP-1抑制剂CEP-6800分别与TMZ、CPT-11(喜树碱类)或顺铂联用治疗临床相应肿瘤(U251MG人类胶质母细胞瘤, HT29人类结肠腺癌和Calu-6非小细胞肺癌)的异种移植模型, 研究表明CEP-6800可分别使TMZ、喜树碱或顺铂处理后的肿瘤细胞停滞于G2/M期的时间和/或比例增加, 并可增强TMZ、喜树碱和顺铂对荷瘤裸鼠的治疗效果. 尽管PARP-1抑制剂联合化疗的研究表明抗肿瘤作用显著增强, 但其安全性和可行性仍然是人们担忧的一个问题. Miknyoczki et al[37]的研究发现, 治疗剂量的CEP-6800并未增加这三种化疗药对离体的人类肠上皮和肾脏细胞造成损害, 也不增加活体小鼠中TMZ的胃肠毒性和顺铂的肾脏毒性, 这预示着CEP-6800联合化疗的价值和可行性. 辉瑞公司开发的一种新型PARP-1抑制剂AG014699有望与烷化剂TMZ联合治疗恶性黑色素瘤, 该药正处于Ⅰ期临床研究阶段[38]. 另外PARP-1抑制剂GPI 15427由于半衰期长、生物利用度高, 通过口服给药局部可达到有效药物浓度, 在与TMZ联用治疗的中枢神经系统肿瘤模型取得较好疗效[39]. Ku-DOS公司也宣布一种口服PARP抑制剂作为抗肿瘤药物(主要针对遗传性乳腺癌和卵巢癌)进入Ⅰ期临床研究阶段[40].
PARP-1抑制剂的另一方面研究在于是否加强放疗的抗肿瘤效应. 肿瘤细胞对放疗的抵抗依赖于酶介导的双链DNA缺口(DSB)修复机制, 尤其是NHEJ修复机制. 后者依赖于DNA-PK的作用, 而已有研究表明PARP-1与DNA-PK之间存在相互作用[18-19], 这一定程度上为PARP-1抑制剂联合放疗的应用研究提供了理论基础. Veuger et al[41]利用小鼠胚胎成纤维细胞和中国仓鼠卵巢细胞研究发现, PARP-1或DNA-PK缺失均可使相应细胞对放射的敏感性提高到4倍, 并降低G0期细胞的潜在致死损伤修复(PLDR). PARP-1特异性抑制剂AG14361和DNA-PK特异性抑制剂NU7026可分别使PARP-1和DNA-PK无缺陷的细胞对放射的敏感性增加, 二者联用具有加和作用. AG14361或NU7026单用分别可使非缺陷细胞的PLDR降至1/3, 二者联用可完全阻止PLDR. 联用AG14361或NU7026后, 即使在放射后24 h仍可阻止90%的DNA-DSB重新结合. 这种对放射介导的DNA-DSB修复抑制作用与其细胞毒作用加强相关. Calabrese et al[36]在活体实验中发现AG14361可增强皮下移植结肠癌细胞的小鼠对局部放疗的敏感性. Brock et al[42]发现PARP-1抑制剂INO1001可提高人类成纤维细胞对放射线的敏感性. 这些预示着PARP-1抑制剂单独应用或与其他抗肿瘤药联合可能有利于提高肿瘤对放疗的敏感性.
此外, 单独抑制PARP-1在肿瘤治疗(主要是乳腺癌)研究方面取得了一定进展. 乳腺癌易感基因BRCA1和BRCA2在DNA同源重组(HR)、单核苷酸切除修复(NER)以及可能在非同源末端连接(NHEJ)中发挥重要作用[43]. 家族性乳腺癌多数由于BRCA1或BRCA2的一个等位基因遗传缺陷引起, 某些杂合子细胞由于残留的一个BRCA1或BRCA2等位基因缺失可能启动了肿瘤的发生. BRCA缺陷细胞(系指BRCA1-/-或BRCA2-/-细胞)同源重组(HR)缺陷, 抑制PARP-1可使正常情况下依赖姐妹染色单体交换(SCE)修复的DNA损伤(lesion)产生增多, 这些损伤在同源重组(HR)缺陷细胞中积聚可导致染色质失常甚至细胞死亡. Farmer et al[44]通过干扰PARP-1的RNA发现, BRCA1缺陷的小鼠胚胎干细胞株比BRCA1正常的野生株克隆存活率低. 类似结果见于BRCA2缺陷的中国仓鼠卵巢细胞, 以及BRCA1 mRNA损耗的人乳腺癌细胞株MCF7中. 同样, 利用PARP-1高效选择抑制剂KU0058684和KU0058948分别作用于野生株、BRCA1缺陷株和BRCA2缺陷株, 发现后两株的敏感性明显高于野生株. 而由于KU0051529不能有效抑制PARP-1和PARP-2, 其作用后三组敏感性无明显差异. KU0058684作用24 h后的细胞多数停滞在G2期. 尽管BRCA缺陷细胞对PARP-1高效抑制剂高度敏感, 杂合子的BRCA1突变细胞和杂合子BRCA2突变细胞均无此效应. Bryant et al[45]将人类乳腺癌细胞系MCF7野生株和MDA-MB-231(p53突变株)分别经BRCA2-siRNA处理, 发现两株细胞对PARP抑制剂NU1025的敏感性无差异, 但NU1025仅使BRCA2-siRNA处理后的两株细胞生存率明显下降. 利用RNA干扰同时造成BRCA2和PARP-1损耗可使细胞生存率明显降低. 将BRCA2-/-细胞和BRCA2+/-细胞移植入裸鼠股部, 用PARP-1抑制剂AG14361作用于两组后, 发现BRCA2-/-组鼠相对BRCA2+/-组鼠肿瘤生长明显受抑制. BRCA2-/-细胞由于同源重组缺陷, 对PARP-1抑制剂特别敏感, 因此在BRCA2+/-患者的肿瘤治疗中(肿瘤组织为BRCA2-/-), PARP-1抑制剂极有可能实现肿瘤特异性治疗. 最近研究表明在同源重组(HR)遗传缺陷细胞中PARP-1是必需的, 而且似乎并不局限于缺陷的是参与HR的某一种具体分子, 因此抑制PARP-1可能为HR缺陷的肿瘤的治疗提供了新的前景[7,44-45].
总之, 对PAPR-1的研究一定程度上推动了DNA修复、细胞凋亡、增殖分化的机制研究, 有助于我们进一步探索肿瘤的发生机制和治疗策略. 将来恶性肿瘤治疗策略是对肿瘤细胞的特异杀伤, 而对正常细胞DNA影响甚微的靶向治疗, PARP-1有望成为有效而特异的抗肿瘤靶分子之一. 今后PARP-1的研究大体有两个方向: 一是对PARP-1在肿瘤和其他疾病的分子机制进一步广泛和深入的研究; 另一方面是PARP-1干预手段的探索和临床应用, 包括高效特异而安全的PARP-1抑制剂的研制开发和RNA干扰等技术的应用. 相信随着PARP-1研究的进展, 以及联合其他抗肿瘤治疗手段的进步, 针对PARP-1的靶向干预在肿瘤防治中必将发挥重要的作用.
PARP-1是真核细胞中催化聚ADP核糖化的一种细胞核酶. 自发现40多年来, PARP-1在DNA修复、细胞凋亡、增殖分化等方面的作用机制引起广大学者的关注, 并逐渐得到阐明. 近年来对PARP-1与肿瘤发生的关系以及利用调节PARP-1来提高肿瘤治疗效果的研究取得了令人鼓舞的成绩. 目前PARP-1抑制剂已进入抗肿瘤药物Ⅰ期临床研究.
本文的内容较为详实, 从PARP-1与肿瘤的各方面联系阐述了其与肿瘤发生与治疗的关系, 有一定的指导意义.
电编:张敏 编辑:潘伯荣
| 1. | Masutani M, Nakagama H, Sugimura T. Poly(ADP-ribose) and carcinogenesis. Genes Chromosomes Cancer. 2003;38:339-348. [PubMed] [DOI] |
| 2. | Nguewa PA, Fuertes MA, Valladares B, Alonso C, Perez JM. Poly(ADP-ribose) polymerases: homology, structural domains and functions. Novel therapeutical applications. Prog Biophys Mol Biol. 2005;88:143-172. [PubMed] [DOI] |
| 3. | Virag L, Szabo C. The therapeutic potential of poly(ADP-ribose) polymerase inhibitors. Pharmacol Rev. 2002;54:375-429. [PubMed] [DOI] |
| 4. | Davidovic L, Vodenicharov M, Affar EB, Poirier GG. Importance of poly(ADP-ribose) glycohydrolase in the control of poly (ADP-ribose) metabolism. Exp Cell Res. 2001;268:7-13. [PubMed] [DOI] |
| 5. | Ikejima M, Noguchi S, Yamashita R, Ogura T, Sugimura T, Gill DM, Miwa M. The zinc fingers of human poly(ADP-ribose) polymerase are differen-tially required for the recognition of DNA breaks and nicks and the consequent enzyme activation. Other structures recognize intact DNA. J Biol Chem. 1990;265:21907-21913. [PubMed] |
| 6. | Rouleau M, Aubin RA, Poirier GG. Poly(ADP-ribosyl)ated chromatin domains: access granted. J Cell Sci. 2004;117:815-825. [PubMed] [DOI] |
| 7. | Haince JF, Rouleau M, Hendzel MJ, Masson JY, Poirier GG. Targeting poly(ADP-ribosyl)ation: a promising approach in cancer therapy. Trends Mol Med. 2005;11:456-463. [PubMed] [DOI] |
| 8. | Leppard JB, Dong Z, Mackey ZB, Tomkinson AE. Physical and functional interaction between DNA ligase IIIalpha and poly (ADP-Ribose) polymerase 1 in DNA single-strand break repair. Mol Cell Biol. 2003;23:5919-5927. [PubMed] [DOI] |
| 9. | El-Khamisy SF, Masutani M, Suzuki H, Caldecott KW. A requirement for PARP-1 for the assembly or stability of XRCC1 nuclear foci at sites of oxidative DNA damage. Nucleic Acids Res. 2003;31:5526-5533. [PubMed] [DOI] |
| 10. | Prasad R, Lavrik OI, Kim SJ, Kedar P, Yang XP, Vande Berg BJ, Wilson SH. DNA polymerase beta -mediated long patch base excision repair. Poly(ADP-ribose)polymerase-1 stimulates strand displacement DNA synthesis. J Biol Chem. 2001;276:32411-32414. [PubMed] [DOI] |
| 11. | Pleschke JM, Kleczkowska HE, Strohm M, Althaus FR. Poly(ADP-ribose) binds to specific domains in DNA damage checkpoint proteins. J Biol Chem. 2000;275:40974-40980. [PubMed] [DOI] |
| 12. | Susse S, Scholz CJ, Burkle A, Wiesmuller L. Poly(ADP-ribose) polymerase (PARP-1) and p53 independently function in regulating double-strand break repair in primate cells. Nucleic Acids Res. 2004;32:669-680. [PubMed] [DOI] |
| 13. | de Murcia JM, Niedergang C, Trucco C, Ricoul M, Dutrillaux B, Mark M, Oliver FJ, Masson M, Dierich A, LeMeur M. Requirement of poly(ADP-ribose) polymerase in recovery from DNA damage in mice and in cells. Proc Natl Acad Sci USA. 1997;94:7303-7307. [PubMed] [DOI] |
| 14. | Schultz N, Lopez E, Saleh-Gohari N, Helleday T. Poly(ADP-ribose) polymerase (PARP-1) has a controlling role in homologous recombination. Nucleic Acids Res. 2003;31:4959-4964. [PubMed] [DOI] |
| 15. | Dominguez I, Mateos S, Cortes F. Yield of SCEs and translocations produced by 3 aminobenzamide in cultured Chinese hamster cells. Mutat Res. 2000;448:29-34. [PubMed] [DOI] |
| 16. | Waldman AS, Waldman BC. Stimulation of intra-chromosomal homologous recombination in mam-malian cells by an inhibitor of poly(ADP-ribos-ylation). Nucleic Acids Res. 1991;19:5943-5947. [PubMed] [DOI] |
| 17. | Essers J, Houtsmuller AB, van Veelen L, Paulusma C, Nigg AL, Pastink A, Vermeulen W, Hoeijmakers JH, Kanaar R. Nuclear dynamics of RAD52 group homologous recombination proteins in response to DNA damage. EMBO J. 2002;21:2030-2037. [PubMed] |
| 18. | Jackson SP. Sensing and repairing DNA double-strand breaks. Carcinogenesis. 2002;23:687-696. [PubMed] [DOI] |
| 19. | Tong WM, Cortes U, Hande MP, Ohgaki H, Cavalli LR, Lansdorp PM, Haddad BR, Wang ZQ. Synergistic role of Ku80 and poly(ADP-ribose) polymerase in suppressing chromosomal aberra-tions and liver cancer formation. Cancer Res. 2002;62:6990-6996. [PubMed] |
| 20. | Audebert M, Salles B, Calsou P. Involvement of poly(ADP-ribose) polymerase-1 and XRCC1/DNA ligase III in an alternative route for DNA double-strand breaks rejoining. J Biol Chem. 2004;279:55117-55126. [PubMed] [DOI] |
| 21. | Koh DW, Dawson TM, Dawson VL. Mediation of cell death by poly(ADP-ribose) polymerase-1. Pharmacol Res. 2005;52:5-14. [PubMed] [DOI] |
| 22. | Yu SW, Wang H, Poitras MF, Coombs C, Bowers WJ, Federoff HJ, Poirier GG, Dawson TM, Dawson VL. Mediation of poly (ADP-ribose) polymerase-1-dependent cell death by apoptosis-inducing factor. Science. 2002;297:259-263. [PubMed] [DOI] |
| 23. | Wang ZQ, Auer B, Stingl L, Berghammer H, Haidacher D, Schweiger M, Wagner EF. Mice lacking ADPRT and poly(ADP- ribosyl) ation deve-lop normally but are susceptible to skin disease. Genes Dev. 1995;9:509-520. [PubMed] [DOI] |
| 24. | Wang ZQ, Stingl L, Morrison C, Jantsch M, Los M, Schulze-Osthoff K, Wagner EF. PARP is important for genomic stability but dispensable in apoptosis. Genes Dev. 1997;11:2347-2358. [PubMed] [DOI] |
| 25. | Nozaki T, Fujihara H, Watanabe M, Tsutsumi M, Nakamoto K, Kusuoka O, Kamada N, Suzuki H, Nakagama H, Sugimura T. Parp-1 deficiency implicated in colon and liver tumorigen-esis induced by azoxymethane. Cancer Sci. 2003;94:497-500. [PubMed] [DOI] |
| 26. | Morrison C, Smith GC, Stingl L, Jackson SP, Wagner EF, Wang ZQ. Genetic interaction between PARP and DNA-PK in V(D)J recombination and tumorigenesis. Nat Genet. 1997;17:479-482. [PubMed] [DOI] |
| 27. | Tsutsumi M, Masutani M, Nozaki T, Kusuoka O, Tsujiuchi T, Nakagama H, Suzuki H, Konishi Y, Sugimura T. Increased susceptibility of poly(ADP-ribose) polymerase-1 knockout mice to nitrosamine carcinogenicity. Carcinogenesis. 2001;22:1-3. [PubMed] [DOI] |
| 28. | Conde C, Mark M, Oliver FJ, Huber A, de Murcia G, Menissier-de Murcia J. Loss of poly(ADP-ribose) polymerase-1 causes increased tumour latency in p53-deficient mice. EMBO J. 2001;20:3535-3543. [PubMed] [DOI] |
| 29. | Tong WM, Ohgaki H, Huang H, Granier C, Kleihues P, Wang ZQ. Null mutation of DNA strand break-binding molecule poly (ADP-ribose) polymerase causes medulloblastomas in p53(-/-) mice. Am J Pathol. 2003;162:343-352. [PubMed] [DOI] |
| 30. | Tong WM, Hande MP, Lansdorp PM, Wang ZQ. DNA strand break-sensing molecule poly(ADP-Ribose) polymerase cooperates with p53 in telo-mere function, chromosome stability, and tumor suppression. Mol Cell Biol. 2001;21:4046-4054. [PubMed] [DOI] |
| 31. | Dandri M, Burda MR, Burkle A, Zuckerman DM, Will H, Rogler CE, Greten H, Petersen J. Increase in de novo HBV DNA integrations in response to oxidative DNA damage or inhibition of poly(ADP-ribosyl)ation. Hepatology. 2002;35:217-223. [PubMed] [DOI] |
| 32. | Nomura F, Yaguchi M, Togawa A, Miyazaki M, Isobe K, Miyake M, Noda M, Nakai T. Enhancement of poly-adenosine diphosphate-ribosylation in human hepatocellular carcinoma. J Gastroenterol Hepatol. 2000;15:529-535. [PubMed] [DOI] |
| 33. | Tentori L, Leonetti C, Scarsella M, d'Amati G, Portarena I, Zupi G, Bonmassar E, Graziani G. Combined treatment with temozolomide and poly(ADP-ribose) polymerase inhibitor enhances survival of mice bearing hematologic malignancy at the central nervous system site. Blood. 2002;99:2241-2244. [PubMed] [DOI] |
| 34. | Delaney CA, Wang LZ, Kyle S, White AW, Calvert AH, Curtin NJ, Durkacz BW, Hostomsky Z, Newell DR. Potentiation of temozolomide and topotecan growth inhibition and cytotoxicity by novel poly (adenosine diphosphoribose) polymerase inhibitors in a panel of human tumor cell lines. Clin Cancer Res. 2000;6:2860-2867. [PubMed] |
| 35. | Tentori L, Leonetti C, Scarsella M, D'Amati G, Vergati M, Portarena I, Xu W, Kalish V, Zupi G, Zhang J. Systemic administration of GPI 15427, a novel poly(ADP-ribose) polymerase-1 inhibitor, increases the antitumor activity of temo-zolomide against intracranial melanoma, glioma, lymphoma. Clin Cancer Res. 2003;9:5370-5379. [PubMed] |
| 36. | Calabrese CR, Almassy R, Barton S, Batey MA, Calvert AH, Canan-Koch S, Durkacz BW, Hostomsky Z, Kumpf RA, Kyle S. Anticancer chemosensitization and radiosensitization by the novel poly(ADP-ribose) polymerase-1 inhibitor AG14361. J Natl Cancer Inst. 2004;96:56-67. [PubMed] [DOI] |
| 37. | Miknyoczki SJ, Jones-Bolin S, Pritchard S, Hunter K, Zhao H, Wan W, Ator M, Bihovsky R, Hudkins R, Chatterjee S. Chemopotentiation of temozolomide, irinotecan, and cisplatin activity by CEP-6800, a poly (ADP-ribose) polymerase inhibitor. Mol Cancer Ther. 2003;2:371-382. [PubMed] |
| 38. | Clinical trials: Cancer Research UK. Available from: http://wwweddn.org/clinicalTr_caResUK.html.. |
| 39. | Tentori L, Leonetti C, Scarsella M, Vergati M, Xu W, Calvin D, Morgan L, Tang Z, Woznizk K, Alemu C. Brain distribution and efficacy as chemosensitizer of an oral formulation of PARP-1 inhibitor GPI 15427 in experimental models of CNS tumors. Int J Oncol. 2005;26:415-422. [PubMed] [DOI] |
| 40. | First Patient Treated With New KuDOS Oral Anticancer Agent. Available from: http://www.kudospharma.co.uk/news/newsdesk.php.. |
| 41. | Veuger SJ, Curtin NJ, Richardson CJ, Smith GC, Durkacz BW. Radiosensitization and DNA repair inhibition by the combined use of novel inhibitors of DNA-dependent protein kinase and poly(ADP-ribose) polymerase-1. Cancer Res. 2003;63:6008-6015. [PubMed] |
| 42. | Brock WA, Milas L, Bergh S, Lo R, Szabo C, Mason KA. Radiosensitization of human and rodent cell lines by INO-1001, a novel inhibitor of poly(ADP-ribose) polymerase. Cancer Lett. 2004;205:155-160. [PubMed] [DOI] |
| 43. | Tutt A, Ashworth A. The relationship between the roles of BRCA genes in DNA repair and cancer predisposition. Trends Mol Med. 2002;8:571-576. [PubMed] [DOI] |