修回日期: 2021-08-28
接受日期: 2021-09-14
在线出版日期: 2021-11-08
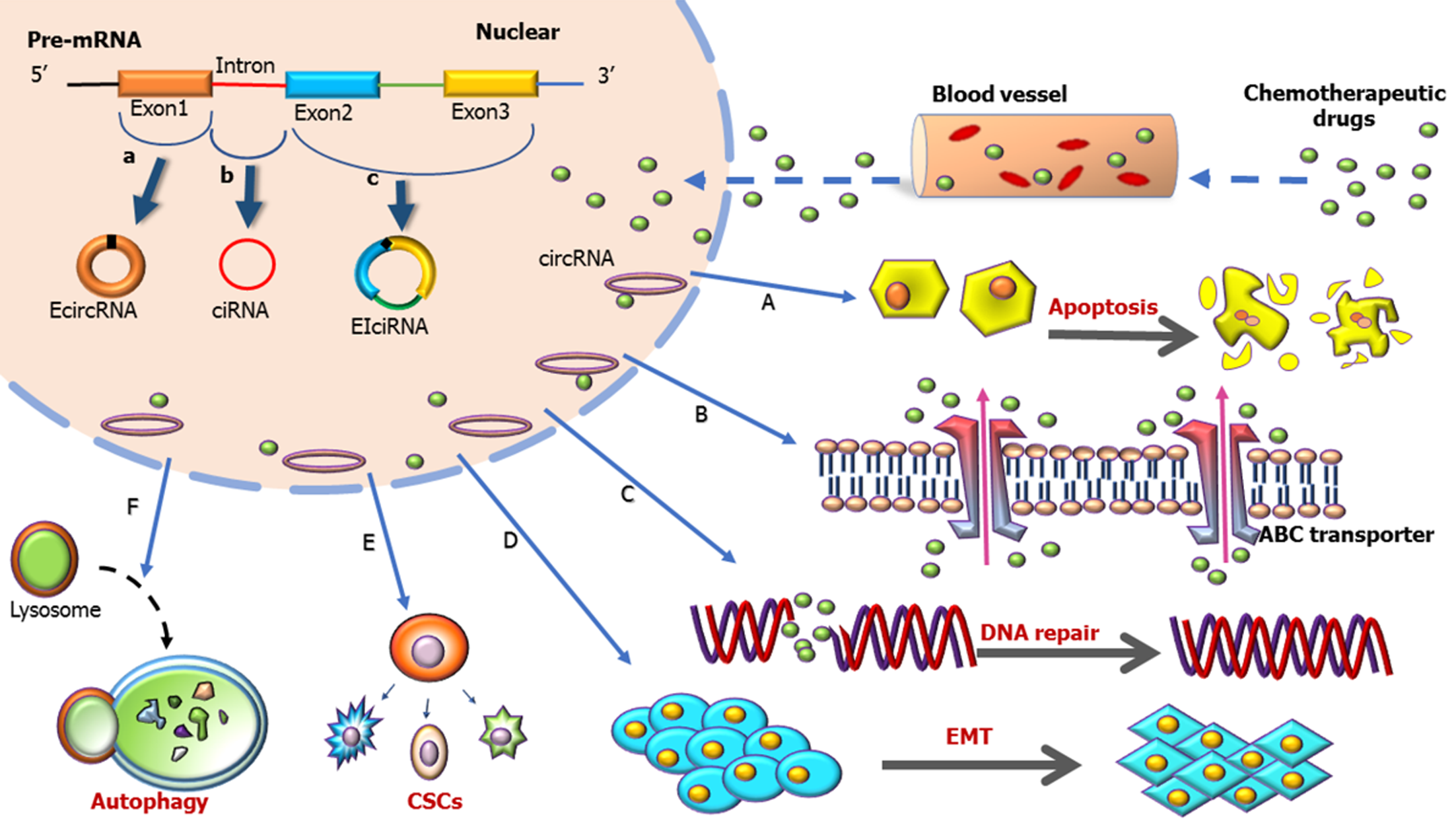
环状RNA (circRNAs)是一类具有独立闭合环状结构的新型非编码RNA. circRNAs在真核细胞中含量丰富, 具有独特的稳定性和组织特异性, 可以在转录和转录后水平上发挥生物调节作用. 越来越多的证据表明, circRNAs在肿瘤的发生、发展以及化疗耐药中起着至关重要的作用. 化疗是大多数消化系肿瘤的主要治疗手段, 它们的治疗效果常受到固有和获得性耐药的影响. circRNAs主要通过影响细胞凋亡、促进药物转运、促进DNA修复、促进上皮-间充质转化、调控肿瘤干细胞特性、影响自噬等多种机制调控肿瘤化疗耐药. 本文综述了circRNAs在消化系肿瘤化疗耐药中的最新研究进展及其机制, 并探讨了其潜在的临床应用前景.
核心提要: 本文就环状RNAs (circular RNAs, circRNAs)在消化系肿瘤化疗耐药中的研究进展作一综述. circRNAs通过一系列复杂的机制参与多种消化系肿瘤化疗耐药的调控, 有可能成为肿瘤防治的新型靶标.
引文著录: 林洁纯, 朱南星, 吴灵飞. circRNAs在消化系肿瘤化疗耐药中的研究进展. 世界华人消化杂志 2021; 29(21): 1237-1247
Revised: August 28, 2021
Accepted: September 14, 2021
Published online: November 8, 2021
Circular RNAs (circRNAs) are a novel class of noncoding RNA molecules with a unique closed continuous loop structure. CircRNAs are abundant in eukaryotic cells, have unique stability and tissue specificity, and can play a biological regulatory role at various levels, such as transcriptional and posttranscriptional levels. Accumulating evidence indicates that circRNAs play critical roles in tumor genesis, development, and chemotherapy. Chemotherapy is a primary type of intervention for most cancers, but its therapeutic efficacy is usually retarded by intrinsic and acquired resistance. CircRNAs regulate tumor chemoresistance through various molecular mechanisms, such as affecting apoptosis, promoting drug transportation, promoting DNA repair, promoting epithelial-mesenchymal transformation, regulating the characteristics of tumor stem cells, and affecting autophagy. This review summarizes the recent progress and mechanisms of circRNAs in cancer cell resistance to chemotherapy.
- Citation: Lin JC, Zhu NX, Wu LF. Research progress of circRNAs in chemotherapy resistance of digestive system neoplasms. Shijie Huaren Xiaohua Zazhi 2021; 29(21): 1237-1247
- URL: https://www.wjgnet.com/1009-3079/full/v29/i21/1237.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v29.i21.1237
肿瘤耐药的机制涉及多方面, 肿瘤生长、免疫系统特性和肿瘤微环境等不同特征决定了肿瘤的生物学行为. 这些特征是由复杂的潜在分子调控机制驱动的. 识别这些过程中的关键分子可以帮助我们了解耐药的发生, 这些分子在预测和逆转肿瘤耐药过程中发挥着重要作用. 近年, 环状RNAs (circular RNAs, circRNAs)在肿瘤化疗耐药中的作用受到广泛关注. 有证据表明, circRNAs参与了细胞多种生命活动, 并与肿瘤的发生进展以及化疗耐药密切相关, 本文结合国内外最新报道, 对circRNAs在消化系肿瘤化疗耐药中的作用作一综述.
circRNAs是在40多年前被首次发现的一类内源性非编码RNA (non-coding RNAs, ncRNAs), 与普通线性lncRNA不同, 它是由真核生物中数千种基因的前体mRNA反向剪接衍生出来的共价闭合结构, 缺少5'帽端和3' Poly A尾端[1,2]. 由于可抵抗RNA外切酶介导的降解, 因而比mRNAs更稳定[3]. 尽管真核生物中含有丰富的circRNAs, 科学家们最初认为它们没有任何功能[4]. 随着高通量RNA测序技术的发展和生物信息学技术的进步, circRNAs已经被证实在多种疾病中异常表达, 并与疾病的不良预后密切相关, 如心血管疾病[5]、神经系统疾病[6]、内分泌系统[6]、衰老[7]以及癌症[8]等. 大多数circRNAs是由外显子环化而成, 也有部分circRNAs是由内含子环化形成套索结构. 虽然环状RNA被广泛表达, 但其表达模式表现出组织特异性和细胞类型特异性. Chen等[9]将circRNAs分为三个亚型: 只包含外显子序列的全外显子型EcircRNAs (exonic circRNAs)[10]、仅包含内含子序列的ciRNAs (intronic circRNAs)[11]以及既包含外显子又有内含子序列的EIciRNAs (exon-intron circRNAs)[12]. 其中, EcircRNAs数量最多. 研究表明, 环状RNA可通过miRNA浸润、蛋白质相互作用、转录调控或在某些情况下进行自我翻译等方式发挥生物学功能, 具体如下: 第一, 作为microRNAs (miRNAs) 分子海棉. miRNAs是一类通过与mRNAs的3'端非翻译区靶点特异性结合负向调节mRNAs翻译的ncRNAs. 研究报道, 多数circRNAs含有miRNAs反应元件, 这些元件通过海棉样吸附miRNAs的作用, 从而调控miRNAs下游靶基因的表达[13]. 第二, 通过RNA结合蛋白(RNA binding proteins, RBPs)发挥作用. circRNAs可与某些RNA结合蛋白结合, 影响RNA的成熟、运输、定位和翻译, 从而调控相关蛋白表达及功能[14]. 第三, 调节基因转录. 一些circRNAs可通过与RNA聚合酶Ⅱ复合体结合并翻译相关蛋白来调节基因转录[15]. 如circRNAs可通过与启动子结合以调节mRNAs的表达[16]. 第四, 翻译蛋白质. 由于circRNAs缺乏帽依赖性翻译的必需元件, 如3'端的多聚腺苷尾和5'端的帽子结构, 以前认为它不具备翻译功能[17]. 然而, 新近研究证实, circZNF609包含从起始密码子开始的开放阅读框, 和线性转录本相似, 它以框内的终止密码子结束形成一个循环, 通过依赖剪切的方式而不需要依赖帽结构即可翻译成特定的蛋白质[18]. 越来越多的研究证明circRNAs通过上述机制参与了消化系肿瘤的化疗耐药, 并可作为肿瘤诊断的生物靶标及潜在治疗靶点[19,20]. 在这篇综述中, 我们重点讨论circRNAs在消化系肿瘤中的作用及其与耐药相关的机制(图1, 表1).
| 耐药相关机制 | 肿瘤 | circRNAs | 化疗药物 | 参考文献 |
| 影响细胞凋亡 | 肝癌 | circRNA-SORE、circFN1、circFOXM1 | 索拉非尼 | [22,41-43] |
| circ_0031242、circRNA_101505 | 铂类 | [46,48] | ||
| 胃癌 | circVAPA、circDONSON、circCCDC66、circFN1、hsa_circ_0081143、circPVT1、circ_0001546 | 铂类 | [56-62,68] | |
| circRACGAP1 | 阿帕替尼 | [34] | ||
| circMTHFD2 | 培美曲塞 | [70] | ||
| circHECTD1 | Diosbulbin-B | [71] | ||
| 结直肠癌 | circ_0032833、circDDX17 | 5-氟尿嘧啶 | [77,78] | |
| circ_0032833、hsa_circ_0079662、circ_0071589 | 铂类 | [77,80,81] | ||
| circCSPP1 | 阿霉素 | [82] | ||
| 食管癌 | circ_0006168 | 紫杉醇 | [87] | |
| circPSMC3 | 吉非替尼 | [88] | ||
| 促进药物转运 | 肝癌 | circFBXO11 | 奥沙利铂 | [25] |
| circFoxo3 | 阿霉素 | [50] | ||
| circPTGR1 | 5-氟尿嘧啶 | [51] | ||
| 胃癌 | circMTHFD2 | 培美曲塞 | [70] | |
| 结直肠癌 | circ_0007031 | 5-氟尿嘧啶 | [75] | |
| hsa_circ_0005963 | 铂类 | [24] | ||
| 促进DNA修复 | 胃癌 | circAKT3 | 铂类 | [27] |
| 促进上皮-间充质转化(EMT) | 肝癌 | circFoxo3、circ_0003998 | 阿霉素 | [50,52] |
| 胃癌 | circPVT1 | 紫杉醇 | [29] | |
| 结直肠癌 | circDDX17 | 5-氟尿嘧啶 | [78] | |
| 胰腺癌 | circHIPK3 | 吉西他滨 | [91] | |
| 调控肿瘤干细胞特性(CSCs) | 胃癌 | circFAM73A | 铂类 | [32] |
| 结直肠癌 | circ_001680 | 伊立替康 | [83] | |
| 影响自噬 | 肝癌 | circARNT2 | 铂类 | [45] |
| 胃癌 | circPVT1、circ_0032821、circCUL2、circMCTP2 | 铂类 | [61,64,66,67] | |
| circRACGAP1 | 阿帕替尼 | [34] |
细胞凋亡是一种程序性细胞死亡, 在基因水平上受到调控, 并以有序且有效的方式清除受损细胞, 这种死亡过程与肿瘤细胞无限增殖、疾病进展及化疗耐药密切相关[21]. 研究表明, 许多circRNAs通过调节凋亡相关基因来抑制癌细胞凋亡, 从而促进化疗耐药. 例如, circRNA-SORE可竞争性结合miR-103a-2-5p和miR-660-3p, 激活Wnt/β-catenin通路, 抑制肝癌(hepatocellular carcinoma, HCC)细胞凋亡, 从而促进HCC细胞对索拉非尼的耐药[22].
ABC转运蛋白家族由48个基因组成, 分为7个亚家族: 从ABCA到ABCG. 这些基因可编码膜蛋白, 在外源物质、代谢物和信号分子通过细胞膜的转运途径中发挥重要作用[23]. 越来越多研究表明, circRNAs可以靶向作用ABC转运蛋白, 通过增强ABC转运蛋白的功能或者直接上调ABC转运家族蛋白表达, 促进药物从细胞内排泄, 降低药物的有效浓度而诱导耐药. 例如, Wang等[24]研究发现hsa_circ_0005963通过miR-122/PKM2轴促进结直肠癌(colorectal cancer, CRC)糖酵解和ATP的合成, 促进ABC转运蛋白对奥沙利铂的排泄, 从而减轻奥沙利铂对肿瘤的毒性作用. 另外, circFBXO11可通过海绵样吸附miR-605上调靶基因FOXO3的表达, 而FOXO3作为转录因子可促进ABCB1的转录水平, 促进药物排泄, 进而诱导HCC的奥沙利铂耐药性[25].
DNA修复是细胞识别并纠正化疗和其他因素引起DNA损伤的生物学事件. 其主要机制包括碱基切除修复、核苷酸切除修复、错配修复、同源重组修复、非同源末端连接和链间交联修复, 它们可导致对DNA靶向化疗药物的耐药性[26]. 目前人们已经发现了多种由circRNAs介导的DNA损伤修复机制诱导的耐药性. 例如, 在胃癌(gastric cancer, GC)中, circAKT3通过miR-198/PIK3R1轴促进DNA损伤修复, 从而诱导GC细胞对顺铂耐药[27].
促进上皮-间充质转化(epithelial-mesenchymal transition, EMT)是指上皮细胞在特定的生理或病理情况下, 失去上皮特性, 获得间充质特征的生理学现象. 它不仅在发育和伤口愈合中起关键作用, 并且参与了多种肿瘤的恶性演进过程, 包括肿瘤的侵袭迁移、抗凋亡和耐药[28]. 研究表明, 某些circRNAs可通过调节EMT导致耐药. 例如, 在紫杉醇耐药的GC细胞中, 高表达的circPVT1通过海绵样吸附miR-124-3p上调ZEB1的表达, 进而促进EMT, 诱导其对紫杉醇耐药[29].
| 癌症 | circRNA | 表达 | 促进/抑制耐药 | 机制 | 细胞系 | 化疗药物 | 参考文献 |
| 肝癌 | circRNA-SORE | ↑ | 促进 | YBX1 | HepG2、LM3和SKHep1 | 索拉非尼 | [41] |
| circRNA-SORE | ↑ | 促进 | miR-103a-2-5p和miR-660-3p/Wnt/β-catenin | HepG2、LM3和SKHep1 | 索拉非尼 | [22] | |
| circFN1 | ↑ | 促进 | miR-1205/E2F1 | Huh7和HepG2 | 索拉非尼 | [42] | |
| circFOXM1 | ↑ | 促进 | miR-1324/MECP2 | HepG2 和 Huh7 | 索拉非尼 | [43] | |
| circARNT2 | ↑ | 促进 | miR-155-5p/PDK1 | Hep3B和Huh7 | 顺铂 | [45] | |
| circ_0031242 | ↑ | 促进 | miR-924/POU3F2 | HuH7和SNU387 | 顺铂 | [46] | |
| circRNA_102272 | ↑ | 促进 | MiR-326/RUNX2 | HepG2和HuH7 | 顺铂 | [47] | |
| circFBXO11 | ↑ | 促进 | miR-605/FOXO3/ABCB1 | HepG2 | 奥沙利铂 | [25] | |
| circRNA_101505 | ↓ | 抑制 | miR-103/NOR1 | Hep3B和Huh7 | 顺铂 | [48] | |
| circ_0003418 | ↓ | 抑制 | Wnt/β-Catenin | Hep3B和Huh7 | 顺铂 | [49] | |
| circFoxo3 | ↑ | 促进 | miR-199a-5p/ABCC1 | SKHEP-1和HepG2 | 阿霉素 | [50] | |
| circular PTGR1 | ↑ | 促进 | miR-129-5p/ABCC1 | Hep3B和Huh7 | 5-氟尿嘧啶 | [51] | |
| circ_0003998 | ↑ | 促进 | miR-218-5p/EIF5A2 | Huh7和HCCLM3 | 阿霉素 | [52] | |
| 胃癌 | circVAPA | ↑ | 促进 | miR-125b-5p/STAT3 | SGC7901和HGC27 | 顺铂 | [56] |
| circAKT3 | ↑ | 促进 | miR-198/PIK3R1 | SGC7901 | 顺铂 | [27] | |
| circFAM73A | ↑ | 促进 | miR-490-3p /HMGA2 | BGC823和SGC7901 | 顺铂 | [32] | |
| circDONSON | ↑ | 促进 | miR-802/BMI1 | AGS和HGC-27 | 顺铂 | [57] | |
| circCCDC66 | ↑ | 促进 | miR-618 /BCL2 | BGC823和SGC7901 | 顺铂 | [58] | |
| circFN1 | ↑ | 促进 | miR-182-5p | SGC7901 | 顺铂 | [59] | |
| hsa_circ_0081143 | ↑ | 促进 | miR-646/CDK6 | SGC7901和MGC803 | 顺铂 | [60] | |
| circ-PVT1 | ↑ | 促进 | miR-30a-5p/YAP1 | HGC-27和AGS | 顺铂 | [61] | |
| circPVT1 | ↑ | 促进 | miR-152-3p/HDGF/PI3K/AKT | AGS和HGC-27 | 顺铂 | [62] | |
| circ_0026359 | ↑ | 促进 | miR-1200/POLD4 | SGC-7901和MKN-45 | 顺铂 | [63] | |
| circ_0032821 | ↑ | 促进 | miR-515-5p/SOX9 | HGC-27和AGS | 奥沙利铂 | [64] | |
| hsa_circ_0000144 | ↑ | 促进 | miR-502-5p/ADAM9 | AGS和MKN45 | 奥沙利铂 | [65] | |
| circCUL2 | ↓ | 抑制 | miR-142-3p/ROCK2 | AGS和SGC-7901 | 顺铂 | [66] | |
| circMCTP2 | ↓ | 抑制 | miR-99a-5p/MTMR3 | BGC823和SGC7901 | 顺铂 | [67] | |
| hsa_circ_0001546 | ↓ | 抑制 | miR-421/ATM/Chk2/p53 | HGC-27 | 奥沙利铂 | [68] | |
| circRACGAP1 | ↑ | 促进 | miR-3657/ATG7 | BGC-823和HGC-27 | 阿帕替尼 | [34] | |
| circ-PVT1 | ↑ | 促进 | miR-124-3p/ZEB1 | MGC-803和AGS | 紫杉醇 | [29] | |
| circNRIP1 | ↑ | 促进 | miR-138-5p/HIF-1α | SGC-7901和 BGC-823 | 5-氟尿嘧啶 | [69] | |
| circMTHFD2 | ↑ | 促进 | miR-124/FDZ5/MDR-1 | MGC-803 | 培美曲塞 | [70] | |
| circHECTD1 | ↑ | 促进 | miR-137/PBX3 | AGS和HGC-27 | Diosbulbin-B | [71] | |
| 结直肠癌 | circ_0007031 | ↑ | 促进 | miR-133b/ABCC5 | HCT116和SW480 | 5-氟尿嘧啶 | [75] |
| circ-PRKDC | ↑ | 促进 | miR-375/FOXM1/Wnt/β-catenin | SW620和SW480 | 5-氟尿嘧啶 | [76] | |
| circ_0032833 | ↑ | 促进 | miR-125-5p/MSI1 | HCT116 | 5-氟尿嘧啶和奥沙利铂 | [77] | |
| circDDX17 | ↓ | 抑制 | miR-31-5p/KANK1 | HCT116和SW480 | 5-氟尿嘧啶 | [78] | |
| hsa_circ_0005963 | ↑ | 促进 | miR-122/PKM2 | HCT116和SW480 | 奥沙利铂 | [24] | |
| circ-CCDC66 | ↑ | 促进 | -- | HCT116和HT-2 | 奥沙利铂 | [79] | |
| hsa_circ_0079662 | ↑ | 促进 | miR-324-5p/TNF-α | HCT116 | 奥沙利铂 | [80] | |
| circ_0071589 | ↑ | 促进 | MiR-526b-3p/KLF12 | HCT116和LOVO | 顺铂 | [81] | |
| circCSPP1 | ↑ | 促进 | miR-944/FZD7 | LoVo和HCT116 | 阿霉素 | [82] | |
| hsa_circ_001680 | ↑ | 促进 | miR-340/BMI1 | SW480和HCT116 | 伊立替康 | [83] | |
| 食管癌 | circ_0006168 | ↑ | 促进 | miR-194-5p/JMJD1C | ECA109和KYSE150 | 紫杉醇 | [87] |
| circPSMC3 | ↓ | 抑制 | miR-10a-5p/PTEN | TE1和KYSE450 | 吉非替尼 | [88] | |
| 胰腺癌 | circHIPK3 | ↑ | 促进 | miR-330-5p/RASSF1 | PANC-1和SW-1990 | 吉西他滨 | [91] |
肝细胞癌是原发性HCC中最常见的肿瘤类型, 其发病率在全球范围内呈上升趋势. 根据2020年癌症统计数据, HCC是全球第七大最常见的癌症和第三大癌症相关死亡原因[35]. HCC主要与慢性肝病和肝硬化有关, 目前主要治疗方法有手术切除、肝移植、射频消融(radiofrequency ablation, RFA)、经导管肝动脉化疗栓塞(transcatheter arterial chemoembolization, TACE)及联合化疗[36,37]. 由于大多数患者确诊时已处于晚期阶段而失去手术机会, 化疗是控制HCC发展和延长患者生命最主要的方法. 然而, 化疗耐药仍然是HCC患者治疗的主要障碍[38]. 因此, 迫切需要寻找新的治疗靶点以及增强化疗药物敏感性的方法.
3.1.1 索拉非尼: 作为FDA批准的第一种分子靶向药物, 索拉非尼可将HCC患者总生存期平均延长3 mo[39]. 然而, 许多HCC患者在应用索拉非尼治疗6 mo后开始对索拉非尼耐药[40].
研究报道, circRNAs可促进HCC细胞索拉非尼耐药性. Xu等[41]通过微阵列分析发现一种新型circRNA在索拉非尼耐药的HCC细胞中显著上调, 并在促进HCC索拉非尼耐药中发挥关键作用, 该circRNA被命名为circRNA-SORE. 进一步研究表明, circRNA-SORE在细胞质中通过与致癌蛋白YBX1结合并阻止其核移位, 从而阻断E3泛素连接酶PRP19介导的YBX1降解, 进而上调了YBX1及其下游靶基因AKT、Raf1、ERK、c-Myc和TGF-β1的表达, 抑制HCC细胞凋亡, 诱导HCC细胞耐药. 同时, 作者还证明circRNA-SORE由外泌体转运, 使得索拉非尼耐药可在HCC细胞间进行传递. 有趣的是, 作者在另一篇文章中报道, circRNA-SORE还可作为分子诱饵吸附miR-103a-2-5p和miR-660-3p进而激活Wnt/β-catenin信号通路, 抑制HCC细胞凋亡, 促进肿瘤细胞对索拉非尼的耐药. 并且, m6A甲基化在上调索拉非尼耐药细胞中的circRNA-SORE水平中发挥了重要作用[22]. Yang等[42]利用circRNA微阵列技术发现circFN1在耐药HCC组织中上调, 实验结果表明circFN1通过竞争性结合miR-1205上调E2F1表达, 抑制索拉非尼诱导的HCC细胞凋亡, 介导HCC细胞对索拉非尼耐药. Weng等[43]研究发现circFOXM1在索拉非尼耐药HCC细胞中上调, 其机制是circFOXM1充当miR-1324分子海绵, 减弱miR-1324对下游靶基因MECP2的抑制, 从而降低了索拉非尼对HCC细胞的毒性作用, 诱导HCC对索拉菲尼耐药.
3.1.2 铂类: 顺铂是第一代铂类化疗药物, 它通过在DNA双链之间形成交联来抑制DNA复制和转录, 具有广谱的抗肿瘤活性. 顺铂是治疗晚期HCC最常用的化疗药物之一. 然而, 对顺铂的耐药亦是HCC患者获得满意疗效的主要障碍[44].
circRNAs参与了HCC组织及细胞对顺铂耐药的调控. Li等[45]研究发现, circARNT2在顺铂耐药的HCC细胞中表达上调, 体内外实验证明, circARNT2可通过ceRNA机制竞争性结合miRNA-155-5p, 从而上调PDK1的表达, 随后抑制HCC细胞自噬, 促进其生长并诱导HCC细胞对顺铂耐药. Fan等[46]报道, circ_0031242通过与miR-924竞争性结合上调POU3F2表达, 从而促进了HCC细胞增殖、迁移和侵袭, 抑制了细胞的凋亡, 产生对顺铂耐药. Guan等[47]报道, circRNA_102272可通过ceRNA机制海绵样吸附miR-326导致RUNX2表达增加, 从而促进HCC细胞增殖和顺铂耐药. Li等[25]报道, circFBXO11在肝细胞癌组织中显著上调, 并且与预后不良呈正相关. circFBXO11通过竞争性结合miR-605而上调靶基因FOXO3表达, 促进ABCB1转录, 在HCC对奥沙利铂耐药中发挥重要的作用.
另外, 某些circRNAs也可在顺铂耐药的HCC组织及细胞中表达下调并增强顺铂化疗敏感性. Luo等[48]研究报道, circRNA_101505在顺铂耐药的HCC组织和细胞系中表达降低, 过表达circRNA_101505可通过海绵样吸附miR-103导致NOR1上调, 促进HCC细胞凋亡, 增强HCC细胞对顺铂的敏感性. Chen等[49]研究亦发现, circ_0003418在顺铂耐药的HCC组织和细胞系中表达下调, 敲低circ_0003418可通过激活Wnt/β-catenin信号通路, 促进HCC细胞对顺铂耐药.
3.1.3 其他化疗药物: 阿霉素和5-氟尿嘧啶(5-FU)广泛应用于HCC患者的联合化疗. 研究报道, circFoxo3在阿霉素耐药HCC组织、HCC细胞系及转移组织中的表达均高于未转移组织. 一方面, circFoxo3可通过海绵样吸附miR-199a-5p, 上调ABCC1的表达; 另一方面, 它可促进N-cadherin和Vimentin的表达, 抑制E-cadherin的表达, 促进EMT进而促进HCC细胞的转移及其产生阿霉素耐药[50]. 另外, circPTGR1在HCC组织中表达上调, 它可海绵样吸附miR-129-5p上调ABCC1水平, 加速5-FU从胞内排泄[51]. Li等[52]报道, circ_0003998亦通过ceRNA机制竞争性结合miR-218-5p, 上调EIF5A2的水平, 促进MMP9, Vimentin和N-cadherin的表达, 抑制E-cadherin进而促进EMT, 介导HCC对阿霉素的耐药.
GC是人类最常见的恶性肿瘤之一, 2020年它是全球第五大新发癌症, 也是全球第四大癌症相关死亡原因[35]. 化疗是治疗晚期GC的首选策略, 顺铂、奥沙利铂、5-氟尿嘧啶通常作为一线治疗用药[53-55], 紫杉醇、培美曲塞、阿帕替尼、Diosbulbin-B(DB)亦用于GC的化疗. 与大多数肿瘤一样, GC在发生发展中亦表现出恶性增殖、凋亡抵制、侵袭增强以及化疗耐药, 化疗耐药的出现限制了患者的总体临床疗效. 新近, circRNAs在GC中的作用受到广泛关注.
3.2.1 铂类: 以铂类化疗药为基础的化疗是临床GC的主要治疗方法, 研究表明circRNAs在调节GC的化疗耐药中发挥截然不同的作用.
一方面, circRNA可以增强GC对铂类化疗药物耐药. Deng等[56]发现, circVAPA在顺铂耐药的GC组织中明显上调, 其机制与circVAPA竞争性结合miR-125b-5p上调STAT3表达, 抑制GC细胞凋亡而介导GC细胞对顺铂耐药有关. Huang等[27]通过微阵列分析发现, circAKT3在顺铂耐药的GC组织和细胞中的表达均显著上调, circAKT3通过海绵样吸附miR-198上调PIK3R1表达, 通过促进DNA损伤修复而产生耐药. Xia等[32]在顺铂耐药的GC组织及细胞中还发现了高表达的circFAM73A. 一方面, 它可通过充当miR-490-3p的分子海绵, 上调HMGA2的表达; 另一方面, 它又可作为RNA结合蛋白, 与hNRNPK结合, 增强hNRNPK与β-catenin之间的相互作用, 并增强β-catenin的稳定性, 进而促进GC干细胞样特性, 产生对顺铂耐药. 另一项研究显示, circDONSON通过ceRNA机制竞争性结合miR-802, 上调BMI1的表达, 抑制Caspase-3和Caspase-9的裂解, 抑制细胞凋亡, 从而增强了GC细胞对顺铂耐药[57]. 新近, Zhang等[58]报道, circCCDC66在顺铂耐药的GC细胞中表达上调, 其机制与调节miR-618/BCL2信号通路抑制GC细胞凋亡有关. Huang等[59]研究报道, circFN1通过吸附miR-182-5p抑制GC细胞凋亡, 产生对顺铂的耐药性. 作为circRNA参与GC顺铂耐药的另一个例子, Xue等[60]研究发现, hsa_circ_0081143通过miR-646/CDK6信号通路抑制GC细胞凋亡, 参与了GC细胞对顺铂耐药. Yao等[61]研究发现, 在顺铂耐药的GC细胞中circPVT1表达上调, 它通过充当miR-30a-5p的分子海绵, 上调YAP1表达及LC3-Ⅱ/Ⅰ比值并下调p62表达水平, 诱导GC细胞自噬, 抑制GC细胞凋亡, 从而促进GC细胞对顺铂耐药. 在另一项研究中也报道了在顺铂耐药的GC细胞中circPVT1表达上调, 它则通过竞争性结合miR-152-3p上调HDGF的表达, 并激活PI3K/AKT通路, 抑制GC细胞凋亡, 并产生化疗耐药[62]. Zhang等[63]研究发现, circ_0026359在顺铂耐药的GC细胞中高表达, 它通过调节miR-1200/POLD4信号通路, 抑制caspase-3/7信号通路介导的细胞凋亡, 从而产生顺铂耐药. Zhong等[64]研究发现, circ_0032821可作为miR-515-5p的分子海绵, 上调SOX9的表达, 并通过外泌体分泌传播, 介导GC细胞对奥沙利铂的耐药. 另外, 在奥沙利铂耐药的GC组织和细胞中hsa_circ_0000144表达上调, 进一步研究表明, 它通过miR-502-5p/ADAM9轴诱导GC细胞对奥沙利铂的耐药性[65].
另一方面, circRNA还可提高GC对铂类化疗药的敏感性. Peng等[66]研究发现, 在顺铂耐药的GC细胞系中circCUL2表达下调, 过表达circCUL2可通过ceRNA机制竞争性结合miR-142-3p, 上调ROCK2和P62的表达, 抑制自噬活性标志物LC3和Beclin1的表达, 从而抑制GC细胞自噬, 增强了顺铂的化疗敏感性. 另外, Sun等[67]发现, 与顺铂敏感的GC细胞相比, 顺铂耐药的GC细胞circMCTP2的表达明显下调, 过表达circMCTP2可通过海绵样吸附miR-99a-5p上调MTMR3和P62的表达, 并下调LC3和Beclin1的表达, 通过抑制自噬而增强GC细胞对顺铂的敏感性. 另一研究发现, circ_0001546在奥沙利铂耐药的GC组织和细胞系中表达下调, 过表达circ_0001546可通过活化miR-421/ATM/Chk2/p53信号通路, 诱导GC细胞凋亡, 增强GC细胞对奥沙利铂的敏感性[68].
3.2.2 其他化疗药物: circRACGAP1在阿帕替尼处理的GC细胞中表达上调, 它作为miR-3657的海绵上调ATG7的表达, 诱导GC细胞自噬并抑制细胞凋亡, 介导GC细胞对阿帕替尼的耐药性[34]. Liu等[29]研究表明, circPVT1在紫杉醇耐药的GC组织和细胞中表达上调, 它通过海绵样吸附miR-124-3p上调ZEB1的表达, 而ZEB1是E-cadherin的关键转录抑制剂, 可加速EMT, 进而诱导其对紫杉醇耐药. 另外, Xu等[69]研究报道, 在GC细胞中, 低氧可诱导circNRIP1表达上调, 它可通过竞争性结合miR-138-5p, 上调HIF-1α的表达, 促进糖酵解, 导致癌细胞在低氧条件下存活, 诱导缺氧条件下GC细胞对5-FU的耐药性. 另外, circMTHFD2是一种在培美曲塞耐药的GC细胞中表达上调的circRNA, 它通过与miR-124竞争性结合, 上调TS和ABCC11的表达, 下调RCF的表达, 促进毒物排泄并抑制GC细胞凋亡, 从而介导GC细胞对培美曲塞的耐药[70]. Lu等[71]研究报道, hsa_circ_0031452(也称circHECTD1)在GC组织及细胞中表达上调, circHECTD1作为miR-137的分子海绵上调PBX3的表达, 抑制GC细胞凋亡, 从而产生对Diosbulbin-B的耐药.
在全球范围内, CRC是第三大最常见的恶性肿瘤, 2020年新增病例超过180万例, 死亡人数近91万人[35]. 针对CRC的基本疗法包括手术、放疗和化疗.
3.3.1 5-氟尿嘧啶: 5-氟尿嘧啶(5-FU)是一种有效的抗肿瘤药物, 已被广泛应用于CRC的化疗, 以5-FU为基础的联合治疗极大地提高了CRC的生存率[72]. 然而, 对5-FU的耐药性已经严重限制了5-FU对CRC患者的疗效[73,74].
He等[75]研究表明, circ_0007031通过ceRNA机制靶向作用miR-133b/ABCC5轴, 促进CRC细胞的恶性行为, 诱导了CRC细胞对5-FU的耐药性. circ-PRKDC通过海绵样吸附miR-375上调FOXM1表达, 激活Wnt/β-catenin通路, 促进了CRC细胞的集落形成及侵袭行为, 促进CRC细胞对5-FU耐药[76]. Li等[77]研究报道, circ_0032833在5-氟尿嘧啶和奥沙利铂耐药的CRC组织及细胞中表达明显上调. 它通过竞争性结合miR-125-5p上调MSI1的表达, 抑制CRC细胞凋亡并促进其转移, 介导癌细胞对5-氟尿嘧啶和奥沙利铂的耐药.
有趣的是, 也有学者发现, 一些circRNAs在CRC组织和细胞中可增强化疗药物敏感性. Ren等[78]研究表明, circDDX17在CRC组织和细胞中表达下调, 而过表达circDDX17可增强肿瘤细胞对5-FU的敏感性并抑制CRC进展. 其机制与circDDX17吸附miR-31-5p上调KANK1表达, 诱导癌细胞凋亡, 抑制细胞侵袭和EMT, 从而提高CRC对5-FU的敏感性有关.
3.3.2 铂类: Wang等[24]研究发现, hsa_circ_0005963(也称ciRS-122)在奥沙利铂耐药CRC组织及细胞中表达上调, 并与化疗耐药呈正相关. 体内外研究表明, 奥沙利铂耐药细胞中的外泌体可将ciRS-122运送到奥沙利铂敏感细胞, 通过海绵样吸附miR-122, 上调PKM2的表达, 从而促进糖酵解和ATP合成, 促进ABC转运家族蛋白对奥沙利铂的排泄, 从而诱导CRC对奥沙利铂产生耐药. 另外, 有学者报道奥沙利铂通过诱导PI3KK介导的底物结合结构域附近的磷酸化导致DHX9功能低下, 进而促进circCCDC66表达, 产生化疗耐药[79]. Lai等[80]研究报道, hsa_circ_0079662通过ceRNA机制与miR-324-5p竞争性结合, 上调HOXA9的表达, 通过TNF-α途径抑制CRC细胞凋亡, 诱导CRC细胞对奥沙利铂的耐药性. Zhang等[81]研究报道, circ_0071589在顺铂耐药的CRC组织和细胞系中表达上调, circ_0071589敲低可抑制CRC对顺铂的耐药性、增殖、迁移和侵袭, 并促进细胞凋亡, 进一步研究表明, circ_0071589可通过竞争性结合miR-526b-3p, 上调KLF12的表达, 抑制细胞凋亡, 进而诱导CRC对顺铂的耐药性.
3.3.3 其他化疗药物: Xi等[82]报道, 在阿霉素耐药的CRC组织和细胞中, circCSPP1的表达上调, 它通过ceRNA机制竞争性结合miR-944, 靶向FZD7的3'非翻译区(UTR)降低FZD7表达水平, 促进CRC细胞的增殖、迁移、侵袭并抑制细胞凋亡, 促进CRC细胞对阿霉素耐药. 在另一篇研究中, circ_001680在CRC组织中的表达高于其配对的癌旁正常组织, 机制上, circ_001680通过靶向miR-340来上调BMI 1的表达, 促进CRC干细胞特性, 并诱导CRC对伊立替康耐药[83].
食管癌(esophageal cancer, EC)是世界范围内常见的恶性肿瘤, 发病率居第9位, 死亡率居第6位[35]. 食管鳞状细胞癌(esophageal squamous cell carcinoma, ESCC)是EC中最常见的组织学类型, 并且近一半的病例发生在中国[84]. 尽管手术治疗、放疗、化疗和靶向治疗都对治疗做出了重大贡献, 但EC患者的5年生存率仍然很低, 因为患者往往被诊断为晚期[85]. 化疗耐药是导致ESCC患者预后不良的重要因素之一[86].
Qu等[87]研究报道, circ_0006168在EC组织、细胞和紫杉醇耐药细胞中表达上调, circ_0006168通过与miR-194-5p竞争性结合上调JMJD1C表达, 进而促进EC细胞增殖并抑制其凋亡, 介导其对紫杉醇的耐药. 另一篇研究报道, 在吉非替尼耐药的食管鳞癌组织和细胞中, circPSMC3表达下调, 过表达circPSMC3可通过ceRNA机制竞争性结合miR-10a-5p上调PTEN表达, 促进EC细胞凋亡, 从而增强EC对吉非替尼的敏感性[88].
总之, 作为近年来非编码RNA家族中的一个新研究热点, circRNAs在消化系肿瘤中化疗耐药的作用已得到越来越多研究的证实. circRNAs具有组织和细胞类型特异性, 也具有癌症和药物反应特异性, 导致circRNAs的表达和相关的调控机制不同. 一方面, 环状RNA可能通过调节其表达作为直接治疗靶点, 一些主要的致癌环状RNA在多种肿瘤中异常表达, 并与多药耐药相关, 其独特的反向剪切连接是一个潜在的特异性治疗靶点, 精确调控这些致癌环状RNA可能对恶性肿瘤发挥治疗作用. 另一方面, 由于circRNAs具有高丰度和高稳定性以及对miRNA具有多重结合位点, 环状RNA还是潜在的治疗海绵载体. 此外, 在外周血、尿液和唾液中分泌的circRNAs更容易被临床检测到, 因此, 需要对体液中存在的circRNAs进行更多的研究, 以开发用于多种肿瘤筛查和预后监测的circRNAs检测试剂盒, 以促进circRNAs在肿瘤诊断、预后和治疗中应用. 虽然目前尚无circRNAs用于抗癌治疗的文献报道, 相信在未来, 随着circRNAs被充分证明参与了抗癌耐药的调控以及在预后监测中的价值, 针对circRNAs的干预措施有望在克服肿瘤化疗耐药中探索一条新途径.
学科分类: 胃肠病学和肝病学
手稿来源地: 广东省
同行评议报告学术质量分类
A级 (优秀): A
B级 (非常好): B
C级 (良好): C
D级 (一般): 0
E级 (差): 0
科学编辑:刘继红 制作编辑:张砚梁
| 1. | Aufiero S, van den Hoogenhof MMG, Reckman YJ, Beqqali A, van der Made I, Kluin J, Khan MAF, Pinto YM, Creemers EE. Cardiac circRNAs arise mainly from constitutive exons rather than alternatively spliced exons. RNA. 2018;24:815-827. [PubMed] [DOI] |
| 2. | Sanger HL, Klotz G, Riesner D, Gross HJ, Kleinschmidt AK. Viroids are single-stranded covalently closed circular RNA molecules existing as highly base-paired rod-like structures. Proc Natl Acad Sci USA. 1976;73:3852-3856. [PubMed] [DOI] |
| 3. | Li D, Yang Y, Li ZQ, Li LC, Zhu XH. Circular RNAs: from biogenesis and function to diseases. Chin Med J (Engl). 2019;132:2457-2464. [PubMed] [DOI] |
| 4. | Capel B, Swain A, Nicolis S, Hacker A, Walter M, Koopman P, Goodfellow P, Lovell-Badge R. Circular transcripts of the testis-determining gene Sry in adult mouse testis. Cell. 1993;73:1019-1030. [PubMed] [DOI] |
| 5. | Zhang L, Han B, Liu H, Wang J, Feng X, Sun W, Cai D, Jia H, Jiang D. Circular RNA circACSL1 aggravated myocardial inflammation and myocardial injury by sponging miR-8055 and regulating MAPK14 expression. Cell Death Dis. 2021;12:487. [PubMed] [DOI] |
| 6. | Ma N, Pan J, Wen Y, Wu Q, Yu B, Chen X, Wan J, Zhang W. circTulp4 functions in Alzheimer's disease pathogenesis by regulating its parental gene, Tulp4. Mol Ther. 2021;29:2167-2181. [PubMed] [DOI] |
| 7. | Kim E, Kim YK, Lee SV. Emerging functions of circular RNA in aging. Trends Genet. 2021;37:819-829. [PubMed] [DOI] |
| 8. | Wang J, Zhang Y, Song H, Yin H, Jiang T, Xu Y, Liu L, Wang H, Gao H, Wang R, Song J. The circular RNA circSPARC enhances the migration and proliferation of colorectal cancer by regulating the JAK/STAT pathway. Mol Cancer. 2021;20:81. [PubMed] [DOI] |
| 9. | Liao KM, Chao TB, Tian YF, Lin CY, Lee SW, Chuang HY, Chan TC, Chen TJ, Hsing CH, Sheu MJ, Li CF. Overexpression of the PSAT1 Gene in Nasopharyngeal Carcinoma Is an Indicator of Poor Prognosis. J Cancer. 2016;7:1088-1094. [PubMed] [DOI] |
| 10. | Zhang XO, Wang HB, Zhang Y, Lu X, Chen LL, Yang L. Complementary sequence-mediated exon circularization. Cell. 2014;159:134-147. [PubMed] [DOI] |
| 11. | Zhang Y, Zhang XO, Chen T, Xiang JF, Yin QF, Xing YH, Zhu S, Yang L, Chen LL. Circular intronic long noncoding RNAs. Mol Cell. 2013;51:792-806. [PubMed] [DOI] |
| 12. | Li Z, Huang C, Bao C, Chen L, Lin M, Wang X, Zhong G, Yu B, Hu W, Dai L, Zhu P, Chang Z, Wu Q, Zhao Y, Jia Y, Xu P, Liu H, Shan G. Exon-intron circular RNAs regulate transcription in the nucleus. Nat Struct Mol Biol. 2015;22:256-264. [PubMed] [DOI] |
| 13. | Hansen TB, Jensen TI, Clausen BH, Bramsen JB, Finsen B, Damgaard CK, Kjems J. Natural RNA circles function as efficient microRNA sponges. Nature. 2013;495:384-388. [PubMed] [DOI] |
| 14. | Wawrzyniak O, Zarębska Ż, Kuczyński K, Gotz-Więckowska A, Rolle K. Protein-Related Circular RNAs in Human Pathologies. Cells. 2020;9. [PubMed] [DOI] |
| 15. | Chen LL. The biogenesis and emerging roles of circular RNAs. Nat Rev Mol Cell Biol. 2016;17:205-211. [PubMed] [DOI] |
| 16. | Chen N, Zhao G, Yan X, Lv Z, Yin H, Zhang S, Song W, Li X, Li L, Du Z, Jia L, Zhou L, Li W, Hoffman AR, Hu JF, Cui J. A novel FLI1 exonic circular RNA promotes metastasis in breast cancer by coordinately regulating TET1 and DNMT1. Genome Biol. 2018;19:218. [PubMed] [DOI] |
| 17. | Pamudurti NR, Bartok O, Jens M, Ashwal-Fluss R, Stottmeister C, Ruhe L, Hanan M, Wyler E, Perez-Hernandez D, Ramberger E, Shenzis S, Samson M, Dittmar G, Landthaler M, Chekulaeva M, Rajewsky N, Kadener S. Translation of CircRNAs. Mol Cell. 2017;66:9-21.e7. [PubMed] [DOI] |
| 18. | Legnini I, Di Timoteo G, Rossi F, Morlando M, Briganti F, Sthandier O, Fatica A, Santini T, Andronache A, Wade M, Laneve P, Rajewsky N, Bozzoni I. Circ-ZNF609 Is a Circular RNA that Can Be Translated and Functions in Myogenesis. Mol Cell. 2017;66:22-37.e9. [PubMed] [DOI] |
| 19. | Tao X, Shao Y, Yan J, Yang L, Ye Q, Wang Q, Lu R, Guo J. Biological roles and potential clinical values of circular RNAs in gastrointestinal malignancies. Cancer Biol Med. 2021;. [PubMed] [DOI] |
| 20. | Li J, Xu Q, Huang ZJ, Mao N, Lin ZT, Cheng L, Sun B, Wang G. CircRNAs: a new target for the diagnosis and treatment of digestive system neoplasms. Cell Death Dis. 2021;12:205. [PubMed] [DOI] |
| 21. | Li K, van Delft MF, Dewson G. Too much death can kill you: inhibiting intrinsic apoptosis to treat disease. EMBO J. 2021;40:e107341. [PubMed] [DOI] |
| 22. | Xu J, Wan Z, Tang M, Lin Z, Jiang S, Ji L, Gorshkov K, Mao Q, Xia S, Cen D, Zheng J, Liang X, Cai X. N6-methyladenosine-modified CircRNA-SORE sustains sorafenib resistance in hepatocellular carcinoma by regulating β-catenin signaling. Mol Cancer. 2020;19:163. [PubMed] [DOI] |
| 23. | Nobili S, Lapucci A, Landini I, Coronnello M, Roviello G, Mini E. Role of ATP-binding cassette transporters in cancer initiation and progression. Semin Cancer Biol. 2020;60:72-95. [PubMed] [DOI] |
| 24. | Wang X, Zhang H, Yang H, Bai M, Ning T, Deng T, Liu R, Fan Q, Zhu K, Li J, Zhan Y, Ying G, Ba Y. Exosome-delivered circRNA promotes glycolysis to induce chemoresistance through the miR-122-PKM2 axis in colorectal cancer. Mol Oncol. 2020;14:539-555. [PubMed] [DOI] |
| 25. | Li J, Qin X, Wu R, Wan L, Zhang L, Liu R. Circular RNA circFBXO11 modulates hepatocellular carcinoma progress and oxaliplatin resistance through miR-605/FOXO3/ABCB1 axis. J Cell Mol Med. 2020;24:5152-5161. [PubMed] [DOI] |
| 26. | Ceccaldi R, Rondinelli B, D'Andrea AD. Repair Pathway Choices and Consequences at the Double-Strand Break. Trends Cell Biol. 2016;26:52-64. [PubMed] [DOI] |
| 27. | Huang X, Li Z, Zhang Q, Wang W, Li B, Wang L, Xu Z, Zeng A, Zhang X, Zhang X, He Z, Li Q, Sun G, Wang S, Li Q, Wang L, Zhang L, Xu H, Xu Z. Circular RNA AKT3 upregulates PIK3R1 to enhance cisplatin resistance in gastric cancer via miR-198 suppression. Mol Cancer. 2019;18:71. [PubMed] [DOI] |
| 28. | Bakir B, Chiarella AM, Pitarresi JR, Rustgi AK. EMT, MET, Plasticity, and Tumor Metastasis. Trends Cell Biol. 2020;30:764-776. [PubMed] [DOI] |
| 29. | Liu YY, Zhang LY, Du WZ. Circular RNA circ-PVT1 contributes to paclitaxel resistance of gastric cancer cells through the regulation of ZEB1 expression by sponging miR-124-3p. Biosci Rep. 2019;39. [PubMed] [DOI] |
| 30. | Bayik D, Lathia JD. Cancer stem cell-immune cell crosstalk in tumour progression. Nat Rev Cancer. 2021;21:526-536. [PubMed] [DOI] |
| 31. | Jing N, Gao WQ, Fang YX. Regulation of Formation, Stemness and Therapeutic Resistance of Cancer Stem Cells. Front Cell Dev Biol. 2021;9:641498. [PubMed] [DOI] |
| 32. | Xia Y, Lv J, Jiang T, Li B, Li Y, He Z, Xuan Z, Sun G, Wang S, Li Z, Wang W, Wang L, Xu Z. CircFAM73A promotes the cancer stem cell-like properties of gastric cancer through the miR-490-3p/HMGA2 positive feedback loop and HNRNPK-mediated β-catenin stabilization. J Exp Clin Cancer Res. 2021;40:103. [PubMed] [DOI] |
| 33. | Chmurska A, Matczak K, Marczak A. Two Faces of Autophagy in the Struggle against Cancer. Int J Mol Sci. 2021;22. [PubMed] [DOI] |
| 34. | Ma L, Wang Z, Xie M, Quan Y, Zhu W, Yang F, Zhao C, Fan Y, Fang N, Jiang H, Wang Q, Wang S, Zhou J, Chen X, Shu Y. Silencing of circRACGAP1 sensitizes gastric cancer cells to apatinib via modulating autophagy by targeting miR-3657 and ATG7. Cell Death Dis. 2020;11:169. [PubMed] [DOI] |
| 35. | Sung H, Ferlay J, Siegel RL, Laversanne M, Soerjomataram I, Jemal A, Bray F. Global Cancer Statistics 2020: GLOBOCAN Estimates of Incidence and Mortality Worldwide for 36 Cancers in 185 Countries. CA Cancer J Clin. 2021;71:209-249. [PubMed] [DOI] |
| 36. | Chan AWH, Zhong J, Berhane S, Toyoda H, Cucchetti A, Shi K, Tada T, Chong CCN, Xiang BD, Li LQ, Lai PBS, Mazzaferro V, García-Fiñana M, Kudo M, Kumada T, Roayaie S, Johnson PJ. Development of pre and post-operative models to predict early recurrence of hepatocellular carcinoma after surgical resection. J Hepatol. 2018;69:1284-1293. [PubMed] [DOI] |
| 37. | Yang JD, Hainaut P, Gores GJ, Amadou A, Plymoth A, Roberts LR. A global view of hepatocellular carcinoma: trends, risk, prevention and management. Nat Rev Gastroenterol Hepatol. 2019;16:589-604. [PubMed] [DOI] |
| 38. | El-Serag HB. Hepatocellular carcinoma. N Engl J Med. 2011;365:1118-1127. [PubMed] [DOI] |
| 39. | Cheng AL, Kang YK, Chen Z, Tsao CJ, Qin S, Kim JS, Luo R, Feng J, Ye S, Yang TS, Xu J, Sun Y, Liang H, Liu J, Wang J, Tak WY, Pan H, Burock K, Zou J, Voliotis D, Guan Z. Efficacy and safety of sorafenib in patients in the Asia-Pacific region with advanced hepatocellular carcinoma: a phase III randomised, double-blind, placebo-controlled trial. Lancet Oncol. 2009;10:25-34. [PubMed] [DOI] |
| 40. | Chen J, Jin R, Zhao J, Liu J, Ying H, Yan H, Zhou S, Liang Y, Huang D, Liang X, Yu H, Lin H, Cai X. Potential molecular, cellular and microenvironmental mechanism of sorafenib resistance in hepatocellular carcinoma. Cancer Lett. 2015;367:1-11. [PubMed] [DOI] |
| 41. | Xu J, Ji L, Liang Y, Wan Z, Zheng W, Song X, Gorshkov K, Sun Q, Lin H, Zheng X, Chen J, Jin RA, Liang X, Cai X. CircRNA-SORE mediates sorafenib resistance in hepatocellular carcinoma by stabilizing YBX1. Signal Transduct Target Ther. 2020;5:298. [PubMed] [DOI] |
| 42. | Yang C, Dong Z, Hong H, Dai B, Song F, Geng L, Lu J, Yang J, Sui C, Xu M. circFN1 Mediates Sorafenib Resistance of Hepatocellular Carcinoma Cells by Sponging miR-1205 and Regulating E2F1 Expression. Mol Ther Nucleic Acids. 2020;22:421-433. [PubMed] [DOI] |
| 43. | Weng H, Zeng L, Cao L, Chen T, Li Y, Xu Y, Peng Y, Ye Y. circFOXM1 contributes to sorafenib resistance of hepatocellular carcinoma cells by regulating MECP2 via miR-1324. Mol Ther Nucleic Acids. 2021;23:811-820. [PubMed] [DOI] |
| 44. | Kalyan A, Nimeiri H, Kulik L. Systemic therapy of hepatocellular carcinoma: current and promising. Clin Liver Dis. 2015;19:421-432. [PubMed] [DOI] |
| 45. | Li Y, Zhang Y, Zhang S, Huang D, Li B, Liang G, Wu Y, Jiang Q, Li L, Lin C, Wei Z, Meng L. circRNA circARNT2 Suppressed the Sensitivity of Hepatocellular Carcinoma Cells to Cisplatin by Targeting the miR-155-5p/PDK1 Axis. Mol Ther Nucleic Acids. 2021;23:244-254. [PubMed] [DOI] |
| 46. | Fan W, Chen L, Wu X, Zhang T. Circ_0031242 Silencing Mitigates the Progression and Drug Resistance in DDP-Resistant Hepatoma Cells by the miR-924/POU3F2 Axis. Cancer Manag Res. 2021;13:743-755. [PubMed] [DOI] |
| 47. | Guan Y, Zhang Y, Hao L, Nie Z. CircRNA_102272 Promotes Cisplatin-Resistance in Hepatocellular Carcinoma by Decreasing MiR-326 Targeting of RUNX2. Cancer Manag Res. 2020;12:12527-12534. [PubMed] [DOI] |
| 48. | Luo Y, Fu Y, Huang R, Gao M, Liu F, Gui R, Nie X. CircRNA_101505 sensitizes hepatocellular carcinoma cells to cisplatin by sponging miR-103 and promotes oxidored-nitro domain-containing protein 1 expression. Cell Death Discov. 2019;5:121. [PubMed] [DOI] |
| 49. | Chen H, Liu S, Li M, Huang P, Li X. circ_0003418 Inhibits Tumorigenesis And Cisplatin Chemoresistance Through Wnt/β-Catenin Pathway In Hepatocellular Carcinoma. Onco Targets Ther. 2019;12:9539-9549. [PubMed] [DOI] |
| 50. | Huang W, Huang F, Feng C. CircFoxo3 Promotes Adriamycin Resistance Through Regulation of miR-199a-5p/ATP Binding Cassette Subfamily C Member 1 Axis in Hepatocellular Carcinoma. Onco Targets Ther. 2020;13:5113-5122. [PubMed] [DOI] |
| 51. | Li X, Zhang T. Circular RNA PTGR1 regulates 5-FU resistance and development of hepatocellular carcinoma cells by modulating miR-129-5p/ABCC1 axis. Cell Biol Int. 2021;. [PubMed] [DOI] |
| 52. | Li X, He J, Ren X, Zhao H, Zhao H. Circ_0003998 enhances doxorubicin resistance in hepatocellular carcinoma by regulating miR-218-5p/EIF5A2 pathway. Diagn Pathol. 2020;15:141. [PubMed] [DOI] |
| 53. | Ham IH, Oh HJ, Jin H, Bae CA, Jeon SM, Choi KS, Son SY, Han SU, Brekken RA, Lee D, Hur H. Targeting interleukin-6 as a strategy to overcome stroma-induced resistance to chemotherapy in gastric cancer. Mol Cancer. 2019;18:68. [PubMed] [DOI] |
| 54. | Zhai J, Shen J, Xie G, Wu J, He M, Gao L, Zhang Y, Yao X, Shen L. Cancer-associated fibroblasts-derived IL-8 mediates resistance to cisplatin in human gastric cancer. Cancer Lett. 2019;454:37-43. [PubMed] [DOI] |
| 55. | Zheng P, Chen L, Yuan X, Luo Q, Liu Y, Xie G, Ma Y, Shen L. Exosomal transfer of tumor-associated macrophage-derived miR-21 confers cisplatin resistance in gastric cancer cells. J Exp Clin Cancer Res. 2017;36:53. [PubMed] [DOI] |
| 56. | Deng P, Sun M, Zhao WY, Hou B, Li K, Zhang T, Gu F. Circular RNA circVAPA promotes chemotherapy drug resistance in gastric cancer progression by regulating miR-125b-5p/STAT3 axis. World J Gastroenterol. 2021;27:487-500. [PubMed] [DOI] |
| 57. | Liu Y, Xu J, Jiang M, Ni L, Ling Y. CircRNA DONSON contributes to cisplatin resistance in gastric cancer cells by regulating miR-802/BMI1 axis. Cancer Cell Int. 2020;20:261. [PubMed] [DOI] |
| 58. | Zhang Q, Miao Y, Fu Q, Hu H, Chen H, Zeng A, Jin Y, Jiang Y, Qian L, Wu L, Xu L, Wang G, Qiu L, Huang X, Xia Y. CircRNACCDC66 regulates cisplatin resistance in gastric cancer via the miR-618/BCL2 axis. Biochem Biophys Res Commun. 2020;526:713-720. [PubMed] [DOI] |
| 59. | Huang XX, Zhang Q, Hu H, Jin Y, Zeng AL, Xia YB, Xu L. A novel circular RNA circFN1 enhances cisplatin resistance in gastric cancer via sponging miR-182-5p. J Cell Biochem. 2020;. [PubMed] [DOI] |
| 60. | Xue M, Li G, Fang X, Wang L, Jin Y, Zhou Q. hsa_circ_0081143 promotes cisplatin resistance in gastric cancer by targeting miR-646/CDK6 pathway. Cancer Cell Int. 2019;19:25. [PubMed] [DOI] |
| 61. | Yao W, Guo P, Mu Q, Wang Y. Exosome-Derived Circ-PVT1 Contributes to Cisplatin Resistance by Regulating Autophagy, Invasion, and Apoptosis Via miR-30a-5p/YAP1 Axis in Gastric Cancer Cells. Cancer Biother Radiopharm. 2021;36:347-359. [PubMed] [DOI] |
| 62. | Wang X, Zhang Y, Li W, Liu X. Knockdown of cir_RNA PVT1 Elevates Gastric Cancer Cisplatin Sensitivity via Sponging miR-152-3p. J Surg Res. 2021;261:185-195. [PubMed] [DOI] |
| 63. | Zhang Z, Yu X, Zhou B, Zhang J, Chang J. Circular RNA circ_0026359 Enhances Cisplatin Resistance in Gastric Cancer via Targeting miR-1200/POLD4 Pathway. Biomed Res Int. 2020;2020:5103272. [PubMed] [DOI] |
| 64. | Zhong Y, Wang D, Ding Y, Tian G, Jiang B. Circular RNA circ_0032821 contributes to oxaliplatin (OXA) resistance of gastric cancer cells by regulating SOX9 via miR-515-5p. Biotechnol Lett. 2021;43:339-351. [PubMed] [DOI] |
| 65. | Gao H, Xu J, Qiao F, Xue L. Depletion of hsa_circ_0000144 Suppresses Oxaliplatin Resistance of Gastric Cancer Cells by Regulating miR-502-5p/ADAM9 Axis. Onco Targets Ther. 2021;14:2773-2787. [PubMed] [DOI] |
| 66. | Peng L, Sang H, Wei S, Li Y, Jin D, Zhu X, Li X, Dang Y, Zhang G. circCUL2 regulates gastric cancer malignant transformation and cisplatin resistance by modulating autophagy activation via miR-142-3p/ROCK2. Mol Cancer. 2020;19:156. [PubMed] [DOI] |
| 67. | Sun G, Li Z, He Z, Wang W, Wang S, Zhang X, Cao J, Xu P, Wang H, Huang X, Xia Y, Lv J, Xuan Z, Jiang T, Fang L, Yang J, Zhang D, Xu H, Xu Z. Circular RNA MCTP2 inhibits cisplatin resistance in gastric cancer by miR-99a-5p-mediated induction of MTMR3 expression. J Exp Clin Cancer Res. 2020;39:246. [PubMed] [DOI] |
| 68. | Wu Q, Wang H, Liu L, Zhu K, Yu W, Guo J. Hsa_circ_0001546 acts as a miRNA-421 sponge to inhibit the chemoresistance of gastric cancer cells via ATM/Chk2/p53-dependent pathway. Biochem Biophys Res Commun. 2020;521:303-309. [PubMed] [DOI] |
| 69. | Xu G, Li M, Wu J, Qin C, Tao Y, He H. Circular RNA circNRIP1 Sponges microRNA-138-5p to Maintain Hypoxia-Induced Resistance to 5-Fluorouracil Through HIF-1α-Dependent Glucose Metabolism in Gastric Carcinoma. Cancer Manag Res. 2020;12:2789-2802. [PubMed] [DOI] |
| 70. | Xu QY, Xie MJ, Huang J, Wang ZW. Effect of circ MTHFD2 on resistance to pemetrexed in gastric cancer through regulating expression of miR-124. Eur Rev Med Pharmacol Sci. 2019;23:10290-10299. [PubMed] [DOI] |
| 71. | Lu Y, Li L, Li L, Wu G, Liu G. Circular RNA circHECTD1 prevents Diosbulbin-B-sensitivity via miR-137/PBX3 axis in gastric cancer. Cancer Cell Int. 2021;21:264. [PubMed] [DOI] |
| 72. | Gustavsson B, Carlsson G, Machover D, Petrelli N, Roth A, Schmoll HJ, Tveit KM, Gibson F. A review of the evolution of systemic chemotherapy in the management of colorectal cancer. Clin Colorectal Cancer. 2015;14:1-10. [PubMed] [DOI] |
| 73. | Zheng GL, Liu YL, Yan ZX, Xie XY, Xiang Z, Yin L, Wang QQ, Chong DC, Xue GL, Xu LL, Zhou K, Wang Q. Elevated LOXL2 expression by LINC01347/miR-328-5p axis contributes to 5-FU chemotherapy resistance of colorectal cancer. Am J Cancer Res. 2021;11:1572-1585. [PubMed] |
| 74. | Sui Q, Peng J, Han K, Lin J, Zhang R, Ou Q, Qin J, Deng Y, Zhou W, Kong L, Tang J, Xiao B, Li Y, Yu L, Fang Y, Ding PR, Pan Z. Voltage-gated sodium channel Nav1.5 promotes tumor progression and enhances chemosensitivity to 5-fluorouracil in colorectal cancer. Cancer Lett. 2021;500:119-131. [PubMed] [DOI] |
| 75. | He X, Ma J, Zhang M, Cui J, Yang H. Circ_0007031 enhances tumor progression and promotes 5-fluorouracil resistance in colorectal cancer through regulating miR-133b/ABCC5 axis. Cancer Biomark. 2020;29:531-542. [PubMed] [DOI] |
| 76. | Chen H, Pei L, Xie P, Guo G. Circ-PRKDC Contributes to 5-Fluorouracil Resistance of Colorectal Cancer Cells by Regulating miR-375/FOXM1 Axis and Wnt/β-Catenin Pathway. Onco Targets Ther. 2020;13:5939-5953. [PubMed] [DOI] |
| 77. | Li S, Zheng S. Down-Regulation of Circ_0032833 Sensitizes Colorectal Cancer to 5-Fluorouracil and Oxaliplatin Partly Depending on the Regulation of miR-125-5p and MSI1. Cancer Manag Res. 2020;12:11257-11269. [PubMed] [DOI] |
| 78. | Ren TJ, Liu C, Hou JF, Shan FX. CircDDX17 reduces 5-fluorouracil resistance and hinders tumorigenesis in colorectal cancer by regulating miR-31-5p/KANK1 axis. Eur Rev Med Pharmacol Sci. 2020;24:1743-1754. [PubMed] [DOI] |
| 79. | Lin YC, Yu YS, Lin HH, Hsiao KY. Oxaliplatin-Induced DHX9 Phosphorylation Promotes Oncogenic Circular RNA CCDC66 Expression and Development of Chemoresistance. Cancers (Basel). 2020;12. [PubMed] [DOI] |
| 80. | Lai M, Liu G, Li R, Bai H, Zhao J, Xiao P, Mei J. Hsa_circ_0079662 induces the resistance mechanism of the chemotherapy drug oxaliplatin through the TNF-α pathway in human colon cancer. J Cell Mol Med. 2020;24:5021-5027. [PubMed] [DOI] |
| 81. | Zhang W, Wang Z, Cai G, Huang P. Downregulation of Circ_0071589 Suppresses Cisplatin Resistance in Colorectal Cancer by Regulating the MiR-526b-3p/KLF12 Axis. Cancer Manag Res. 2021;13:2717-2731. [PubMed] [DOI] |
| 82. | Xi L, Liu Q, Zhang W, Luo L, Song J, Liu R, Wei S, Wang Y. Circular RNA circCSPP1 knockdown attenuates doxorubicin resistance and suppresses tumor progression of colorectal cancer via miR-944/FZD7 axis. Cancer Cell Int. 2021;21:153. [PubMed] [DOI] |
| 83. | Jian X, He H, Zhu J, Zhang Q, Zheng Z, Liang X, Chen L, Yang M, Peng K, Zhang Z, Liu T, Ye Y, Jiao H, Wang S, Zhou W, Ding Y, Li T. Hsa_circ_001680 affects the proliferation and migration of CRC and mediates its chemoresistance by regulating BMI1 through miR-340. Mol Cancer. 2020;19:20. [PubMed] [DOI] |
| 84. | Arnold M, Soerjomataram I, Ferlay J, Forman D. Global incidence of oesophageal cancer by histological subtype in 2012. Gut. 2015;64:381-387. [PubMed] [DOI] |
| 85. | Abnet CC, Arnold M, Wei WQ. Epidemiology of Esophageal Squamous Cell Carcinoma. Gastroenterology. 2018;154:360-373. [PubMed] [DOI] |
| 86. | Feng W, Xiaoyan X, Xuan Y, Xiangke L, Zichang Y, Ran Z, Liuxing W, Qingxia F. Silencing stathmin-modulating efficiency of chemotherapy for esophageal squamous cell cancer with paclitaxel. Cancer Gene Ther. 2015;22:115-121. [PubMed] [DOI] |
| 87. | Qu F, Wang L, Wang C, Yu L, Zhao K, Zhong H. Circular RNA circ_0006168 enhances Taxol resistance in esophageal squamous cell carcinoma by regulating miR-194-5p/JMJD1C axis. Cancer Cell Int. 2021;21:273. [PubMed] [DOI] |
| 88. | Zhu H, Du F, Cao C. Restoration of circPSMC3 sensitizes gefitinib-resistant esophageal squamous cell carcinoma cells to gefitinib by regulating miR-10a-5p/PTEN axis. Cell Biol Int. 2021;45:107-116. [PubMed] [DOI] |
| 89. | Oneda E, Zaniboni A. Are We Sure that Adjuvant Chemotherapy is the Best Approach for Resectable Pancreatic Cancer? Are We in the Era of Neoadjuvant Treatment? A Review of Current Literature. J Clin Med. 2019;8. [PubMed] [DOI] |
| 90. | Lin KI, Yang JL, Lin YC, Chou CY, Chen JH, Hung CC. Network Meta-Analysis of Efficacy and Safety of Chemotherapy and Target Therapy in the First-Line Setting of Advanced Pancreatic Cancer. Cancers (Basel). 2019;11. [PubMed] [DOI] |
| 91. | Liu Y, Xia L, Dong L, Wang J, Xiao Q, Yu X, Zhu H. CircHIPK3 Promotes Gemcitabine (GEM) Resistance in Pancreatic Cancer Cells by Sponging miR-330-5p and Targets RASSF1. Cancer Manag Res. 2020;12:921-929. [PubMed] [DOI] |