修回日期: 2008-02-25
接受日期: 2008-04-11
在线出版日期: 2008-04-18
目的: 探讨肽核酸(peptide or polyamide nucleic acid, PNA)对大肠癌细胞端粒酶活性的抑制作用及对大肠癌细胞生长的影响.
方法: 应用脂质体LipfectamineTM介导不同浓度(25, 50, 100, 200, 400 nmol/L)PNA-DNA杂交混合物转染LS-174T细胞, 通过细胞克隆生长抑制试验来测定PNA对细胞生长的最佳抑制剂量; 选用最佳抑制剂量PNA转染LS-174T细胞, 同时设置脂质体组(LipfectamineTM)组和空白对照组, 观察克隆形成率、细胞形态及群体生长差异.
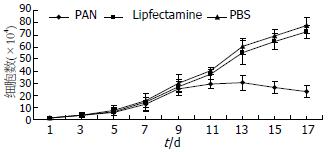
结果: PNA对LS-174T细胞最佳抑制浓度为200 nmol/L. PNA实验组端粒酶活性和克隆形成率明显低于脂质体组和空白对照组(0.003±0.001 vs 2.334±0.025, 2.528±0.032, P<0.01; 5.20% vs 45.45%, 47.13%, P<0.01). PNA组癌细胞生长出现典型形态学改变, 细胞变圆、胞膜皱缩、细胞核边集于核膜, 通过群体生长曲线, 转染后11 d出现抑制作用, 15、17 d抑制作用最显著.
结论: 体外试验中PNA对大肠癌LS-174T细胞生长有明显的抑制作用, 其作用机制可能是PNA对大肠癌细胞端粒酶活性的抑制.
引文著录: 赵旭海, 王贵玉, 王轶慧, 姜世雄, 王锡山. 肽核酸对大肠癌LS-174T细胞生长抑制作用. 世界华人消化杂志 2008; 16(11): 1179-1183
Revised: February 25, 2008
Accepted: April 11, 2008
Published online: April 18, 2008
AIM: To explore the inhibitory effect of peptide nucleic acid (PNA) on the telomerase activity and the growth of colorectal cancer cells.
METHODS: LS-174T cells were transfected with the mixture of PNA-DNA at different concentrations (25, 50, 100, 200, 400 nmol/L) mediated by LipfectamineTM. Cell cloning experiment was used to select the best inhibition concentration of PNA, and then LS-174T cells were transfected with this concentration. At the same time, liposome (using LipfectamineTM) group and empty control group were established. The clone formation rate, cancer cell morphology and group growth difference were compared between groups.
RESULTS: Cell cloning experiment identified that the best inhibition concentration was 200 nmol/L. The telomerase activity and clone formation rate were notably lower in PNA-transfected group than those in LipfectamineTM and empty control group (0.003 ± 0.001 vs 2.334 ± 0.025, 2.528 ± 0.032, P < 0.01; 5.20% vs 45.45%, 47.13%, P < 0.01). In the PNA-transfected group, cancer cells showed typical morphological changes, such as becoming round, membrane shrinkage, and nucli concentrating at karyotheca, etc. Group growth curve manifested that the inhibition emerges 11 d after transfection and became notable 15 d or 17 d later.
CONCLUSION: PNA can inhibit the growth of LS-174T cells obviously through suppressing telomerase activity.
- Citation: Zhao XH, Wang GY, Wang YH, Jiang SX, Wang XS. Inhibitory effect of peptide nucleic acid on the growth of colorectal cancer LS-174T cells. Shijie Huaren Xiaohua Zazhi 2008; 16(11): 1179-1183
- URL: https://www.wjgnet.com/1009-3079/full/v16/i11/1179.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v16.i11.1179
胃端粒酶是一种核糖核蛋白酶, 在体细胞的表达很少, 但85%以上的恶性肿瘤细胞存在有端粒酶活性, 以端粒酶为靶点的端粒酶抑制剂研究是近年抗肿瘤领域研究的新热点[1-4]. 在诸多端粒酶抑制剂中, Nielsen et al[5-8]人工合成的肽核酸(peptide nucleic acids, PNA), 因其生物稳定性高和对核酸的高度亲和力, 是恶性肿瘤治疗中最有前途的基因治疗试剂. 本研究首次采用脂质体LipfectamineTM作为载体[9-10], 成功地转染大肠癌LS-174T细胞, 抑制了细胞端粒酶活性并明显地抑制其生长繁殖. PNA为大肠癌及其他恶性肿瘤创立新的基因治疗方法奠定了实验基础[11-15].
大肠癌LS-174T细胞系, 购于中国科学院上海细胞研究所; 肽核酸(PNA): 结构为5'-TAGGGTTAGGACAA-3', 由派德公司合成; PNA寡核苷酸: 结构为5'-TTGTCT-3', 由上海生工生物工程技术服务有限公司合成; 脂质体(LipfectamineTM)、端粒酶PCR-ELISA试剂盒和MEM培养基等.
1.2.1 PNA对LS-174T细胞提取液端粒酶活性的抑制试验: 提取LS-174T细胞裂解液, 应用PCR-ELISA方法检测LS-174T细胞端粒酶活性. 并将裂解液随机分成5份, 加入PNA, 浓度分别为12.5、25、50、100、200 nmol/L. 然后进行PCR扩增, 按端粒酶PCR-ELISA试剂盒说明书操作, 制备的样本用酶标仪测450 nm和690 nm波长的A值. A差值ΔA = A450-A690代表细胞端粒酶活性.
1.2.2 PNA-DNA寡核苷酸嵌合体脂质体转染剂的制备: 将浓度为200 μmol/L的PNA和DNA寡核苷酸液各100 μL混合杂交后[2-3,16-17], 与脂质体 LipfectamineTM混合, 制成终浓度为1 μmol/L的PNA-DNA和脂质体转染剂[5], 即为转染细胞试验的工作液. 同时配制浓度为12 mg/L的LipfectamineTM工作液1.5 mL, 作为转染试验中脂质体的对照样品.
1.2.3 PNA转染LS-174T细胞及最佳剂量试验: 取对数生长的LS-174T细胞稀释至浓度4×105个/L. 接种24孔板, 每孔500 μL, 分6组, 每组4孔. 对照组每孔加500 μL培养基. 实验组加PNA-DNA杂交体混合液, 分5个浓度25、50、100、200、400 nmol/L, 连续培养15 d. 实验结束时, 用Giemsa染色, 在相差显微镜下, 计数50个细胞以上的克隆数. 根据细胞集落形成抑制情况选择PNA最佳作用剂量.
1.2.4 PNA对LS-174T细胞集落形成和细胞形态影响试验: 将LS-174T细胞消化计数成8×105个/L细胞浓度, 接种3块六孔板, 分3组. 空白对照组, PNA-DNA转染组, LipfectamineTM组, 每组6孔. 每孔加1 mL细胞悬液. 空白对照组各孔加PBS 400 μL; 转染组各孔加PNA-DNA转染剂工作液(浓度为1 μmol/L) 400 μL; 脂质体组各孔加浓度为12 mg/L的LipfectamineTM 400 μL. 各孔终体积用MEM补至2 mL培养15 d, 隔日换液加药. 并从第6天起观察各组细胞生长状况和细胞形态变化并拍摄照片. 培养停止后计数细胞克隆形成率.
1.2.5 PNA对LS-174T细胞群体生长抑制试验: 将LS-174T细胞消化稀释成2×107个/L, 接种3块24孔培养板, 每孔0.5 mL细胞数1×104个. 分3组, 分别加PBS, 1 μmol/L PNA-DNA工作液, 12 mg/L的LipfectamineTM各200 μL, 并用MEM补足终容积为1 mL/孔. 隔日每组取2孔细胞进行台酚兰染色, 计数活细胞数, 取2孔平均数, 其余孔换液加药, 最后绘制生长曲线. 同时对生长第11, 15天的PNA实验组细胞进行端粒酶活性测定.
统计学处理 实验结果经方差分析、t检验进行统计学分析, 数值以mean±SD表示.
2.1.1 PCR-ELISA检测结果: LS-174T细胞端粒酶PCR-ELISA检测端粒酶活性为阳性. PNA在12.5、25、50、100、200 nmol/L浓度下对LS-174T细胞端粒酶提取液端粒酶活性均有明显的抑制作用, 并且对PNA浓度(在200 nmol/L范围内)具有依赖性, 即PNA浓度越高其抑制越明显; PNA转染LS-174T细胞后11-15 d范围, LS-174T细胞端粒酶活性阴性, 200 nmol/L PNA-DNA实验组细胞端粒酶活性明显低于PBS对照组和LipfectamineTM组(P<0.01, 表1).
| 分组 | 实验性质 | 端粒酶活性A值 |
| 293肾细胞 | 阳性对照 | 2.749±0.042 |
| 三蒸水 | 阴性对照 | 0.001±0.001 |
| LS-174T细胞裂解液 | 空白对照 | 2.528±0.032 |
| LS-174T细胞裂解液 | 12.5 nmol/L | 0.405±0.003 |
| 加入不同浓度的PNA | 25 nmol/L | 0.115±0.015 |
| 50 nmol/L | 0.071±0.006 | |
| 100 nmol/L | 0.028±0.001 | |
| 200 nmol/L | 0.003±0.001 | |
| 第11天提取物 | 0.065±0.005b | |
| 200 nmol/L浓度PNA转染 | 第15天提取物 | 0.038±0.006b |
| LS-174T细胞 | 第11天提取物 | 2.466±0.036 |
| 脂质体LipfectamineTM对照 | 第15天提取物 | 2.334±0.025 |
将细胞集落分成如表2中A、B、C、D 4组进行实验, 可见PNA在低浓度下对LS-174T细胞集落形成有抑制作用且与剂量成正比. 在200 nmol/L浓度下, PNA对LS-174T细胞集落形成抑制率达到87.8%. 因此我们选择200 nmol/L作为PNA作用的最佳剂量.
| PNA-DNA(nmol/L) | 克隆数 | mean±SD | 抑制率(%) | |||
| A | B | C | D | |||
| 0 | 118 | 120 | 107 | 106 | 112.75±7.27 | |
| 25 | 78 | 75 | 82 | 86 | 80.25±4.78 | 28.82 |
| 50 | 72 | 77 | 70 | 68 | 71.75±3.86 | 36.36 |
| 100 | 36 | 30 | 40 | 38 | 36.00±4.32 | 68.07 |
| 200 | 17 | 13 | 15 | 10 | 13.75±2.99 | 87.80 |
| 400 | 10 | 6 | 8 | 11 | 8.75±2.22 | 92.24 |
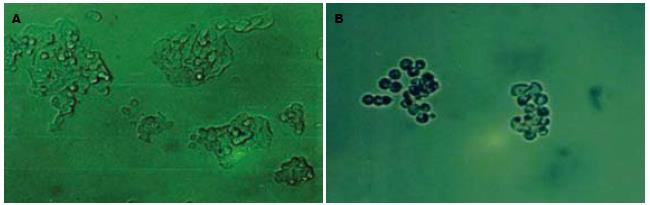
将细胞集落分成如表3中A、B、C、D、E、F 6组进行实验, 可见200 nmol/L PNA-DNA实验组的LS-174T细胞集落形成率明显低于PBS对照组和LipfectamineTM组(P<0.01). LipfectamineTM组集落形成率与PBS对照组比较无显著差异(P>0.05). PNA对LS-174T细胞具有很强的抑制作用, 抑制率达88.95%. 镜下观察实验组细胞有明显的形态学改变(图1), 在培养过程中, 细胞逐渐变圆, 细胞膜皱缩, 细胞核边集于胞膜, 有凋亡样的改变, 也有部分细胞变圆, 脱壁, 漂浮死亡, 主要表现集落细胞少, 不紧密.
| 克隆数 | mean±SD | 克隆形成率(%) | ||||||
| A | B | C | D | E | F | |||
| PNA(200 nmol/L) | 41 | 46 | 42 | 38 | 38 | 45 | 41.67±3.39 | 5.20 |
| LipfectamineTM(12 mg/L) | 383 | 355 | 343 | 367 | 345 | 389 | 363.67±19.38 | 45.45 |
| PBS(相同体积) | 392 | 372 | 363 | 381 | 365 | 389 | 377.00±12.25 | 47.13 |
大肠癌的发病率呈逐年上升的趋势[18], 尽管传统的外科手术治疗、化疗和放疗以及中药治疗在恶性肿瘤治疗中发挥了重要作用, 但迄今仍无法根治大肠癌[19-21]. 上述治疗进程均会对正常细胞及免疫系统造成损伤, 使预后较差. 分子生物学技术的飞跃发展, 特别是肿瘤生物治疗方法的研究和应用, 为人类抗癌治疗带来了新的曙光. 其中, 基因治疗又是国内外学者研究的热门课题.
自从"端粒-端粒酶-肿瘤"理论的提出[22-25], 国内外学者一直致力于探索以端粒酶为靶点治疗恶性肿瘤的新方法[26-27]. 本研究的结果显示, 无论在细胞水平(胞内转染)还是在分子水平, PNA对LS-174T细胞端粒酶活性均有显著的抑制作用, 且呈现剂量依赖模式; 同时PNA在细胞内的抑制作用有一定的潜伏期, 即在LS-174T细胞中约10-15 d左右, 癌细胞发生凋亡或死亡. PNA结构稳定, 不易被核酸酶和蛋白酶降解, 杂交特异性高而且杂交速度快, 因此在恶性肿瘤治疗方面具有光明的前景. 但是PNA分子质量较大, 且不带电荷[28-30], 因而不易进入细胞内, 影响抗癌作用的发挥. 应用脂质体介导在体内情况下是否可行? 有无毒副作用? 如何选择给药途径等一系列问题尚待进一步研究解决.
本实验结果证实, PNA在体外能有效地抑制大肠癌LS-174T细胞的生长增殖, 其作用的靶点是癌细胞的端粒酶; 作用的机制是通过抑制端粒酶活性, 使癌细胞端粒缩短而导致癌细胞的凋亡和消失, 达到抗肿瘤的目的; 脂质体LipfectamineTM介导PNA转染大肠癌LS-174T细胞是解决PNA不易进入肿瘤细胞的有效方法, 体外试验的成功为体内试验和应用奠定了一定的理论和实践基础.
大肠癌的发病率有逐年上升的趋势, 目前传统的治疗方法无法根治大肠癌. "端粒-端粒酶-肿瘤"理论提出后, 国内外学者一直在探索以端粒酶为靶点治疗恶性肿瘤的新方法, 端粒酶抑制剂是近年抗肿瘤领域研究的新热点, 是恶性肿瘤治疗中最有前途的基因治疗试剂. PNA因分子量较大且不带电荷, 不易进入细胞内, 影响了抗癌作用的发挥. 本研究首次采用脂质体作为载体, 成功地转染大肠癌LS-174T细胞, 抑制了细胞端粒酶活性并明显地抑制其生长繁殖, 为大肠癌及其他恶性肿瘤的基因治疗奠定了实验基础.
许玲, 副教授, 中国人民解放军第二军医大学长征医院中医科
端粒酶普遍存在于恶性肿瘤中, 是肿瘤细胞获得永生性的关键和肿瘤组织无限增殖的必要条件. 目前, 以端粒酶为靶点、以端粒酶抑制剂作为肿瘤治疗药物的研究已经得到广泛开展, 并显示出非常乐观的应用前景.
肽核酸分子质量较大, 且不带电荷, 因此不易进入细胞内, 这为其成为端粒酶抑制剂带来障碍, 但应用寡核苷酸杂交, 脂质体转染成功地使其成为理想的端粒酶抑制剂.
肽核酸: 是一种修饰的寡核苷酸, DNA类似物, 包含一个假晶状体骨架由N-(2-氨乙基)甘氨酸单位组成.
本文是体外实验研究, 文章条理清晰, 实验运用的技术成熟, 研究结果可信, 有可读性.
编辑:程剑侠 电编:郭海丽
| 1. | Vidaurreta M, Maestro ML, Rafael S, Veganzones S, Sanz-Casla MT, Cerdan J, Arroyo M. Telomerase activity in colorectal cancer, prognostic factor and implications in the microsatellite instability pathway. World J Gastroenterol. 2007;13:3868-3872. [PubMed] |
| 2. | Eitsuka T, Nakagawa K, Miyazawa T. Down-regulation of telomerase activity in DLD-1 human colorectal adenocarcinoma cells by tocotrienol. Biochem Biophys Res Commun. 2006;348:170-175. [PubMed] |
| 3. | Ohnishi T, Watanabe T, Nozawa H, Nagawa H. Telomerase subunit immunoreactivity and recurrence in colorectal cancer. Hepatogastroenterology. 2006;53:188-191. [PubMed] |
| 4. | Wege H, Brummendorf TH. Telomerase activation in liver regeneration and hepatocarcinogenesis: Dr. Jekyll or Mr. Hyde? Curr Stem Cell Res Ther. 2007;2:31-38. [PubMed] |
| 5. | Nielsen PE, Egholm M, Berg RH, Buchardt O. Sequence-selective recognition of DNA by strand displacement with a thymine-substituted polyamide. Science. 1991;254:1497-1500. [PubMed] |
| 6. | Yamamoto Y, Uehara A, Miura K, Watanabe A, Aburatani H, Komiyama M. Development of artificial restriction DNA cutter composed of Ce(Iv)/EDTA and PNA. Nucleosides Nucleotides Nucleic Acids. 2007;26:1265-1268. [PubMed] |
| 7. | Folini M, Bandiera R, Millo E, Gandellini P, Sozzi G, Gasparini P, Longoni N, Binda M, Daidone MG, Berg K. Photochemically enhanced delivery of a cell-penetrating peptide nucleic acid conjugate targeting human telomerase reverse transcriptase: effects on telomere status and proliferative potential of human prostate cancer cells. Cell Prolif. 2007;40:905-920. [PubMed] |
| 8. | Hu J, Corey DR. Inhibiting gene expression with peptide nucleic acid (PNA)--peptide conjugates that target chromosomal DNA. Biochemistry. 2007;46:7581-7589. [PubMed] |
| 9. | Wang XF, Chen YJ, Wang Q, Yang KF, Dai Q, Wang FM, Zhang XG. Establishment of cell line transfected with human BTLA gene and preliminary study on its biological function. Xibaoyufenzi Mianyixue Zazhi. 2007;23:201-204. [PubMed] |
| 10. | Li B, Zhang X, Gao M, Chu X. Effects of CD59 on antitumoral activities of phycocyanin from Spirulina platensis. Biomed Pharmacother. 2005;59:551-560. [PubMed] |
| 11. | Robak T. Therapy of chronic lymphocytic leukaemia with purine nucleoside analogues: facts and controversies. Drugs Aging. 2005;22:983-1012. [PubMed] |
| 12. | Pession A, Tonelli R. The MYCN oncogene as a specific and selective drug target for peripheral and central nervous system tumors. Curr Cancer Drug Targets. 2005;5:273-283. [PubMed] |
| 13. | Su X, Teh HF, Lieu X, Gao Z. Enzyme-based colorimetric detection of nucleic acids using peptide nucleic acid-immobilized microwell plates. Anal Chem. 2007;79:7192-7197. [PubMed] |
| 14. | Schatz P, Distler J, Berlin K, Schuster M. Novel method for high throughput DNA methylation marker evaluation using PNA-probe library hybridization and MALDI-TOF detection. Nucleic Acids Res. 2006;34:e59. [PubMed] |
| 15. | Tian X, Aruva MR, Wolfe HR, Qin W, Sauter ER, Thakur ML, Waldman SA, Wickstrom E. Tumor-targeting peptide-PNA-peptide chimeras for imaging overexpressed oncogene mRNAs. Nucleosides Nucleotides Nucleic Acids. 2005;24:1085-1091. [PubMed] |
| 16. | Roy S, Tanious FA, Wilson WD, Ly DH, Armitage BA. High-affinity homologous peptide nucleic acid probes for targeting a quadruplex-forming sequence from a MYC promoter element. Biochemistry. 2007;46:10433-10443. [PubMed] |
| 17. | Bender M, Holben WE, Sorensen SJ, Jacobsen CS. Use of a PNA probe to block DNA-mediated PCR product formation in prokaryotic RT-PCR. Biotechniques. 2007;42:609-610, 612-614. [PubMed] |
| 18. | Li M, Gu J. Changing patterns of colorectal cancer in China over a period of 20 years. World J Gastroenterol. 2005;11:4685-4688. [PubMed] |
| 19. | Kawamura Y, Konishi F. Surgery and chemotherapy for colorectal cancer: current status and perspective. Nippon Shokakibyo Gakkai Zasshi. 2007;104:1018-1024. [PubMed] |
| 20. | Pfannschmidt J, Dienemann H, Hoffmann H. Surgical resection of pulmonary metastases from colorectal cancer: a systematic review of published series. Ann Thorac Surg. 2007;84:324-338. [PubMed] |
| 21. | Platell CF. Changing patterns of recurrence after treatment for colorectal cancer. Int J Colorectal Dis. 2007;22:1223-1231. [PubMed] |
| 22. | Zhang Y, Cao EH, Liang XQ, Qin JF. Increasing sensitivity to arsenic trioxide-induced apoptosis by altered telomere state. Eur J Pharmacol. 2003;474:141-147. [PubMed] |
| 23. | Zhang YL, Zhang ZS, Wu BP, Zhou DY. Early diagnosis for colorectal cancer in China. World J Gastroenterol. 2002;8:21-25. [PubMed] |
| 24. | Sampedro Camarena F, Cano Serral G, Sampedro Santalo F. Telomerase and telomere dynamics in ageing and cancer: current status and future directions. Clin Transl Oncol. 2007;9:145-154. [PubMed] |
| 25. | Calcagnile O, Gisselsson D. Telomere dysfunction and telomerase activation in cancer--a pathological paradox? Cytogenet Genome Res. 2007;118:270-276. [PubMed] |
| 26. | Wang XS, Wang K, Li X, Fu SB. Effects of phosphorothioate anti-sense oligodeoxynucleotides on colorectal cancer cell growth and telomerase activity. World J Gastroenterol. 2004;10:3455-3458. [PubMed] |
| 27. | Vonderheide RH. Universal tumor antigens for cancer vaccination: targeting telomerase for immunoprevention. Discov Med. 2007;7:103-108. [PubMed] |
| 28. | Doyle DF, Braasch DA, Simmons CG, Janowski BA, Corey DR. Inhibition of gene expression inside cells by peptide nucleic acids: effect of mRNA target sequence, mismatched bases, and PNA length. Biochemistry. 2001;40:53-64. [PubMed] |
| 29. | Bukanov NO, Demidov VV, Nielsen PE, Frank-Kamenetskii MD. PD-loop: a complex of duplex DNA with an oligonucleotide. Proc Natl Acad Sci U S A. 1998;95:5516-5520. [PubMed] |
| 30. | Tackett AJ, Morris PD, Dennis R, Goodwin TE, Raney KD. Unwinding of unnatural substrates by a DNA helicase. Biochemistry. 2001;40:543-548. [PubMed] |