修回日期: 2007-02-01
接受日期: 2007-02-08
在线出版日期: 2007-04-18
目的: 探讨Runx3基因甲基化与胃癌的关系.
方法: 采用MSP法检测38例配对胃癌组织、癌旁正常组织和转移淋巴结中Runx3基因甲基化的情况.
结果: 73.7%的胃癌组织中存在Runx3基因异常甲基化, 而相应的癌旁正常组织和转移淋巴结中该基因的甲基化率分别为21.1%和65.8%. 癌组织和转移淋巴结中Runx3基因甲基化的发生率显著高于癌旁正常组织(P<0.05). 胃癌组织中, 该基因甲基化与肿瘤大小显著相关(P = 0.021), 但与肿瘤大体类型、分化程度、浸润深度及生长方式等临床病理特征无关.
结论: Runx3基因异常甲基化是胃癌发生、发展过程中的频繁事件, 通过检测胃黏膜组织及淋巴结中该基因的甲基化情况, 可能会对胃癌的早期诊断及判断淋巴结的微转移提供一定的参考价值.
引文著录: 杨少辉, 戴冬秋. 胃癌Runx3基因的甲基化. 世界华人消化杂志 2007; 15(11): 1283-1287
Revised: February 1, 2007
Accepted: February 8, 2007
Published online: April 18, 2007
AIM: To investigate the relationship between the methylation status of Runx3 gene and human primary gastric cancer.
METHODS: The methylation status of Runx3 gene in 38 gastric carcinoma specimens, the corresponding normal gastric mucosa and lymph nodes was detected by methylation-specific polymerase chain reaction (MS-PCR).
RESULTS: Aberrant methylation of Runx3 gene was detected in 73.7% of gastric cancer specimens, 65.8% of lymph nodes and 21.1% of the corresponding normal mucosa, and there were significant differences between the former two and the latter. Runx3 methylation was significantly correlated with the tumor size (P = 0.021), but not with the general types, differentiation degree, invasion depth and growth style of gastric cancer.
CONCLUSION: Aberrant methylation of Runx3 gene is a common event in the occurrence and progression of gastric cancer, which may provide useful information for the early diagnosis and assessment of lymph node metastasis.
- Citation: Yang SH, Dai DQ. Methylation status of Runx3 gene in human gastric cancer. Shijie Huaren Xiaohua Zazhi 2007; 15(11): 1283-1287
- URL: https://www.wjgnet.com/1009-3079/full/v15/i11/1283.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v15.i11.1283
胃癌是世界上最常见的恶性肿瘤之一, 其发生、发展与多种肿瘤抑制基因和肿瘤相关基因的失活有关. 目前的研究发现, 胃癌中许多肿瘤抑制基因和肿瘤相关基因的失活是由于基因启动子区的异常甲基化引起的, 通过去甲基化试剂干预, 一些胃癌细胞株可以恢复这些基因的功能[1-3]. Runx3蛋白是TGF-β超家族信号传导通路下游的一个转录因子, 介导TGF-β生长抑制的功能, 在哺乳动物的发育及组织分化过程中发挥重要作用, 对防止肿瘤的发生及遏制肿瘤的发展具有重要意义[4]. 胃癌中, Runx3基因功能缺失严重, 但突变率很低, 提示存在其他的失活方式. 鉴于Runx3基因启动子区具有典型的CpG岛序列, 我们采用甲基化特异性聚合酶链反应(methylation-specific PCR, MSP)法检测了胃癌原发灶、癌旁正常组织及转移淋巴结中Runx3基因的甲基化状态, 以期揭示Runx3基因甲基化与胃癌发生、发展以及与临床病理特征的关系.
收集中国医科大学附属第一医院肿瘤外科2005-02/08手术切除的胃癌组织, 相应的癌旁正常组织和转移淋巴结. 所取组织离体后立即一分为二, 一份液氮中速冻10 min, 然后置-70 ℃冰箱中保存. 另一份以40 g/L甲醛溶液固定, 制作常规病理切片, HE染色, 确定病理诊断. 所有胃癌患者术前均未行化疗或放疗, 男26例, 女性2例, 年龄为36-80(中位57)岁. 所有癌旁组织经病理检查未发现肿瘤细胞, 所取淋巴结组织经病理证实存在癌细胞. 正常对照标本取自无消化系统疾病及任意肿瘤的正常人外周血, EDTA抗凝. hydroquinone购自Fluka公司, sodium bisulfite购自Sigma公司, Wizard DNA Clean-Up System购自Promega公司, Taq酶购自大连宝生物公司. 甲基化酶SssⅠ为New England公司产品, 引物委托大连宝生物公司合成.
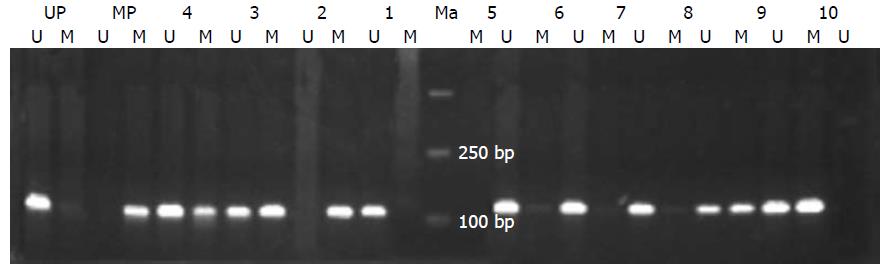
常规酚/氯仿抽提法提取DNA, -20 ℃暂存. 取4 μg DNA, 去离子水稀释至90 μL, 按文献[5]中的方法进行DNA预处理, 之后用Wizard DNA Clean-up Systerm纯化, 方法按说明书操作. 用50 mL TE溶解DNA, -20 ℃贮存备用. 冰上配制PCR反应体系(20 μL): 去离子水13.2 μL, 10×Buffer 2 μL, dNTP (2.5 mmol/L) 1.6 μL, sodium bisulfite修饰后的DNA模本2 μL, 引物(0.01 mol/L)各0.5 μL, Taq酶(5×106 U/L) 0.2 μL. 95 ℃预变性5 min之后进行40个循环的反应: 95 ℃变性20 s, 55 ℃复性20 s, 72 ℃延伸30 s. 最后一个循环后延伸7 min. RUNX3甲基化特异性引物序列: 正向: 5'-ATA ATA GCG GTC GTT AGG GCG TCG-3'; 反向: 5'-GCT TCT ACT TTC CCA CTT CTC ACA-3'; 产物大小115 bp. Runx3非甲基化特异性引物: 正向: 5'-ATA ATA GTG GTT GTT AGG GTG TTG-3'; 反向: 5'-ACT TCT ACT TTC CCA CTT CTC ACA-3'; 产物大小115 bp. GenBank Accession No. AL023096. 取5 μL PCR产物在25 g/L琼脂糖凝胶上电泳, GeneFinder染料染色. 每批标本均同时扩增甲基化酶SssⅠ修饰和未修饰的正常人外周血DNA作为甲基化阳性和阴性对照.
统计学处理 采用SPSS13.0软件进行Fisher's精确检验, P<0.05为差异有统计学意义.
用Runx3甲基化特异性引物和Runx3非甲基化特异性引物扩增甲基化酶SssⅠ修饰(甲基化阳性对照)和未修饰(甲基化阴性对照)的正常人外周血DNA时, 分别只有M引物和U引物得到预期大小的扩增片段(图1), 提示实验技术及所用引物和试剂正确, 实验结果可信. Runx3基因在胃癌组织、癌旁正常组织和转移淋巴结中均存在甲基化修饰, 其阳性率分别是73.7%, 21.1%, 65.8%, 部分甲基化特异性PCR检测结果见图1. 结果表明胃癌组织与转移淋巴结中甲基化率无差异(P>0.05), 两者与癌旁正常组织比较分别存在差异(P<0.01). 原发灶中, Runx3基因甲基化与癌灶大小相关(P = 0.021), 但与肿瘤大体类型、分化程度、浸润深度及生长方式等临床病理特征无关(表1).
| 临床病理特征 | n | 癌旁组织甲基化 | 原发灶甲基化 | 转移淋巴结甲基化 |
| 肿瘤大小 | ||||
| ≤5 cm | 14 | 1(7.1) | 7(50.0) | 7(50.0) |
| >5 cm | 24 | 7(29.2) | 21(87.5)a | 18(75.0) |
| Borrmann分型 | ||||
| Borrmann Ⅰ+Ⅱ | 1 | 0(0) | 1(100.0) | 1(100.0) |
| Borrmann Ⅲ | 31 | 7(22.6) | 23(74.2) | 20(64.5) |
| Borrmann Ⅳ | 5 | 1(20.0) | 4(80.0) | 4(80.0) |
| 生长方式 | ||||
| 巢状+团状 | 15 | 3(20.0) | 10(66.7) | 10(66.7) |
| 弥漫性 | 23 | 5(21.7) | 18(78.3) | 15(65.2) |
| 分化程度 | ||||
| 高/中分化 | 13 | 3(23.1) | 10(76.9) | 8(61.5) |
| 低分化 | 25 | 5(20.0) | 18(72.0) | 17(68.0) |
| 浸润深度 | ||||
| T1+2 | 26 | 4(15.4) | 19(73.1) | 16(61.5) |
| T3+4 | 12 | 4(33.3) | 9(75.0) | 9(75.0) |
DNA甲基化是具有可逆性与遗传性的一种基因修饰方式, 是指DNA双螺旋中, 胞嘧啶核苷的嘧啶环第5位碳原子在DNA甲基转移酶(DNA methyltrasferases, DNMTs)介导下以S-腺苷蛋氨酸作为甲基供体而发生甲基化[6]. 目前普遍认为, DNA异常甲基化改变是除缺失与突变之外导致肿瘤抑制基因失活的第3种机制. Kang et al[7]研究了一系列胃癌癌前病变组织和癌组织中12个基因的甲基化状态, 发现平均每例胃癌组织中有5.2个基因存在甲基化. 可见肿瘤抑制基因发生甲基化是胃癌形成过程中的频繁事件, 其在肿瘤的形成和发展中的作用及其作为分子治疗的靶向的潜在价值不容忽视. Runx3蛋白是TGF-β超家族信号传导通路下游的一个转录因子, Li et al[8]发现, 缺乏Runx3基因的小鼠胃黏膜上皮细胞增殖加快, 而其凋亡受到抑制, 使黏膜明显增厚, 并且细胞对生长抑制因子(例如TGF-β)表现出拮抗效应, 表明Runx3基因是胃上皮细胞生长和增殖的调控者. 大约一半以上的胃癌组织和更高比例的胃癌细胞株中, Runx3基因表达明显降低甚至缺失[9-12], 将外源性Runx3基因转入Runx3缺失的裸鼠, 结果显示转基因组的胃癌细胞集落大小和数目均明显少于对照组[10]. 与之相比, 目前虽然已经发现多种与胃癌相关的肿瘤抑制基因, 但他们与胃癌的相关性偏小, 因而, Runx3很可能是与胃癌发生相关的一个关键性基因[7]. Guo et al[13]以化疗药物处理Runx3基因高表达的胃癌细胞系, 发现其对化疗药物的敏感性显著提高, 说明Runx3基因对胃癌的治疗也具有重要的意义. 目前的研究发现, Runx3基因在肿瘤中很少发生突变[8,14-15], 却存在很高的甲基化率[16-17]. 我们采用MSP法检测了38例胃癌组织中Runx3基因的甲基化情况, 发现73.7%(28/38)的胃癌组织中, Runx3基因启动子存在高甲基化, 这与先前报道的甲基化率基本相似[8-11,15]. 证实Runx3基因启动子甲基化是胃癌中的频繁事件. 我们研究发现, 胃癌组织中该基因甲基化与肿瘤大小显著相关, 肿瘤长>5 cm者发生基因甲基化的几率大于长径≤5 cm者. Waki et al[18]认为, 就胃黏膜组织而言, Runx3基因甲基化具有非常高的癌组织特异性. 在他们的实验中, 只有年龄≥77岁的非胃癌个体的胃黏膜才存在该基因甲基化, 即使在胃癌患者的癌旁正常组织, Runx3基因甲基化率也仅为8%(7/93). 但在我们的研究中, 癌旁正常组织Runx3基因甲基化率却高达21.1%(8/38). 对于两项研究结果间的差异, 我们认为主要是标本构成不同引起的. 在Waki et al[18]研究发现, 35.5%(33/93)的患者为早期胃癌, 而我们的患者中只有2.6%(1/38)是早期胃癌, 绝大多数是进展期胃癌, 且已经出现淋巴结转移. 我们推测, 一方面我们所检测的癌旁组织的性质更接近癌组织, 其发生甲基化的可能性要高. Kim et al[10]研究发现, Runx3基因在慢性胃炎、肠化生和胃腺瘤、胃癌组织中的甲基化率呈递增趋势(P<0.05). 遗憾的是我们无法得知Waki et al[18]所检测的癌旁组织的病理性质, 因而不能作出较为明确的比较. 另一方面, 虽然我们所检测的癌旁组织经病理证实无癌细胞, 但可能已经有形态学无法观察到的癌细胞发生或浸润. 我们对癌旁正常组织的检测分析同时也说明Runx3基因甲基化是胃癌发生的早期事件, 除了检测癌组织和癌旁正常组织Runx3基因甲基化情况外, 我们还首次检测了转移淋巴结中该基因的甲基化情况. 由于转移淋巴结中存在与原发灶同质的癌细胞, 因而转移淋巴结中Runx3 基因的甲基化趋势与原发灶基本一致是容易理解的. 另外, 不同于原发灶, 92.1%(35/38)转移淋巴结标本都得到了Runx3非甲基化特异性引物的扩增片段, 这可能是由于Runx3基因不仅在胃黏膜细胞中表达, 他在T淋巴细胞的发育过程中也具有重要作用[19]. 既然我们在存在癌细胞转移的淋巴结中可以检测到Runx3基因的甲基化, 且阳性率是如此之高, 那我们是否可以通过检测淋巴结中Runx3基因的甲基化状态来判断癌细胞的微转移呢? 这有待于我们进一步研究.
我们的研究发现, Runx3基因甲基化与肿瘤大体类型、生长方式、浸润深度等临床病理特征无明显的相关性, 这可能与我们所采用的样本量小有关, 有待于扩大样本量以进一步研究.
DNA甲基化是哺乳动物细胞基因表达调控的重要方式之一, 研究发现, 恶性肿瘤组织中普遍存在着肿瘤抑制基因启动子高甲基化的现象, 通过去甲基化试剂干预, 一些胃癌细胞株可以恢复这些基因的功能.
淋巴结转移是胃癌转移的主要途径, 以往对胃癌甲基化的研究主要集中于胃癌组织和癌旁正常组织, 本研究首次采用MSP法同时检测了胃癌组织、癌旁正常组织和转移淋巴结中Runx3基因的甲基化现象.
本研究发现, Runx3基因甲基化是胃癌发生、发展过程中的频繁事件, 通过检测胃黏膜组织中该基因的甲基化情况, 有助于胃癌的早期诊断, 而检测胃癌相应淋巴结中该基因的甲基化情况有可能成为发现胃癌微转移的新方法.
甲基化特异性PCR: 用亚硫酸氢钠处理DNA后, 未甲基化的胞嘧啶转变为尿嘧啶, 而甲基化的胞嘧啶保持不变. 据此, 设计甲基化特异性引物(假设模本链与引物结合的片段中胞嘧啶发生甲基化)和非甲基化特异性引物(假设模本链与引物结合的片段中胞嘧啶未发生甲基化)对模本进行PCR扩增.
本文用甲基化特异性PCR的方法检测了胃癌, 转移淋巴结及癌旁组织中Runx3基因的甲基化情况, 文章书写规范, 方法可靠, 结果可信, 有一定的学术价值.
编辑:王晓瑜 电编:张敏
| 1. | Hong SH, Kim HG, Chung WB, Kim EY, Lee JY, Yoon SM, Kwon JG, Sohn YK, Kwak EK, Kim JW. DNA hypermethylation of tumor-related genes in gastric carcinoma. J Korean Med Sci. 2005;20:236-241. [PubMed] [DOI] |
| 2. | Homma N, Tamura G, Honda T, Jin Z, Ohmura K, Kawata S, Motoyama T. Hypermethylation of Chfr and hMLH1 in gastric noninvasive and early invasive neoplasias. Virchows Arch. 2005;446:120-126. [PubMed] [DOI] |
| 3. | Kim TY, Jong HS, Jung Y, Kim TY, Kang GH, Bang YJ. DNA hypermethylation in gastric cancer. Aliment Pharmacol Ther. 2004;20 Suppl 1:131-142. [PubMed] [DOI] |
| 4. | Bangsow C, Rubins N, Glusman G, Bernstein Y, Negreanu V, Goldenberg D, Lotem J, Ben-Asher E, Lancet D, Levanon D. The RUNX3 gene--sequence, structure and regulated expression. Gene. 2001;279:221-232. [PubMed] [DOI] |
| 5. | Herman JG, Graff JR, Myöhänen S, Nelkin BD, Baylin SB. Methylation-specific PCR: a novel PCR assay for methylation status of CpG islands. Proc Natl Acad Sci U S A. 1996;93:9821-9826. [PubMed] [DOI] |
| 7. | Kang GH, Lee S, Kim JS, Jung HY. Profile of aberrant CpG island methylation along the multistep pathway of gastric carcinogenesis. Lab Invest. 2003;83:635-641. [PubMed] [DOI] |
| 8. | Li QL, Ito K, Sakakura C, Fukamachi H, Inoue Ki, Chi XZ, Lee KY, Nomura S, Lee CW, Han SB, Kim HM, Kim WJ, Yamamoto H, Yamashita N, Yano T, Ikeda T, Itohara S, Inazawa J, Abe T, Hagiwara A, Yamagishi H, Ooe A, Kaneda A, Sugimura T, Ushijima T, Bae SC, Ito Y. Causal relationship between the loss of RUNX3 expression and gastric cancer. Cell. 2002;109:113-124. [PubMed] [DOI] |
| 9. | Oshimo Y, Oue N, Mitani Y, Nakayama H, Kitadai Y, Yoshida K, Ito Y, Chayama K, Yasui W. Frequent loss of RUNX3 expression by promoter hypermethylation in gastric carcinoma. Pathobiology. 2004;71:137-143. [PubMed] [DOI] |
| 10. | Kim TY, Lee HJ, Hwang KS, Lee M, Kim JW, Bang YJ, Kang GH. Methylation of RUNX3 in various types of human cancers and premalignant stages of gastric carcinoma. Lab Invest. 2004;84:479-484. [PubMed] [DOI] |
| 11. | Guo WH, Weng LQ, Ito K, Chen LF, Nakanishi H, Tatematsu M, Ito Y. Inhibition of growth of mouse gastric cancer cells by Runx3, a novel tumor suppressor. Oncogene. 2002;21:8351-8355. [PubMed] [DOI] |
| 12. | Sakakura C, Hagiwara A, Miyagawa K, Nakashima S, Yoshikawa T, Kin S, Nakase Y, Ito K, Yamagishi H, Yazumi S. Frequent downregulation of the runt domain transcription factors RUNX1, RUNX3 and their cofactor CBFB in gastric cancer. Int J Cancer. 2005;113:221-228. [PubMed] [DOI] |
| 13. | Guo C, Ding J, Yao L, Sun L, Lin T, Song Y, Sun L, Fan D. Tumor suppressor gene Runx3 sensitizes gastric cancer cells to chemotherapeutic drugs by downregulating Bcl-2, MDR-1 and MRP-1. Int J Cancer. 2005;116:155-160. [PubMed] [DOI] |
| 14. | Mori T, Nomoto S, Koshikawa K, Fujii T, Sakai M, Nishikawa Y, Inoue S, Takeda S, Kaneko T, Nakao A. Decreased expression and frequent allelic inactivation of the RUNX3 gene at 1p36 in human hepatocellular carcinoma. Liver Int. 2005;25:380-388. [PubMed] [DOI] |
| 16. | Suzuki M, Shigematsu H, Shames DS, Sunaga N, Takahashi T, Shivapurkar N, Iizasa T, Frenkel EP, Minna JD, Fujisawa T. DNA methylation-associated inactivation of TGF-beta-related genes DRM/Gremlin, RUNX3, and HPP1 in human cancers. Br J Cancer. 2005;93:1029-1037. [PubMed] [DOI] |
| 17. | Kim WJ, Kim EJ, Jeong P, Quan C, Kim J, Li QL, Yang JO, Ito Y, Bae SC. RUNX3 inactivation by point mutations and aberrant DNA methylation in bladder tumors. Cancer Res. 2005;65:9347-9354. [PubMed] [DOI] |
| 18. | Waki T, Tamura G, Sato M, Terashima M, Nishizuka S, Motoyama T. Promoter methylation status of DAP-kinase and RUNX3 genes in neoplastic and non-neoplastic gastric epithelia. Cancer Sci. 2003;94:360-364. [PubMed] [DOI] |